【トピックス】
芳香族化合物を発酵生産するプラスミドフリーな大腸菌株の作製
駒 大輔
阪技術研
1.はじめに
2019年策定されたバイオ戦略では、2030年に世界最先端のバイオエコノミー社会を実現することが目標とされている。バイオエコノミー社会とは、環境・食料・健康等の諸課題の解決を図りながら、化石資源に依存しない持続的な経済成長を促進する社会を意味する。バイオエコノミー市場のひとつであるバイオものづくり・バイオ由来製品で、2030年を目標に50兆円規模の市場を創出するという野心的なロードマップが描かれている (内閣府バイオエコノミー戦略、https://www8.cao.go.jp/cstp/bio/index.html)。筆者が研究を開始した2006年には、バイオエコノミーという言葉は耳にしなかったが、現在はまさにバイオエコノミー社会を構築する一助になればという思いで、バイオものづくりに関連する研究開発を行っている。
バイオものづくりには、多様なバイオテクノロジー技術が関与するが、中核技術の一つに微生物を用いた発酵生産技術が挙げられる。近年は、再生可能なバイオマス資源を利用して、多様な化合物を自然界ではありえないような濃度で生産する微生物が開発されている。その微生物開発を支えているのは、合成生物学、代謝工学、酵素工学といった学問である。それらの学問に基づいて、新たな化合物の発酵生産技術の開発や、生産性のラボスケールでの向上についての研究成果を多く見かけるようになったが、多くの化合物が市場に流通して社会実装されるには至っていないのが現状である。
筆者は公的な研究機関に所属しており、企業を研究支援するということが職場理念である。そのため、いかにして新しい化合物を生産するか、どのような新しい発想に基づく菌株開発を行うか、というアカデミアの新規性を重視する最先端研究よりは、いかに化合物を安価に作って社会実装するか、という進歩性を念頭において発酵生産菌の開発を行ってきた。そのために、菌株開発では高収量・高収率での生産に加え、培養をスケールアップした際の菌株の安定性を強く意識している。本トピックスでは、そのような研究スタンスの中で生まれた芳香族化合物の高生産大腸菌株について、開発の経緯やその利用例を含めて紹介させて頂く。
2.染色体DNAへの遺伝子導入方法の確立
2-1 プラスミドから染色体へ
微生物の代謝を改変し、目的とする化合物を高生産するためには、目的化合物への代謝フラックスを増大させる必要がある。そのためには、代謝の分岐点にあるような、合成経路上でキーとなる反応に関与する酵素をコードする遺伝子を高発現させる手法が一般的に用いられる。例えば、大腸菌での芳香族アミノ酸の生合成においては、ホスホエノールピルビン酸 (PEP) とエリスロース4-リン酸酸 (E4P) から3-デオキシ-D-アラビノヘプツロン酸7-リン酸酸 (DAHP) を生じる反応がキーである。DAHPシンターゼをコードするaroGやaroF遺伝子を大腸菌で高発現することで、著量のフェニルアラニンとチロシンをグルコースから生産する大腸菌株を簡単に作製することができる1,2)。大腸菌で遺伝子を高発現する手段としては、目的遺伝子 (Gene of Interest:GOI) をコードするプラスミドで大腸菌を形質転換体する方法が広く用いられている。筆者も、研究開始当初はプラスミドを用いて大腸菌にGOIを導入することで、芳香族化合物の高生産菌を作製しようと考えていた。しかし、2000年に報告されたJonesらの文献を見て、菌株作製の方針を大きく転換した。彼らの研究成果は、端的に言えば、バクテリアの代謝工学には高コピープラスミドよりも低コピープラスミドの方が適していますよ、という内容である3)。タンパク質の生産においてはコピー数の多いプラスミドがしばしば用いられるが、本論文ではpMB1系プラスミド (20~30コピー) を用いると菌体量と菌体当たりの物質生産量が低下するという結果が示されていた。代謝工学でmetabolic burden (代謝負荷) と呼ばれる現象である4)。一方、低コピーのプラスミド (mini-Fプラスミド、1~2コピー) を用いた場合には、プラスミドを用いていない場合と同等の菌体量が維持され、かつ物質生産量の向上が見られた。この研究結果から、筆者は、低コピーのプラスミドを用いるのであれば、工業的な菌株の利用も考慮して染色体DNAにGOIを導入した方がよいと考えた。
2-2 Red相同組み換え、P1形質導入、FLP/FRT部位特異的相同組み換えを利用した染色体導入法の確立
染色体DNAへの遺伝子導入法としては、相同組み換えを利用した方法が一般的である。2000年にDatsenkoとWannerが報告したλファージのRedリコンビナーゼを用いた大腸菌の遺伝子欠失方法は画期的であった5)。ダブルクロスオーバー (2か所の相同組み換え) による遺伝子欠失法では、欠失したい染色体座位に対応した相同性配列を導入カセットの両端に付加する必要がある。Red相同組み換えの最大の特徴は、この相同性配列がわずか50bpの長さで十分な点である。大腸菌のRecシステムを用いた場合、直線状のDNAフラグメントは大腸菌のRecBCDにより分解されるため、500~1,000 bp程度の比較的長い相同性配列を付加する必要がある。長い相同性配列が必要な場合、染色体DNA上の導入したい座位 (ターゲット座位) の遺伝子と導入するGOIを含む導入カセットを作製する必要がある (図1A)。すなわち、ターゲット毎に専用のプラスミドを構築するか、複数回のPCRで3種類のDNA断片をつなぎ合わせる必要がある。一方、相同性配列が50 bpになると、色体上のターゲット座位をプライマーの付加配列のみで設定することが可能となるため、1回のPCR増幅のみで導入カセットが作製できる (図1B)。この利便性を活用して、2006年にBabaらは大腸菌の網羅的1遺伝子欠失株ライブラリー (KEIOコレクション) を作製した6)。現在、KEIOコレクションは世界中で広く利用されている大腸菌の菌株ライブラリーのひとつである。さらにDatsenkoとWannerは、挿入する薬剤耐性遺伝子をフリパーゼ認識 (FRT) 配列で挟み込むことで、出芽酵母のフリパーゼ (FLP) を用いて染色体DNA上から薬剤耐性遺伝子を除去できるようにし、マーカーレスな欠失株を作製できるようにした。筆者は、このRedリコンビナーゼを用いた遺伝子欠失法が、遺伝子導入法として利用できると考えた。DatsenkoとWannerが利用していた遺伝子欠失カセットの下流に導入したいGOIを連結するだけの非常にシンプルな発想である (図1C)。実際に、Red相同組み換えを用いて、3 kbp程度のGOIを染色体DNA上のターゲット座位に問題なく導入できることを確認した。
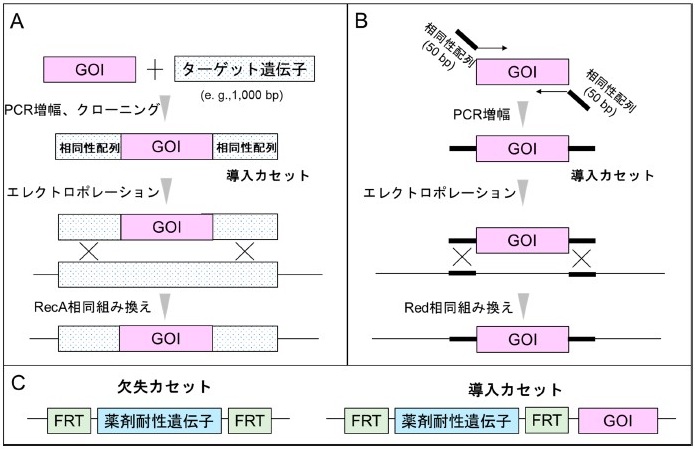

Red相同組み換えとFLP/FRT部位特異的相同組み換えを用いたマーカーリサイクリングにより、原理的には、複数のGOIを染色体DNA上の異なる座位に導入できると思われた。しかしながら、1つめのGOIを染色体DNAに導入した場合には、得られた薬剤耐性株は100%近い確率でGOIをターゲット座位に有していたのに対して、2つめのGOIを染色体DNA上の異なる座位に導入した場合には、そのGOIがターゲット座位に導入される確率は3割程度であった。さらに3つめのGOIを導入した場合には、ターゲット座位に導入される確率はゼロであった。これは、2回目のRed相同組み換えによりGOIを導入した際、遺伝子発現のためのプロモーターやターミネーターなどの配列が染色体DNA上に2か所導入されることになるため、3回目のRed相同組み換えでは先に導入されたそれらの配列と相同組み換えを起こしているのが原因であった。導入するGOIごとにプロモーターとターミネーターの配列をカスタマイズすることで問題回避できると考えたが、実験する上で非効率的であるために別の手段を検討し、P1形質導入を用いるに至った。P1形質導入は、染色体DNAの特定の位置に組み込まれるλファージなどの特異的形質導入とは異なり、染色体DNAへの導入位置が限定されない一般形質導入と呼ばれる。野生型大腸菌A株と変異型大腸菌B株がいる場合に、B株の遺伝的変異をA株に移すことができる。P1形質導入には、溶原化能を欠失させた変異型のP1kcファージが用いられる。P1ファージは、パッケージングされる際に0.1%程度の確率で大腸菌のDNA断片 (100 kbp程度) のみを取り込む7)。この不完全なファージは大腸菌への感染能を有しており、感染後、パッケージングしたDNA断片を宿主に導入する。導入されたDNA断片は、大腸菌内のRecAの働きにより染色体DNAとの相同組み換えを起こす。例えば、薬剤耐性遺伝子の近傍にGOI1を持つ菌株A (Red相同組み換えで作製した菌株) をP1ファージでライセート化し、GOI2を有する菌株Bに感染させれば、薬剤耐性を指標としてGOI1と2の両方を有する菌株Cが得られる (図2)。
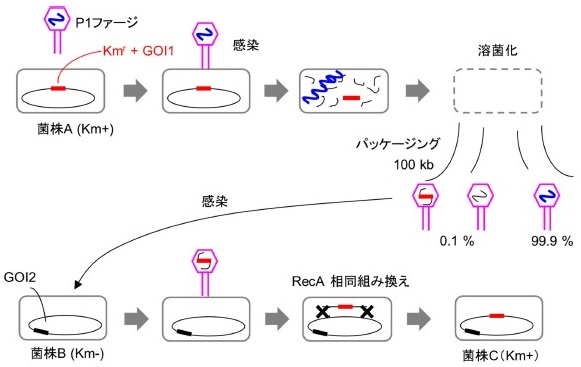
図2 P1ファージを用いた一般形質導
Red相同組み換え、FLP/FRT部位特異的相同組み換え、およびP1形質導入を利用して、薬剤耐性マーカーをリサイクルしながら、大腸菌の染色体DNAに順次GOIを導入する方法を確立した (図3)8)。最初に、染色体DNA上の異なる座位にGOIを導入した菌株をRed相同組み換えで作製しておく。例えば、A、B、またはC遺伝子を導入した菌株を作製しておき、A遺伝子を持つ菌株から、薬剤耐性遺伝子をFLP/FRT部位特異的相同組み換えにより削除しておく。一方、BとC遺伝子を持つ菌株は、それぞれP1ファージを用いてライセート化しておく。上記のA遺伝子を持つ菌株を受容菌株として、B遺伝子を持つ菌株のライセートを用いてP1形質導入し薬剤耐性株を選抜することで、染色体DNAにAとB遺伝子を持つ菌株を得ることができる。同様の手順を繰り返すことで、染色体DNA上にA、B、およびC遺伝子を持つ菌株が得られる。この方法を用いることで、染色体DNAに複数のGOIを有する安定株を作製することができる。実際に、筆者らは後述するプラスミドフリーなフェニルアラニンやチロシンの高生産株 (染色体DNA上の6か所に10種類のGOIを導入)、ヒドロキシチロソールの高生産株 (染色体DNA上の10か所に15種類のGOIを導入) を作製することに成功した9,10)。この方法の欠点は、2つ以上のGOIを染色体DNAに導入する場合に、近い距離にある座位には導入できない点である。P1形質導入の特性上、導入するGOIは少なくとも数万bp離れている必要がある (相同性配列が非常に長いため)。また、染色体DNA上に複数の傷 (FRT配列) が残るので、GOIが近い位置関係にあると、FLPを発現した際に近くのFRT配列間での意図せぬ組み換えが起こりやすい。筆者の経験上、2つのGOI間の距離を5~10万 bpほど確保するのが好ましい。
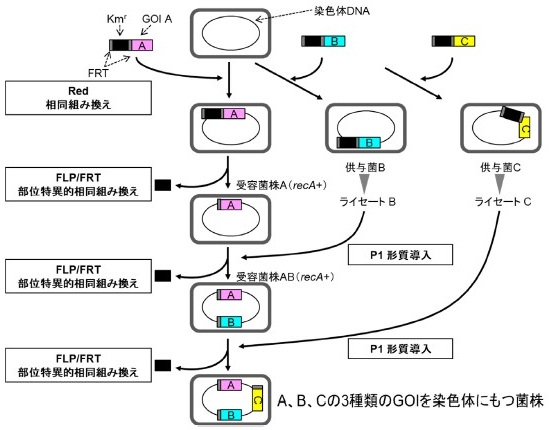
図3 複数のGOIを染色体に段階的に導入する方法。参考文献8の図を改変した。
2-3 1コピーでの高発現を目指して
~T7発現系の利用~
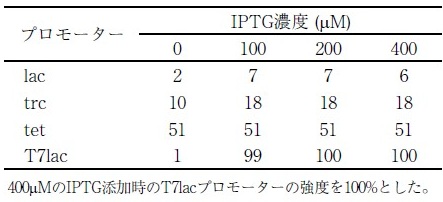
GOIの発現量は、転写レベルでは、遺伝子のコピー数とプロモーターの強度の積算で決定される。よって、1コピーの染色体導入株で十分な発現量を得るために、プロモーターの検討を行った。その際、筆者らは大腸菌で最も高い転写活性を示すT7プロモーター/T7 RNAポリメラーゼを用いた発現系 (T7発現系) を利用した8)。T7発現系は、T7ファージ由来の遺伝子発現システムであり、タンパク質の生産のためにpET系のプラスミドベクターなどで利用されている11)。この発現系は、lacプロモーターなどのコンベンショナルなプロモーターを利用してT7 RNAポリメラーゼ遺伝子を一旦発現し、続いてT7 RNAポリメラーゼとT7プロモーターを利用してGOIを高発現する、2段構えの発現系である (図4)。従来はプラスミドで用いられていたT7発現系を染色体導入株へと応用し、遺伝子発現や物質生産について、コンベンショナルなプロモーターを用いた場合と比較した。まず、sfGFPをlac、trc、tet、またはT7lacプロモーター (T7プロモーター下流にlacオペレーターが配置した構造) に連結し、大腸菌の染色体DNAの同じ座位に1コピー導入した。次に、培養しながらGFPの蛍光を測定し、各プロモーターの強度 (蛍光強度/培養液濁度) を測定・比較した。その結果、T7lacプロモーターはコンベンショナルなプロモーターの2~14倍の強度であり (表1)、染色体導入株にT7発現系を利用することで、通常のコンベンショナルなプロモーターを利用するよりもGOIを高発現できることが示唆された。
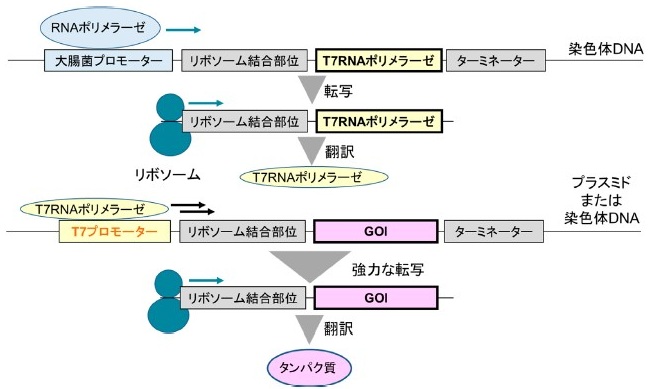
図4 T7発現系を用いたGOIの発現
表1 相対的なプロモーター強度 (%)
次に、βガラクトシダーゼ (LacZ) を用いたレポーターアッセイにより、導入するGOIのコピー数と発現量の関係について調べた。LacZの活性は、位置効果により、導入した染色体DNAの座位間で若干のバラつきがあった。1コピーの導入では、pET系ベクター (20~40コピー) を用いた場合の29~52%の活性値であった8)。FLP/FRT部位特異的相同組み換えとP1形質導入により、lacZ遺伝子の染色体導入コピー数を増加させていくと、4~5コピーでLacZ活性はプラトーに達した (図5A)。この時の活性値は、pET系ベクターを用いた場合と同等以上であった。SDS-PAGEで発現解析したところ、1コピーの導入で、クマシー染色で確認できるレベルでのタンパク質が生産され、コピー数の増加に応じてタンパク質量の増加が確認できた (図5B)。また、異種生物由来の遺伝子の発現についても確認した結果、lacZ遺伝子と同様に、1コピーでの高発現が確認された (図5C)。2種類の遺伝子を同時に高発現させた場合には、対応する2種類のタンパク質の高生産が確認された。これらの結果から、染色体DNAへの遺伝子導入とT7発現系との組み合わせは、少なくともタンパク質の生産レベルでは利用可能であることが明らかとなった。
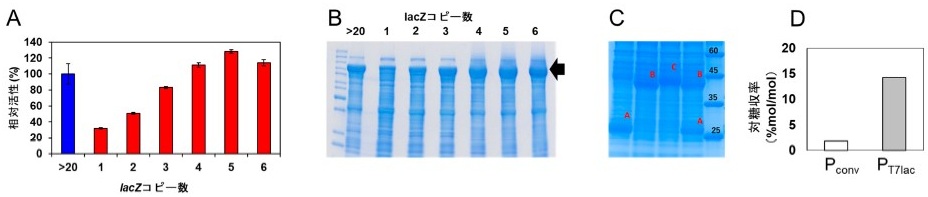

次に、T7発現系が発酵生産菌株の開発に有効であるかどうかを検証した。2012年にJuminagaらによって報告されたプラスミド型のチロシン生産株を作製する遺伝子セット用いて、染色体導入型のチロシン生産株を作製した12)。各遺伝子の発現には、文献記載のコンベンショナルなプロモーターセット (lac、trc、およびtet) またはT7lacプロモーターを用いた。その結果、コンベンショナルなプロモーターセットを用いた場合と比較して、T7lacプロモーターを用いた場合にはチロシンの対糖収率が7.7倍に向上した (図5D)。
3.芳香族化合物の高生産シャーシ株の作製とその利用
3-1 さまざまな芳香族化合物を生産する大腸菌株の作製
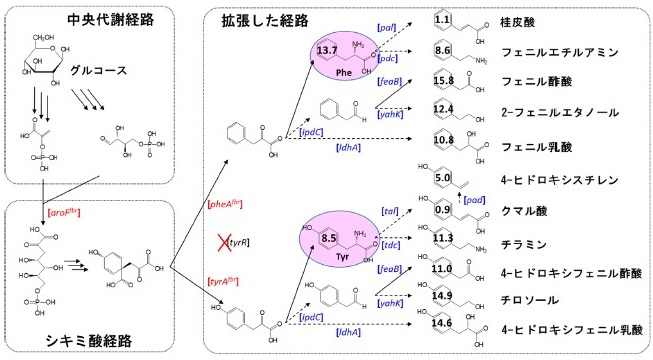
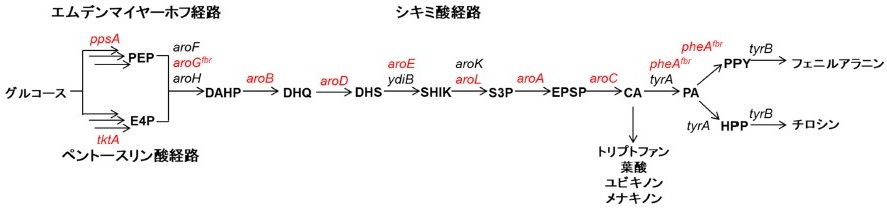
アミノ酸発酵は工業的に成功している代表的な発酵生産のひとつである。芳香族アミノ酸であるフェニルアラニンとチロシンも例外ではない。これまでに、大腸菌のさまざまな遺伝子改変についての多くの知見が蓄積されている13-15)。よって筆者は、フェニルアラニンとチロシンの合成経路を利用することで、さまざまな芳香族化合物を高収量・高収率で生産できると考えた。基本的な研究コンセプトは、外来生物種の遺伝子を用いて、フェニルアラニンまたはチロシンの発酵生産経路を拡張することで、さまざまな芳香族化合物を糖質原料から発酵生産するというものである。そこで、上述の染色体DNAへの遺伝子導入法を用いて、阻害脱感作したDAHPシンターゼ遺伝子 (aroFfbr、fbrはfeedback resistance) とコリスミ酸ムターゼ/プレフェン酸デヒドラターゼ遺伝子 (pheAfbr) またはコリスミ酸ムターゼ/プレフェン酸デヒドロゲナーゼ遺伝子 (tyrAfbr) を導入することで、フェニルアラニンまたはチロシン高生産株を作製した。次に、フェニルアラニンやチロシン、およびその前駆体であるフェニルピルビン酸や4-ヒドロキシフェニルピルビン酸から派生する化合物を調査し、関連する遺伝子情報を収集した。人工遺伝子合成があまり普及していない時代だったため、それらの遺伝子を有する菌株をカルチャーコレクションから取り寄せて培養し、ゲノムDNA調製後にPCR増幅等により遺伝子を取得し、大腸菌での発現や活性の有無を確認した後にフェニルアラニンまたはチロシン高生産株に導入した。その結果、さまざまな芳香族化合物をグルコースから発酵生産することに成功した (図6)16)。具体的には、新規な発酵生産菌として、フェニルエチルアミン、フェニル酢酸、フェニル乳酸、チラミン、4-ヒドロキシフェニル酢酸、4-ヒドロキシフェニル乳酸の発酵生産株を作製した。また、2-フェニルエタノール、チロソールについても、既報の菌株よりも高い収率で発酵生産する菌株を作製することに成功した。これらの芳香族化合物の対糖収率は~15.8%mol/molであった。フェニルアラニンとチロシンの対糖収率が~13.7%であったことから、より高い収率を達成するためには、フェニルアラニンとチロシンの対糖収率の向上が必要であると考えた。

3-2 芳香族化合物を高生産するためのシャーシ株の作製
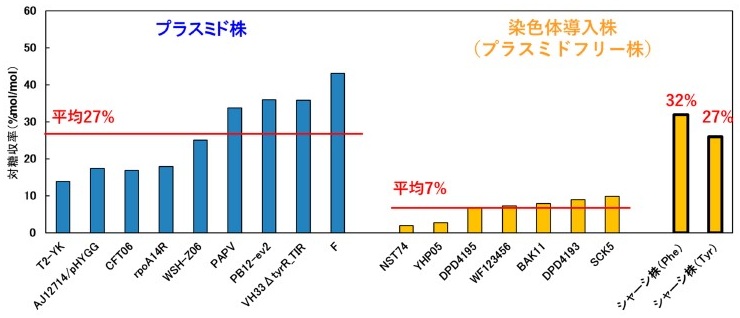
さまざまな芳香族化合物を高生産することを目的として、フェニルアラニンとチロシンの生産能をより高めたシャーシ株の作製を目指した。シャーシ株はプラスミドを用いてさまざまな遺伝子を導入するための宿主となる可能性があるため、プラスミドの不和合性を考慮する必要のない「染色体導入株 (プラスミドフリー株) 」であることが開発条件のひとつであった。目指すべき対糖収率の数値を設定するべく、これまでに報告されているフェニルアラニンとチロシンの対糖収率について、プラスミド株とプラスミドフリー株とに分けて比較した (図7)9)。驚くべきことに、それぞれのグループで対糖収率の平均値を算出したところ、プラスミド株が27%mol/molであったのに対して、プラスミドフリー株はわずか7%mol/molであった。そこで、プラスミドフリー株でプラスミド株並みの対糖収率を達成することを目標として、シャーシ株の開発に着手した。
![]()
既知の報告では、フェニルアラニンやチロシンの対糖収率を高めるためには、シキミ酸経路の遺伝子の高発現のみならず、中央代謝経路の改変が重要であることが示されていた。具体的には、DAHP合成のための基質となるPEPとE4Pの細胞内在量を高めることである。PEPに関しては、大腸菌はPEP依存型のグルコース取り込み機構 (PTS) によりグルコースを細胞内に取り込むことが知られており、1分子のグルコースを取り込む度に1分子のPEPがピルビン酸へと変換される17)。変換されたピルビン酸は、アセチルCoAを経由してTCAサイクルに流れ込むため、フェニルアラニンやチロシンの合成に利用することができない。よって、PEPの内在量を向上させるために、PTSではないグルコースの取り込み機構を利用することや (ptsG:glucose-specific PTS enzyme IIBC component遺伝子の欠失、galP:galactose permease遺伝子の高発現など)、ATPを使いピルビン酸をPEPに再生すること (ppsA:PEP synthetase遺伝子の高発現) が有効な手段であることが知られていた。グルコースに対するフェニルアラニンとチロシンの理論最大収率は、上述の手段により2倍向上し、60%mol/molに達する18)。一方、E4Pについては、細胞内在量の低さがDAHP合成反応の律速段階となっていることが報告されていた18)。E4Pの供給方法として、ペントースリン酸経路 (PPP) に含まれるtktA (transketolase) 遺伝子を高発現させることが最も有効と考えられている。そこで、aroGfbrとpheAfbrを染色体DNAに導入した菌株に、さらにppsAとtktA遺伝子を導入し、フェニルアラニンを高生産する菌株の作製を試みた。しかしながら、tktA遺伝子を導入して高発現すると、菌株の生育が極端に悪くなり、さらにフェニルアラニンの対糖収率は大幅に低下した (図8)。tktA遺伝子の高発現に用いるプロモーターに、活性の低下した変異型T7lacプロモーターなども用いて発現量と対糖収率との関係を調べたところ、プロモーター活性の向上に伴ってフェニルアラニンの対糖収率の低下が認められた。よって、tktA遺伝子の過剰発現が代謝負荷を引き起こしていると考えられた。
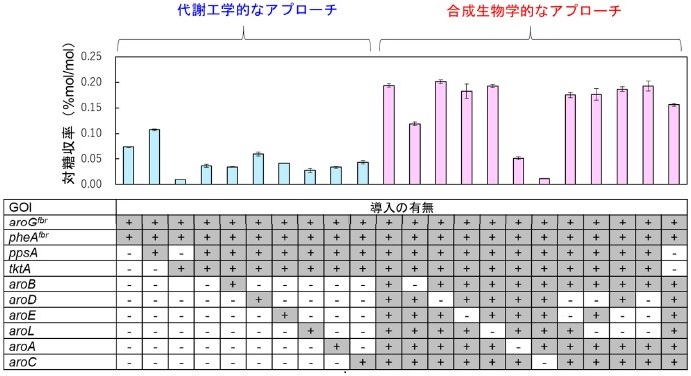
図8 GOIの導入パターンの違いによるフェニルアラニンの対糖収率の違い
既報のプラスミド菌株の文献を調査したが、tktA遺伝子の発現による代謝負荷についての原因を見つけることができなかった。そこで、tktA遺伝子を高発現させても高い対糖収率でフェニルアラニンを生産する菌株を作製することを目指した9)。T7lacプロモーターに連結した4つの遺伝子、aroGfbr、pheAfbr、ppsA、およびtktAを染色体DNAに導入し、さらに追加で遺伝子を導入し、フェニルアラニンの対糖収率が向上する菌株を探索した。シキミ酸経路に含まれる、aroB、aroD、aroE、aroL、aroA、およびaroC遺伝子をそれぞれ導入して対糖収率を測定したが、対糖収率が向上する菌株は得られなかった (図8)。また、シキミ酸経路ではなく、エムデン-マイヤーホフ経路 (EMP) やPPPのさまざまな遺伝子の高発現や欠失を試みたが、高収率で生産する菌株は得られなかった。そこで、律速段階となる反応を探索し改善する代謝工学的なアプローチではなく、システムを一度組み上げて検証する合成生物学的なアプローチを取り入れた。具体的には、aroGfbr、pheAfbr、ppsA、およびtktAの4遺伝子に加えて、シキミ酸経路のaroB、aroD、aroE、aroL、aroA、およびaroCの6遺伝子を同時に染色体に導入して菌株を作製した (図9)。さらに、それらの遺伝子を1つずつ欠失させることで、高収率に必要な遺伝子セットの同定を試みた。10遺伝子を導入した菌株は、高い対糖収率でフェニルアラニンを生産した (図8)。さらに、1つずつ遺伝子を削除した結果、aroA、aroB、およびaroCの3遺伝子の導入が高収率に必須であることが明らかとなった。ppsAとtktA遺伝子を削除した場合には、対糖収率が低下したことから、ppsAとtktA遺伝子の導入も高収率に寄与していた。最終的に、ppsAとtktA遺伝子を導入して高発現し、20%mol/mol以上の対糖収率でフェニルアラニンを生産する菌株を作製することに成功した。フェニルアラニン生産株の作製に用いたpheAfbr遺伝子をtyrAf br遺伝子に置換することで、高収率でチロシンを生産する菌株も作製した。さらに、既報の文献を参考にして培地に若干の改良を加えることで、フェニルアラニンとチロシンの対糖収率は約30% mol/molに達し、プラスミドフリー株でプラスミド株並みの対糖収率で生産するフェニルアラニンとチロシンの生産株の開発に成功した (図7)。

次に、作製した菌株が、さまざまな芳香族化合物を発酵生産するためのシャーシ株として利用できるかどうかを検証した。フェニル乳酸、4-ヒドロキシフェニル乳酸、チラミン、およびチロソールの合成に必要な遺伝子セットをシャーシ株の染色体DNAに導入し、それぞれの化合物を発酵生産する菌株を作製した。従来のaroFfbr-pheAfbrのみを高発現した菌株と比較して、各化合物の対糖収率は2倍程度向上し、20~30%mol/molに達した9)。
作製したシャーシ株はフラスコレベルのバッチ培養で高い対糖収率を示すことが実証されたが、ミニジャーファメンターを用いたフェドバッチ培養での収量 (g/L) は未検証であった。遺伝子組み換えにより作製した大腸菌株は、フラスコ培養では目的化合物を効率的に生産するが、ジャーファメンターで培養した際には増殖が悪く、目的化合物の生産性が低いことがしばしば見受けられる。そこで、3Lのミニジャーファメンターを用いたグルコースの流加培養を行い、シャーシ株の増殖、フェニルアラニンの収量、および対糖収率の測定を行った。その結果、菌株は良好に増殖し、フェニルアラニンの収量は、72時間で76 g/Lに達した (図10)。対糖収率は30%mol/molであった。対糖収率がフラスコ培養と同等であることから、シャーシ株はスケールアップ培養にも十分対応可能な菌株であることが実証された。フェニルアラニンに関しては、天津大学のLiuらが2018年に72.9 g/L、江南大学のWangらが2024年に80.48 g/Lの収量で発酵生産することに成功している19,20)。本菌株は、芳香族化合物生産のためのシャーシ株であるためにフェニルアラニンの排出系の強化等は行っていないが、それらの菌株と同レベルでの収量でフェニルアラニンを生産することから、非常に優れたシャーシ株になりえると考えられた。
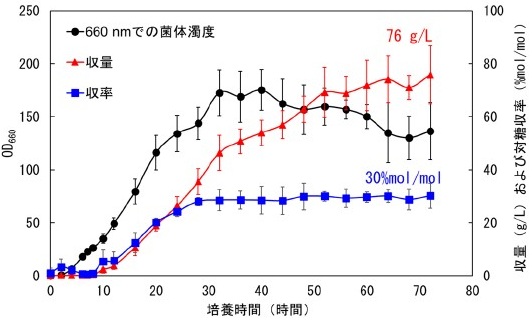
図10 シャーシ株の3Lジャーファメンターを用いた流加培養 (Phe生産)
3-3 シャーシ株を利用したヒドロキシチロソール発酵生産菌の開発
ヒドロキシチロソール (HT) はオリーブ由来のポリフェノールであり、アスコルビン酸 (ビタミンC) の20倍以上の抗酸化活性を有する化合物である。HTはオリーブ中の健康成分と言われており、生理学的な機能についての非常に多くの研究が行われている。HTの機能として、動脈硬化・血栓形成予防、LDL酸化抑制、メラニン生成抑制作用、心臓・神経・細胞保護作用、高脂血症改善、美白作用、抗炎症・抗腫瘍・抗ガン効果などの多様な機能、また抗菌活性や抗ウィルス活性が知られている21-28)。HTは化粧品原料、食品添加物、栄養補助食品として利用されている。現在市場に流通しているHTは、オリーブ油抽出残渣から抽出・精製したもの、または化学合成したものである29)。しかしながら、アスコルビン酸 (3,000円/kg程度) などと比較すると、生産量が少なく非常に高価 (100,000円/kg) であるため、市場に十分量を供給できないのが現状である。これまでに、安価な製造法を目指して、グルコースを原料としたHTの発酵生産法の開発が行われていたが、筆者が研究を開始した2019年当時、文献値のHTの収量はわずか1 g/L未満であり、実用化を目指す上では十分な値とは言えなかった30,31)。
そこで、シャーシ株を用いて、HTを高収量で発酵生産する菌株の開発に着手した。シャーシ株では染色体DNAの6か所に10種類の遺伝子を導入しているが、HT生産株では必要な遺伝子をさらに追加で導入し、最終的に染色体DNAの10か所に15種類の遺伝子 (17遺伝子) を導入した (図11A)10)。簡単に説明すると、シャーシ株の菌体内にチロソールの合成経路を構築し、さらにチロソールをHTに変換するモノオキシゲナーゼを導入することで、HTを発酵生産する大腸菌を作製した (図11B)。同時に、ヒドロキシチロソールの生産量を低下させるようないくつかの遺伝子を欠失させた (tyrB:tyrosine aminotransferase とfeaB:phenylacetaldehyde dehydrogenase)。作製した菌株をジャーファメンターで培養したところ、およそ9 g/Lの収量でHTを発酵生産することに成功し、従来の収量値を大きく更新した (図12)。しかし、HTは抗菌活性を有する化合物であり、フェニルアラニンと比較して大腸菌に対する毒性が高く、フェニルアラニン並みの収量では発酵生産できていない。現在は、HTのさらなる収量向上に取り組みながら、糖鎖を付加した低毒性かつ活性を保ったHT誘導体の発酵生産にも取り組んでいる。
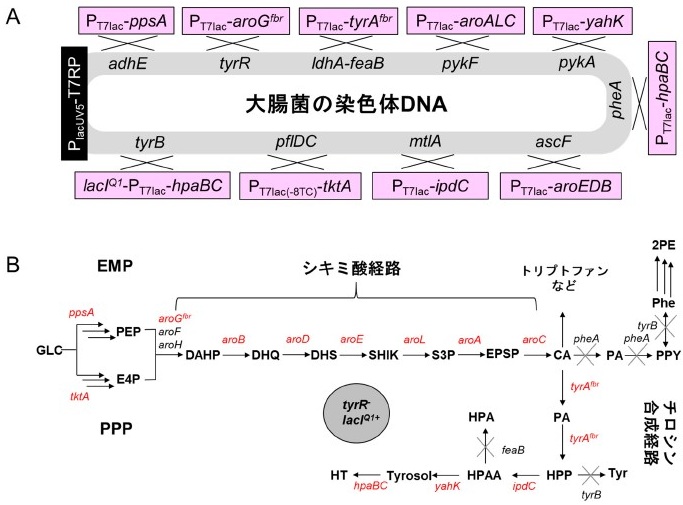

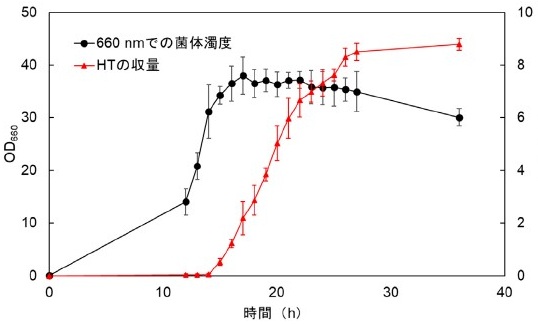
図12 シャーシ株から派生したHT生産菌の3 Lジャーファメンターを用いた流加培養
4.おわりに
筆者は、2015年に合成生物学の大家であるUCバークレーのJay D. Keasling教授がCEOを務めるJoint BioEnergy Instituteで研究に従事した。そこでは、10種類程度の遺伝子を2つのプラスミドを用いて発現させる「2プラスミドシステム」と呼ばれる方法を用いて有用化合物の生産株を作製していた。留学時は、それらの遺伝子を染色体DNAに導入して、同等以上の生産能を有する菌株を開発する研究が行われていた。また、2019年に、菌株の育種技術では国内有数の技術とノウハウを持っている某王手食品会社の菌株の育種部門の方と、菌株開発についてのお話しをさせて頂く機会があった。その際、遺伝子を染色体DNAに導入するのは基本です、といわれたことを覚えている。2025年となった今、染色体DNAへ遺伝子を導入した安定生産株の開発は、世界中の多くの研究機関で行われている。特に、CRISPR-Cas9の登場により、染色体DNAに傷のない安定導入株が作製できるようになったことの影響が大きいと思われる。
染色体DNAに遺伝子を導入した菌株は、培養コストの低減 (抗生物質が不要) と遺伝子の安定保持が利点である。コスト低減に関しては、ラボスケールでの実験ではあまり気にはならないが、発酵槽をkLスケールにすればそのメリットを感じる。リン酸緩衝液にグルコース、硫酸アンモニウム、マグネシウム、微量金属、チアミン、抗生物質等を加えた大腸菌用の合成培地を用い、遺伝子発現をIPTGで行った場合、各原料の単価を用いて培地コストを計算すると、~80円/Lとなる (2019年度版17019の化学商品や試薬価格表に基づく粗い試算)。このうち、20円くらいが抗生物質の価格となる。この場合、例えば150 kLのタンクで仕込み100 kLで培養した場合には、ザクっとした計算で、培地コストは1,000万円、うち抗生物質のコストは200万円となる。各原料について、試薬とバルク品ではそもそもの単価の違いはあるが、参考程度にはなると思う。ちなみに大型タンクで培養した場合には、IPTGの試算コストも数百万円となるため (1,000円/gの安価なバルク品を用いた場合でも)、高コストなIPTG誘導型の遺伝子発現スイッチを、安価なスイッチに変えることも検討している32)。近年では、コストと汎用性に優れる光スイッチのような物理刺激スイッチの導入も検討している33)。
芳香族化合物の高生産シャーシ株 (フェニルアラニンおよびチロシンの高生産株) の開発が成功したのは、上述のKeasling教授の研究室への留学のおかげである。それまでは、本文に記載のように、代謝工学的な視点からの菌株開発を行っており、合成経路中の律速反応を決定するということに注力を置いていた (その結果、数年間は研究に行き詰っていた)。Keasling教授の研究室は合成生物学の研究室で、2プラスミドシステムを用いて、関連する合成経路のすべての遺伝子を大腸菌に導入するという手段が用いられていた。筆者は、染色体導入型のフェニルアラニンとチロシンの生産株の開発にその手段を転用したに過ぎない。余談ではあるが、芳香族化合物のシャーシ株は公開後、国内外の多くのアカデミア機関にMTA締結後に無償分譲してきた。近年、ポルトガルのMinho大学のGomesらは、構築したプラスミドで本シャーシ株を形質転換し、著量のナリンゲニンをde novo合成することに成功している34)。今後も、本菌株がアカデミアや企業の多くの研究開発に貢献できれば幸いである。
謝辞
本稿にて詳細した研究成果は、JSPS科研費 (08055255、09020698、15K07402)、JST A-STEP (JPMJTM19CC、JPMJTM20QJ、JPMJTM23U4)、およびJST GteX (JPMJGX23B4) の支援を受け実施したものです。ジャーファメンターでの培養実験は、大阪工業大学の長森英二教授との共同研究にて実施しました。ここに感謝いたします。
文献
1) Zhou, H., Liao, X., Wang, T., Du, G., Chen, J.: Bioresour Technol, 101, 4151 (2010).
2) Santos, C. N., Xiao, W., Stephanopoulos, G.: Proc Natl Acad Sci USA, 109, 13538 (2012).
3) Jones, K. L., Kim, S. W., Keasling, J. D.: Metab Eng, 2, 328 (2000).
4) Flores, S., de Anda-Herrera, R., Gosset, G., Bolivar, F. G.: Biotechnol Bioeng, 87, 485 (2004).
5) Datsenko, K. A., Wanner, B. L.: Proc Natl Acad Sci USA, 97, 6640 (2000).
6) Baba, T., Ara, T., Hasegawa, M., Takai, Y., Okumura, Y., Baba, M., Datsenko, K. S., Tomita, M., Wanner, B. L., Mori, H.: Mol Syst Biol, 2, 2006 (2006).
7) Thomason, L. C., Costantino, N., Court, D. L.: Curr Protoc Mol Biol, Chapter 1, Unit 1 17 (2007).
8) Koma, D., Yamanaka, H., Moriyoshi, K., Ohmoto, T., Sakai, K.: Appl Microbiol Biotechnol, 93, 815 (2012).
9) Koma, D., Kishida, T., Yoshida, E., Ohashi, H., Yamanaka, H., Moriyoshi, K., Nagamori, E., Ohmoto, T.: Appl Environ Microbiol, 86, e00525-20 (2020).
10) Koma, D., Fujisawa, M., Ohashi, H., Yamanaka, H., Moriyoshi, K., Nagamori, E., Ohmoto, T.: J Agric Food Chem, 71, 9451 (2023).
11) 東端啓貴: 生物工学会誌, 91, 96 (2013).
12) Juminaga, D., Baidoo, E. E., Redding-Johanson, A. M., Batth, T. S., Burd, H., Mukhopadhyay, A., Petzold, C. J., Keasling, J. D.: Appl Environ Microbiol, 78, 89 (2012).
13) Bongaerts, J., Kramer, M., Muller, U., Raeven, L., Wubbolts, M.: Metab Eng, 3, 289 (2001).
14) Sprenger, G. A.: Appl Microbiol Biotechnol, 75, 739 (2007).
15) Hirasawa, T., Satoh, Y., Koma, D.: World J Microbiol Biotechnol, 41, 65 (2025).
16) Koma, D., Yamanaka, H., Moriyoshi, K., Ohmoto, T., Sakai, K.: Appl Environ Microbiol, 78, 6203 (2012).
17) Carmona, S. B., Moreno, F., Bolivar, F., Gosset, G., Escalante, A.: J Mol Microbio. Biotechnol, 25, 195 (2015).
18) Patnaik, R., Liao, J. C.: Appl Environ Microbiol, 60, 3903 (1994).
19) Liu, Y., Xu, Y., Ding, D., Wen, J., Zhu, B., Zhang, D.: BMC Biotechnol, 18, 5 (2018).
20) Wang, X., Qiu, C., Chen, C., Gao, C., Wei, W., Song, W., Wu, J., Liu, L., Chen, X.: J Agric Food Chem, 72, 11029 (2024).
21) Bertelli, M., Kiani, A. K., Paolacci, S., Manara, E., Kurti, D., Dhuli, K., Bushati, V., Miertus, J., Pangallo, D., Baglivo, M., Beccari, T., Michelini, S.: J Biotechnol, 309, 29, (2020).
22) Karković Marković, A., Torić, J., Barbarić, M., Jakobušić Brala, C.: Molecules, 24, 2001 (2019).
23) Medina-Martínez, M. S., Truchado, P., Castro-Ibáñez, I., Allende, A.: Biosci Biotechnol Biochem, 80, 801 (2016).
24) Bedoya, L. M., Beltrán, M., Obregón-Calderón, P., García-Pérez, J., de la Torre, H. E., González, N., Pérez-Olmeda, M., Auñón, D., Capa, L., Gómez-Acebo, E., Alcamí, J.: AIDS, 30, 2767 (2016).
25) Peyrol, J., Riva, C., Amiot, M. J.: Nutrients, 9 (2017).
26) Robles-Almazan, M., Pulido-Moran, M., Moreno-Fernandez, J., Ramirez-Tortosa, C., Rodriguez-Garcia, C., Quiles, J. L., Ramirez-Tortosa, M.: Food Res Int, 105, 654 (2018).
27) Kiani, A. K., Dhuli, K., Anpilogov, K., Bressan, S., Dautaj, A., Dundar, M., Beccari, T., Ergoren, M. C., Bertelli, M.: Acta Biomed, 91, e2020008 (2020).
28) de Las Hazas, M. C. L., Rubio, L., Macia, A., Motilva, M. J.: Curr Pharm Des, 24, 2157 (2018).
29) Britton, J., Davis, R., O’Connor, K. E.: Appl Microbiol Biotechnol, 103, 5957 (2019).
30) Li, X., Chen, Z., Wu, Y., Yan, Y., Sun, X., Yuan, Q.: ACS Synth Biol, 7, 647 (2018).
31) Trantas, E., Navakoudis, E., Pavlidis, T., Nikou, T., Halabalaki, M., Skaltsounis, L., Ververidis, F.: PLoS One, 14, e0212243 (2019).
32) Koma, D., Kishida, T., Yamanaka, H., Moriyoshi, K., Nagamori, E., Ohmoto, T.: J Biosci Bioeng, 126, 586 (2018).
33) Kawano, F., Okazaki, R., Yazawa, M., Sato, M.: Nat Chem Biol, 12, 1059 (2016).
34) Gomes, D., Rodrigues, J. L., Rodrigues, L. R.: Appl Microbiol Biotechnol, 108, 435 (2024).