【トピックス】
フィールドワークx 情報解析から知る野生酵母資源の機能多様性
清家泰介
九工大院・情報工、JST・さきがけ
1.はじめに
酵母は古くから人類の生活に深く関わってきた微生物であり、酒類や発酵食品の製造をはじめ、近年ではバイオ燃料や高付加価値化学品の生産、医薬品合成、酵素工学など、多様な分野で利用されている。一方で、現在研究や産業利用の対象となっている酵母は、限られたモデル株や産業株に偏っており、自然界に生息する野生酵母の大部分は未利用・未解明のままである。野生酵母は過酷な環境条件や特殊な栄養源に適応してきた進化的背景をもち、その中には既存株には見られない代謝経路や耐性機構、さらには未知の酵素を有する可能性が高い。しかし、野生酵母の体系的な探索と機能解明は、採集の偶発性や培養・同定の難しさから十分に進んでいないのが現状である。
私たちは、この課題を克服するために、フィールドワークを通じた野生酵母資源の収集に取り組んできた。特に昆虫、なかでもショウジョウバエを媒介とした酵母収集法は、自然界で実際に植物や果実と関わりながら生きる酵母群を効率的に取得できる有効な戦略である。本稿では、この手法を用いて得られた野生酵母ライブラリの概要、未知種の発見例、表現型解析の結果、そしてバイオインフォマティクスを駆使した利活用の可能性について紹介する。
2.野生酵母の取得とライブラリ構築
2-1 そもそも酵母とは何か
酵母は多くの人にとって身近な微生物であり、酒やパン、味噌など発酵食品の製造に欠かせない存在である。しかし「酵母とは何か」と問われると、その答えは意外に難しい。酵母とは、生活環の一部に単細胞世代をもち、出芽または分裂によって増殖する真菌の総称である。現在、少なくとも2,200種以上が知られている。意外かもしれないが、酵母は正式な分類群の名称ではなく、生活型を示す呼称である。真核微生物でありながら遺伝的系統は多岐にわたり、子嚢菌門と担子菌門の2門にまたがって存在する。さらに少なくとも6亜門─サッカロミケス亜門 (Sacchamycotina)、タフリナ菌亜門 (Taphrinomycotina)、チャワンタケ亜門 (Pezizomycotina)、ハラタケ亜門 (Agaricomycotina)、サビキン亜門 (Pucciniomycotina)、クロボキン亜門 (Ustilaginomycotina) ─に酵母型の種が含まれる1)。
酵母は地球上のあらゆる場所に生息し、果実や植物、土壌、海水、動物の表面や昆虫の体内など、多様な環境で見つかる。むしろ「いない環境を探す方が難しい」とさえ言える。一方で、酵母が自然界でどこに生息し、どのような生活史を送っているかという生態学的知見は未だ十分ではない。酵母はそれぞれの環境において、温度、pH、栄養源の有無といった環境要因に加え、他の生物との相互作用を含む複合的な要因の中で生活している。したがって、酵母の生息環境を知り、その多様性を理解することは、酵母が持つ機能を解明し、その利活用につなげるうえで欠かせない。
2-2 自然界からの野生酵母の取得
酵母は「どこにでもいる」と言われるが、実際に多様な種類を集めるのは容易ではない。そこで私たちは、ショウジョウバエを媒介して酵母を集めるアプローチを考案した。体長わずか数mmのショウジョウバエは、屋外を飛び回り果実や植物に集まり、主な餌として自然界の酵母からアミノ酸、ビタミン、脂質などを摂取している2,3)。興味深いことに、その消化管には「クロップ」と呼ばれる袋状の器官があり、酵母が一時的に蓄えられる4)。言い換えれば、このクロップは野生酵母の宝庫であると考えられる。
この発想から、私は約5年前にショウジョウバエを採取し、その体内から酵母を単離する試みを始めた。採集方法は比較的簡単で、近所のスーパーで購入したバナナをしばらく熟成させ、それを牛乳パックなどの容器に入れて木に吊るす (図1)。翌日にはどこからともなくショウジョウバエが集まり、容器内に入っている。経験的には7〜8割の確率で採集に成功し、条件が良ければ100匹以上を得られることもある。得られたハエは、容器の上をジップロックで覆い、軽く叩いて上方に逃がすことで回収する。遠方での採集ではCO2ガスで一時的に麻酔をかけ、素早くハエを培地に移す。研究室に持ち帰ったハエから微生物を取り出し、酵母を単離する。当然、バクテリアも混在するため、アンピシリンやクロラムフェニコールなどの抗生物質を加えた酵母用寒天培地に希釈して撒く。


ここからがやや難しい。酵母はカビとは異なり形態的な差が乏しく、コロニーの色が黄色や赤色を呈することもあるが、それだけでは種を見分けるのは困難である。経験を頼りに形態や色調が異なりそうなコロニーを選び出し、ストックする。最終的な同定にはDNA解析が有効であり、かつては発酵能力、炭素源や窒素源の資化能、細胞形態観察などの生理学的特徴が主流であった (現在も重要) が、現在ではリボソームRNA大サブユニット (26S rDNA) D1/D2領域やITS領域の配列を決定すればおおよその種判定が可能である5)。ただし、これは簡易的な方法であり、確定的な同定には生理学的性質を含めた総合的判断が必要である。こうしてショウジョウバエを中心に、果物や植物、土壌など多様な環境から採集を行い、研究者や一般協力者の力を得ながら、これまでに2,000クローン以上の酵母株を取得した。この成果は、酵母があらゆる環境に存在すること、そしてショウジョウバエを利用した採集が野生酵母収集の有効的な手段であることを示している。
2-3 独自に採集した野生酵母株ライブラリの特徴
自然界から酵母を単離し、それを解析や商品化に使用する事例は少なくない。「天然酵母のパン」や「地元の酵母で作った酒」などはよく耳にするフレーズだろう。それでも、なぜわざわざ自ら酵母を探すのか。日本にはナショナルバイオリソースプロジェクト (NBRPの酵母:https://yeast.nig.ac.jp/yeast/about/index.xhtml)、理化学研究所・微生物材料開発室 (JCM:https://jcm.brc.riken.jp/ja/)、独立行政法人・製品評価技術基盤機構 (NITE:https://www.nite.go.jp) などの寄託機関があり、海外にもオランダのWesterdijk Fungal Biodiversity Institute (https://wi.knaw.nl) のように6万株以上の酵母・糸状菌株を保有する機関がある。にもかかわらず、自ら採集する理由は、身近な環境にも今まで知られていなかった株が存在し、同じ種でも性質が異なる場合が多いからである。私はこの4月に九州工業大学で研究室を立ち上げ、前任校から来てくれた学生や研究員とともに、この6月に鹿児島で野生酵母の探索を行った (図2)。桜島周辺の植物や土壌から少なくとも23種以上の酵母を単離し、その中には未記載種も含まれていた。執筆時点 (2025年8月) までに、少なくとも86属231種の酵母株を収集しており、この中には先述の6亜門すべてが含まれる。未記載種は約50種に達する。


私はこれまでに3種の新種酵母の認定を受けた6-8)。特に沖縄県のショウジョウバエから単離されたHanseniaspora drosophilae sp. nov.は、琉球大学や沖縄科学技術大学院大学周辺でショウジョウバエからのみ繰り返し分離され、周辺の植物や花からは見つからないことから、極めてニッチな環境に適応している可能性がある8)。新種認定には、外見だけでは種を識別できない微生物の特性上、DNA配列解析と詳細な生理学的試験が不可欠である。まずD1/D2領域やITS領域の配列を既知種と比較し、類縁度が一定以上異なれば新種の可能性が高まるが、DNAだけでは決め手にならない。そこで糖発酵能、利用可能なエネルギー源、増殖温度など70項目以上の生理学的・生化学的特性を調べ、顕微鏡で形態や有性生殖の有無、菌糸形成の有無などを確認する。これらの総合的な差異によって近縁種と区別できれば新種として認定される。この種はシクロへキシミドに対して極めて高い耐性を示し、他属の酵母より利用可能な炭素源が広く、発酵能も高いため産業利用の可能性がある8)。
さらに、ショウジョウバエから得られる酵母株は、その環境特性から一般的な株とは異なる性質を示す場合がある。特に胞子形成能に顕著な違いが見られ、分裂酵母Schizosaccharomyces japonicusは通常、窒素源が枯渇すると胞子を形成するが、ショウジョウバエ由来株の多くは窒素源が豊富な条件でも胞子を形成することが分かった9)。これは捕食圧による適応の可能性があり、胞子は細胞よりもストレス耐性能が高く10)、さらにショウジョウバエの体内でも10倍以上高い生存率を示すことが実験的に確認されている11)。また、クロップ内では多種多様な酵母が混在し、昆虫を媒介した異型交配が起こりやすいことが知られている12)。このため、そこで雑種形成や遺伝子の水平伝播が生じ、多様性が促進されている可能性がある。つまり、このライブラリには通常とは異なる性質を持った酵母株が含まれており、宿主としても遺伝子資源としても高い価値を持つと考えられる。
3.表現型解析と利活用の可能性
3-1 バイオインフォマティクスを活用した野生酵母株の表現型空間の探索
良い酵母とは何だろうか。美味しい食品を作ったり、付加価値の高い生産物を作ったりする酵母がそれにあたるだろうか。では、そのような性質を知るためには、まず酵母が持つポテンシャルの幅を把握する必要がある。酵母は、生理学的試験が種の同定に重要であることから、炭素源や窒素源の資化能や発酵能に関する情報は比較的豊富である。2011年出版のThe Yeasts (第5版) には約1,300種の酵母のこうした情報が記載されているが、情報は散在的で使いやすくはなく、例えば生育可能温度の情報は多く記載されている一方で、pH耐性や酸・塩存在下での生育可否といったストレス耐性に関する情報はほとんどない。
そこで私たちは、単離した約220種の酵母について、こうした表現型を体系的に調査し、その潜在能力を評価することを目指した。本ライブラリには未記載種も多く含まれるため、炭素源34種類、窒素源5種類の資化能に加え、温度・pH変動に対する生育可否、酸・塩・浸透圧・アルコール耐性、通気条件の違いなどについてデータを取得し、遺伝的系統と合わせて比較した。また、合成培地による回分培養により、増殖速度やエタノール発酵能も評価した。これらの表現型データは、いくつかの興味深い知見を与えてくれる。例えば、発酵能を有する酵母は子嚢菌門に集中していたり、低pH (pH1.5程度)で生育可能な種は特定の属に偏っていたり、高いエタノール耐性を示す種はエタノール生産能も高いなど、直感的に理解しやすい相関が見られた。一方で、両立しにくい表現型の組み合わせも明らかになった。詳細は現在執筆中の科学論文で報告予定であるが、炭素源や窒素源利用の広さと、生育可能なある特定の環境条件の間には、酵母種全体を通じて広い関係性が存在する。なお、Saccharomycotina亜門に属する広範な酵母株を対象に、こうした炭素源スペクトルの幅と表現型の関係性を解析した研究は報告されているが13)、本研究のように幅広い遺伝系統間でストレス耐性との関係性を明らかにした研究はほとんどない。こうした発見は、酵母の単離環境や生育条件の詳細な記録と表現型の定量的記述、既知情報の解析を組み合わせることで初めて得られるものであり、体系的な俯瞰が酵母理解の深化につながる。有用な酵母を見つけ、あるいは構築するためには、このような情報基盤を整備し、理解することが不可欠である。
3-2 野生酵母株の利活用の可能性
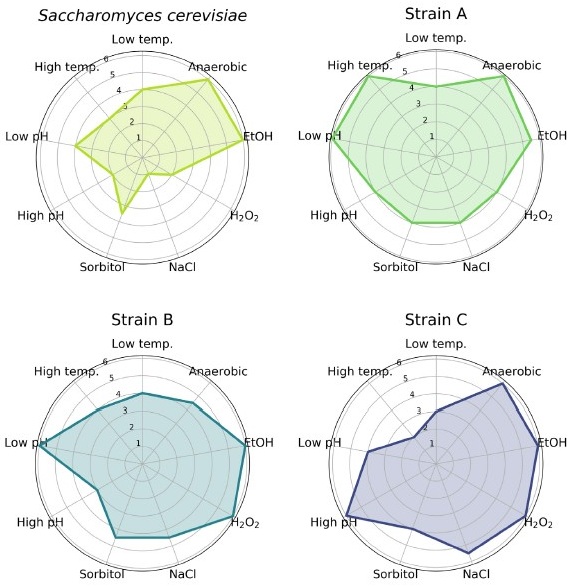
ストレス耐性能をスコア化すると、酵母の潜在能力が可視化される。これは今回調査した表現型に基づく評価であることに留意する必要があるが、レーダーチャートで比較すると、モデル酵母Saccharomyces cerevisiaeよりも高い能力を示す株が存在する (図3)。例えば、酵母株A, Bは沖縄県のショウジョウバエから単離された種で、複数の過酷環境で満遍なく高い性能を示す。酵母株Cは宮崎県の植物から単離されたCandidozyma属の新種と考えられ、同様に能力が高い。他にも潜在能力の高い酵母種は多数見つかっており、これらは次世代を担う有用な酵母宿主となる可能性がある。また、特定の表現型が突出して優れている株も存在する。3M NaCl下で生育可能な種や、30 mM H2O2存在下でも問題なく増殖する種は、特定の産業プロセスで利用価値が高い。

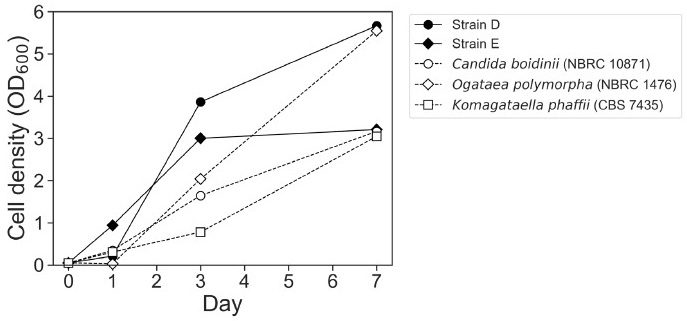
さらに、酵母が最も利用しにくい炭素源の一つはメタノールであり、単一炭素源として利用できる種は全体の数%にすぎない。Candida boidiniiのような古典的メタノール酵母は、葉面から単離されることが多いと言われている14,15)。一方、私たちのライブラリにもメタノール資化性酵母が含まれている。図4に示すように、青森県のタンポポから単離された酵母株Dは、既存酵母3株に比べメタノール培地での増殖が良かった。また、熊本県のショウジョウバエから単離された酵母株Eもメタノール資化能を示した。メタノールはCO2から化学合成可能な資源であるため、これを資化できる酵母は温室効果ガス削減にも寄与し得る。こうした酵母は特定の代謝経路や酵素を保有している可能性があり、現在ゲノム解析や表現型解析を進めている。また、酵母の機能的特徴を簡便に検索・比較できる情報基盤ツールの構築も進めており、このツールにより研究者は目的に応じた酵母の単離戦略をより的確に立案できるようになる。これにより、特定機能を有する酵母の発掘や利活用が飛躍的に加速すると期待される。酵素工学研究会会員の中で興味のある方は、ぜひご連絡いただきたい。

4.おわりに
「有用な酵母とは何か」という問いの答えは、人によって異なる。「優れた人とはどういう人か」という問いに答えが一つでないように、増殖が早いことを良しとする人もいれば、遺伝子操作の容易さを重視する人もいる。逆に言えば、求める性質に応じて適切な酵母を提供するために、野生酵母カタログは大きな役割を果たすだろう。また、酵母の潜在能力を知ることは、その限界を知ることにもつながる。酵母が適している条件や目的が明らかになれば、有用な形質を持つ株同士を交配したり、特定の遺伝子や酵素を異所的に導入したり、などの手段も取り得る。しかし、表現型が両立しにくい組み合わせがあることも確かであり、こうした基礎データは育種デザインにおいて重要な判断材料となる。野生酵母の多様性を知り、その性質を体系的に整理することは、基礎研究と産業利用の双方において新たな可能性を切り拓く。本稿が、皆さまの研究やものづくりの現場において、新しい酵母との出会いのきっかけとなれば幸いである。
謝辞
本稿で紹介した成果は、科学研究費補助金 (21K05342)、JST戦略的創造研究推進事業 (ACT-X:JPMJAX21B9、GteX:JPMJGX23B4、さきがけ:JPMJPR24N9)、発酵研究所一般助成 (G-2023-1-026) の支援を受けて実施した。当研究室の支援研究員・山本真理さん、修士2年・井手円佳さんには、野生酵母株の単離や情報解析に多大な協力をいただいた。また、これまでの約5年間で野生酵母の採集・単離に協力してくださった方は50名以上にのぼる。お一人ずつのお名前をここに記すことは誌面の都合上難しいため、当研究室のホームページ (https://tbtsjlpmghrs.wixsite.com/saypombe) に記載しているので、ぜひご参照いただきたい。
文献
1) 高島昌子: 生物と化学, 57, 669 (2019).
2) Baumberger, J. P.: Proc. Natl. Acad. Sci. USA, 3, 122 (1917).
3) Broderick, N. A., Lemaitre, B.: Gut. Microbes., 3, 307 (2012).
4) Nayduch, D., Burrus, R. G.: Ann. Entomol. Soc. Am., 110, 6 (2017).
5) Kurtzman, C. P., Robnett, C. J.: Antonie. van. Leeuwenhoek., 73, 331 (1998).
6) Seike, T., Takekata, H., Uchida, Y., Sakata, N., Furusawa, C., Matsuda, F.: Int. J. Syst. Evol. Microbiol., 73, 005739 (2023).
7) Seike, T., Takekata, H., Sakata, N., Furusawa, C., Matsuda, F.: Int. J. Syst. Evol. Microbiol., 74, 006537 (2024).
8) Seike, T., Takekata, H., Kono, K., Sakata, N., Kotani, H., Furusawa, C., Matsuda, F.: Int. J. Syst. Evol. Microbiol., 75, 006661 (2025).
9) Seike, T., Sakata, N., Matsuda, F., Furusawa, C.: J. Fungi., 7, 302 (2021).
10) Coluccio, A., Bogengruber, E., Conrad, M. N., Dresser, M. E., Briza, P., Neiman, A. M.: Eukaryot. Cell., 3, 1464 (2004).
11) Coluccio, A. E., Rodriguez, R. K., Kernan, M. J., Neiman, A. M.: Plos. One., 3, e2873 (2008).
12) Reuter, M., Bell, G., Greig, D.: Curr. Biol., 17, R81 (2007).
13) Opulente, D. A., LaBella, A. L., Harrison, M.-C., Wolters, J. F., Liu, C., Li, Y., Kominek, J., Steenwyk, J. L., Stoneman, H. R., VanDenAvond, J., Miller, C. R., Langdon, Q. K., Silva, M., Gonçalves, C., Ubbelohde, E. J., Li, Y., Buh, K. V., Jarzyna, M., Haase, M. A. B., Rosa, C. A., Čadež, N., Libkind, D., DeVirgilio, J. H., Hulfachor, A. B., Kurtzman, C. P., Sampaio, J. P., Gonçalves, P., Zhou, X., Shen, X.-X., Groenewald, M., Rokas, A., Hittinger, C. T.: Science., 384, eadj4503
(2024).
14) Nakase, T., Imanishi, Y., Ninomiya, S., Takashima, M.: J. Gen. Appl. Microbiol., 56, 169 (2010).
15) Santos, A. R. O., Faria, E. S., Lachance, M. A., Rosa, C. A.: Int. J. Syst. Evol. Microbiol., 65, 1855 (2015).