【トピックス】
大腸菌におけるタンパク質生産改善のための技術開発
加藤晃代
名大院・生命農
1.はじめに
タンパク質の機能解析や開発において、「タンパク質を作る」という工程は必要不可欠である。1970年代に遺伝子組換えによる異種タンパク質生産が報告されて半世紀が経過した今、様々な宿主ベクター系が開発され、核酸合成技術も進歩し、我々はほぼどのようなタンパク質であっても任意の宿主で生産を試みることが可能となってきた。しかしながら、それらが実際に活性のある可溶性タンパク質として問題なく生産されるか否かは全くの別問題であり、強力な転写プロモーターと宿主に最適化されたコドンを用いても生産されない場合がある。本稿では、そのようななぜか生産できないタンパク質を、主に大腸菌発現系で簡単に生産可能とするための筆者らの取り組みを紹介する。
2.タンパク質の生産
2-1 大腸菌によるタンパク質生産
酵素、抗体、輸送体および分化因子などのタンパク質は、生体を構成する重要な分子であると同時に、生命科学研究およびバイオ産業を広く支えている。したがって、任意のタンパク質を低コストで自由に作る、という技術の重要性はますます高まってきている。
タンパク質は、DNAからmRNAへの転写、そしてリボソームがmRNA情報に従いアミノ酸一つ一つを連結させていく翻訳という生物に普遍的なシステムを利用して合成される。その中でも、大腸菌はその扱いやすさや利用できるツールの豊富さ故に、バイオ系研究室における異種タンパク質発現の第一選択肢である。利用できる転写プロモーターとしてIPTGやラクトースで誘導可能なT7、lac、tac、trc系、アラビノース誘導のaraBAD系、低温誘導可能なcspAなどがあり、宿主株も多く開発されている (表1)。
表1 タンパク質発現に利用可能な大腸菌宿主の例

大腸菌の細胞質内発現においては、thioredoxin reductaseやglutathione reductaseにより還元環境であるため、ジスルフィド結合を有するタンパク質の形成が困難であるが、表1に示すように、さまざまな改良株が開発されており、ジスルフィド結合の形成が必要な抗体をはじめとする従来生産困難であったタンパク質細胞質内生産も可能となってきている。また、in vitroで転写翻訳反応を完結できる無細胞タンパク質合成系においては、宿主に対し毒性を示すようなタンパク質であっても、わずか90分程度の反応で合成可能であり、ハイスループットスクリーニング系の構築や翻訳研究に欠かせないツールとなっている。
2-2 タンパク質生産における課題
昨今は、発現宿主だけでなく、コドン最適化や人工遺伝子合成などの技術も発達し、異種タンパク質を生産するための技術的ハードルは下がってきている。生産させたいタンパク質のアミノ酸配列情報さえ分かっていれば、コドン最適化された人工遺伝子として購入することができるため、自然界に由来するゲノムやcDNAなどに依存したDNA増幅とクローニング作業を行うことなく、異種発現を試みることができる。
では、コドン最適化された遺伝子と強力なプロモーターを用いれば必ず目的タンパク質を大量に合成できるのだろうか。残念ながら、「必ずしもうまくいくとは限らない」というのが現状の答えである。大腸菌を宿主とした場合、目的タンパク質のフォールディングが正しくされず、不溶性になってしまうことがあるが、その場合は敢えてレアコドンを使用し翻訳を遅くしたり、培養温度を下げたりすることにより、フォールディング効率を改良できることが知られている。また、表1に挙げたような改良宿主を使用することで改善する場合もある。しかしながら、それ以前に、可溶性にも不溶性にも目的タンパク質が殆どあるいは全く生産されない場合は、手の打ちようもなく、培養条件やシャペロンとの共発現、他のタンパク質との融合発現、最終的には宿主―ベクター系の変更など、非常に時間と手間のかかる試行錯誤が必要となる。
2-3 タンパク質難生産の原因
目的タンパク質が生産されない原因として、レアコドンなどのコドンバイアスの他、mRNAの構造的問題や、生産されたタンパク質自体がプロテアーゼによる分解を受けやすいといったことなどが要因として考えられているが、個別事象に対する明確な原因は殆ど明らかになっていない。細胞内のmRNAと対応するタンパク質量がすべて相関するわけではないことも報告されており、転写と翻訳の関係性は単純ではないことが示唆される。
3.ペプチドによる翻訳促進技術の開発
3-1 N末端配列の重要性
筆者らのグループでも、主にウサギやヒト由来のモノクローナル抗体遺伝子を大腸菌無細胞タンパク質合成系で生産させ評価する系を開発する中で、コドン最適化したにも関わらず原因不明で殆ど生産されないことや、同一抗原に対する抗体であってもクローンによっては問題なく生産されること等もあり、何がタンパク質の生産性に影響を与える要因なのかが分からず頭を悩ませていたことがあった。当時、「そもそも哺乳類のタンパク質は大腸菌では生産されにくい」といった通説が存在していたが、哺乳類由来のオリジナルコドンであっても大腸菌発現系で問題なく生産できるものもあり、単に「哺乳類由来だから」や「コドンが適切ではないから」といった理由だけでは片付かない現象であるように思われた。また、プロテアーゼやRNaseなどの混入が殆どない再構成系無細胞タンパク質合成系でも同様の現象が認められたことから、mRNAやタンパク質の分解の問題でもないように思われた。
一方、タンパク質の生産性を支配する要因が何かを解明するために、ディープシーケンス技術やプロテオミクス技術を用いたアプローチが行われており、例えば、Boël らは、大腸菌の6,348のタンパク質発現データ解析結果から、使用コドンがタンパク質濃度およびmRNA濃度と強く相関することや、N末端から16アミノ酸残基分の塩基配列がmRNAのフォールディング構造に大きく影響することを報告していた4)。また、Goodman らは、大腸菌において開始コドン付近にレアコドンが存在するとmRNA構造が緩み、タンパク質生産量を増加させるという興味深い傾向を報告していた5)。Bivona らは大腸菌によりIgαの異種生産を試みる中で、開始コドンに対応するメチオニンに続く2番目のアミノ酸の種類がコドンよりも生産性に影響を与えることを示しており、大腸菌細胞質内でもともと存在量の多いタンパク質はN末端から数アミノ酸の出現頻度に偏りがあることを報告していた6,7)。これらのことから、目的タンパク質の生産のされやすさは、塩基配列だけでなくアミノ酸にも左右される可能性や、特にN末端側の配列に影響される可能性が示唆される。
3-2 翻訳を促進するSKIKペプチドの開発
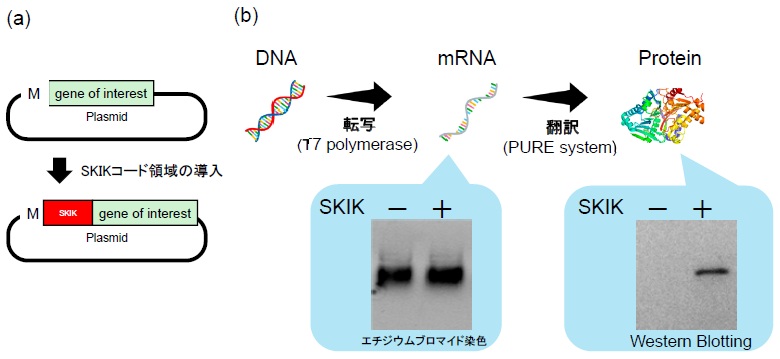
そこで、そのような報告からヒントを得て、大腸菌系で生産困難であったタンパク質のN末端に、元来生産性の高いタンパク質において出現頻度の高いアミノ酸で構成される数残基のペプチドを導入してはどうかと着想した。ペプチド配列の種類として幾つかの候補が考えられたが、Ser-Lys-Ile-Lys (SKIK) という、最初に検討した4アミノ酸残基に対応する12塩基をN末端に導入するというシンプルな方法により、目的タンパク質 をコードする遺伝子の内部コドンを変更することなく、その生産性を数十倍にも増大できることを見いだした8)。面白いことに、このSKIKをN末端に持つタンパク質は大腸菌ではコールドショックタンパク質の一種類のみであった。SKIKペプチド付加による目的タンパク質の生産量増大効果は、大腸菌無細胞タンパク質合成系、大腸菌生細胞系および真核微生物である出芽酵母発現系において確認された。また、抗体遺伝子のみならず、コドン最適化してもなぜか生産できなかったような様々なタンパク質に対しても、百発百中ではないものの、生産量増大効果を示すことが確認されている9)。
では、なぜこのような短いペプチドを付加するのみでタンパク質生産量が増大するのだろうか。これまでに、SKIKペプチドをコードする塩基配列は何でも良いこと、SKIK配列の有無でmRNAの予測構造に大きな違いは見受けられないこと、ペプチド導入により転写ではなく翻訳が促進されることが明らかとなっている (図1)。

3-3 翻訳を停滞させるアレストペプチド配列に対する効果
近年、翻訳工程で合成されたばかりの新生ペプチド (新生鎖とも呼ばれる) の中には、翻訳を停滞させるアレストペプチドと呼ばれるものが存在することが分かってきている10)。アレストペプチドは2000年代以降初めて報告されてから、生物種を超えて見つかってきており、翻訳停滞は、塩基配列ではなくアレストペプチドを構成するアミノ酸に起因することや、リボソームと相互作用することにより翻訳の一時停止を引き起こすこと等が明らかになっている。大腸菌においては、SecM の一部 (FSTPVWISQAQGIRAGP) や、人工的に作られたFQKYGIWPPPという連続したプロリン残基を有する配列が非誘導型のアレストペプチドとして知られている。
そこで、筆者らは、翻訳促進能が確認されたSKIKペプチドをアレストペプチドと組み合わせて何かが起きれば、SKIKの翻訳促進現象のメカニズム解明に近づけるかもしれないと考えた。そして、SKIKとアレストペプチドを様々な組み合わせで融合し無細胞タンパク質合成系により翻訳反応を行うと、非常に驚くべきことに、SecMのアレストペプチドのすぐ上流にSKIKペプチド配列を付加すると、翻訳停滞が見かけ上相殺されて、結果的にタンパク質生産量が増大するという現象が観測された。また、SKIKの導入箇所がN末端ではない場合でも相殺効果が観測された11)。さらに、翻訳のペプチジル転移反応が妨げられ翻訳を停滞させることで知られる連続したプロリンペプチド配列を有するアレストペプチドを用いた検証においては、翻訳停滞を効果的に相殺するためには、SKIKペプチドとアレストペプチドの距離関係が重要であり、距離が近いほど相殺効果が高くなるということも明らかとなった (図2)12)。この現象は、無細胞翻訳系で顕著であったが、生細胞のBL21 (DE3) による発現系を用いた場合も同様の傾向が認められた。
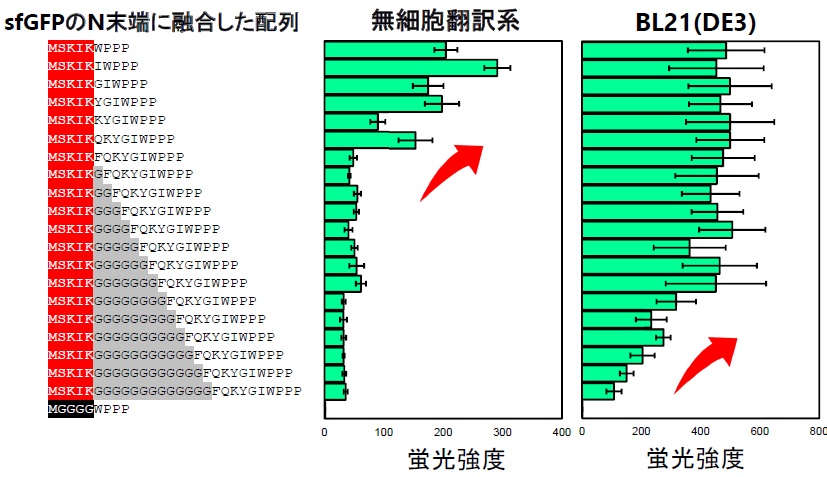
図2 連続プロリンを有し翻訳停滞を引き起こすアレストペプチドに対するSKIKの効果と距離の影響
上述した通り、アレストペプチドによる翻訳停滞は塩基配列ではなくペプチドの配列が原因である。また、ここには示していないが、無細胞翻訳系内に化学合成したSKIKペプチドを追添加しても翻訳反応は何の影響も受けなかった。以上を統合すると、翻訳で生じたSKIKという新生ペプチドが翻訳促進を引き起こしており、その翻訳促進の結果として、アレストペプチドによる翻訳停滞が見かけ上相殺されたり、生産性の低いタンパク質の生産量が増大したりするような現象が観測されているものと推測される。わずか数アミノ酸のペプチド配列が翻訳動態に大きな影響を与えるという事実は科学的に非常に興味深いものであり、現在は、その分子メカニズム解明に向けて研究を進めている。
4.翻訳促進ペプチドの開発と利用
4-1 生産困難タンパク質への利用
ペプチドによる翻訳動態の変化は科学的に興味深い一方で、タンパク質生産にも応用できる重要な知見であると考えている。これまでに、SKIKという配列が翻訳促進能を発揮することや、下流のアレストペプチドの効果を相殺し得ることを明らかとしたが、この知見を活用することにより、より効果的にタンパク質の生産量を増大可能と考えられる。例えば、図3は、筆者らがSKIKの導入効果を検討した中でも、特徴的なアミノ酸配列を有するタンパク質の一例である。これは243残基をコードする好塩性細菌Halomonas elongata由来のepsF遺伝子であり、C末端側にGWPPPというアレスト様配列を含み、さらに、難翻訳であるとされる連続アルギニン配列も有していた。無細胞タンパク質合成系での評価の結果、SKIKを付加していないオリジナル配列と比較し、N末端にSKIKを付加したものでは生産量の増加が確認された。このように、配列情報を確認しながら翻訳促進配列導入の是非や位置を検討することが有効な手段となり得るであろう。ただし、現状では翻訳停滞を決定付ける配列的特徴の全貌は明らかになっておらず、後述する翻訳促進配列と同様に停滞配列の情報整備も重要であると考えられる。


4-2 SKIK以外の配列の開発
筆者らは、SKIKのようなペプチドを翻訳促進ペプチド (Translation-Enhancing Peptide: TEP) と称し、現在は様々な強度で翻訳促進能を発揮しうるTEPの探索と同定を進めている。これまでに、大腸菌を中心として複数のTEPを見いだしており、機械学習により、4アミノ酸から構成される全160,000パターンのペプチドの翻訳促進強度を予測する技術を開発した13)。では、多数のTEPデータを用いて何ができるであろうか。ここでは、N末端だけでなく、遺伝子内部への導入であってもTEPが翻訳促進能を発揮可能である点が重要になる。SKIKの開発以来、タンパク質生産量増大のためにはN末端にSKIKを付加することが多かったが、それのみでは効果が不十分あるいはあまりない場合もあった。一方、最近は、内部にTEPを導入するとN末端単独のときよりも生産量をさらに増大できることがわかってきた。実際に、放線菌由来の酵素遺伝子に関し、N末端のSKIKに加え内部への新規TEP導入により、可溶性の活性型酵素生産量を15倍以上にも増大できることが示された (未発表データ)。内部へのTEP導入で注意すべき点は、タンパク質本来の構造や活性に影響を与えない導入箇所を選定することである。そのためには、数アミノ酸のTEP配列を挿入するのではなく、オリジナルタンパク質配列内の1あるいは数アミノ酸を置換しTEP配列に変更する戦略が有効であると考えられる。これにより、欠失・挿入等を伴わず翻訳効率を変更することが可能になるであろう。
5.おわりに
本稿では、筆者らが開発してきた翻訳促進ペプチドSKIKと大腸菌系での利用について中心に紹介した。今後、AIを活用した構造予測や機能予測が発展し、生物の転写翻訳システムを利用して、「実際に作って調べる」ための技術はますます重要になると考えられる。タンパク質を構成する短いペプチド配列が翻訳を正にも負にも制御しうるという興味深い知見を基に、目的タンパク質の生産性を簡単かつ高い自由度で改善できる技術開発を進め、バイオ研究や産業の発展に少しでも役立てていきたいと考えている。
謝辞
本研究はJST FOREST Program (grant No. JPMJFR2204)、科研費23K04989、GteXProgram Japan Grant Number JPMJGX23B6 およびJPMJGX23B4の支援を受けて実施したものである。本研究に携わっていただいた共同研究者の皆様、学生の皆様に感謝申し上げる。
文献
1) Studier, F. W., Moffatt, B. A.: J. Mol. Biol., 189, 113 (1986).
2) Hatahet, F., Nguyen, V. D., Salo, K. E. H., Ruddock, L. W.: Microb. Cell. Fact., 9, 67 (2010).
3) Meyer, A. D., Segall-Shapiro, R. H., Glassey, E., Zhang, J., Voigt, C. A.: Nat. Chem. Biol., 15, 196 (2019).
4) Boël, G., Letso, R., Neely, H., Price, W. N., Wong, K. H., Su, M., Luff, J., Valecha, M., Everett, J. K., Acton, T. B.,
Xiao, R., Montelione, G. T., Aalberts, D. P., Hunt, J. F.: Nature, 529, 358 (2016).
5) Goodman, D. B., Church, G. M., Kosuri, S.: Science, 342, 475 (2013).
6) Bivona, L., Zou, Z., Stutzman, N., Sun, P. D.: Protein Expr. Purif., 74, 248 (2010).
7) Ishihama, Y., Schmidt, T., Rappsilber, J., Mann, M., Hartl, F. U., Kerner, M. J., Frishman, D.: BMC Genomics, 9, 102 (2008).
8) Ojima-Kato, T., Nagai, S., Nakano, H.: J. Biosci.Bioeng., 123, 540 (2017).
9) Ojima-Kato, T.: Biosci. Biotechnol. Biochem., 89, 1 (2024).
10) Ito, K., Chiba, S.: Annu. Rev. Biochem., 82, 171 (2013).
11) Ojima-Kato, T., Nishikawa, Y., Furukawa, Y., Kojima, T., Nakano, H.: J. Biol. Chem., 299, 104676 (2023).
12) Nishikawa, Y., Fujikawa, R., Nakano, H., Kanamori, T., Ojima-Kato, T.: ACS Synth. Biol., 13, 3908 (2024).
13) Ojima-Kato, T., Yokoyama, G., Nakano, H., Hamada, M., Motono, C.: bioRxiv, https://doi.org/10.1101/2025.07.17.665026 (2025).