【トピックス】
グリコール酸発酵生産に資する酢酸菌由来グリオキシル酸還元酵素の基質・補酵素特異性
井上真男、三原久明
立命大・R-GIRO、立命大・生命
1.はじめに
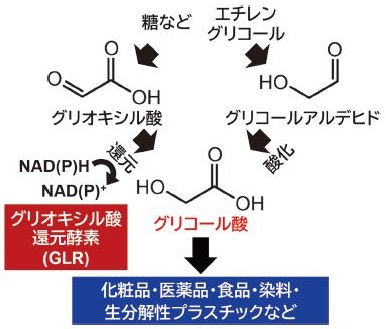
グリコール酸は、ヒドロキシ基とカルボキシ基の両方を持つ最もシンプルな化合物である (図1)。グリコール酸の需要は近年著しく増加しており、その世界市場価値は2030年までに800億円を超えると予想されている1)。特に、化粧品や医薬品、食品、染料、生分解性プラスチックなどの原料になるため、微生物を用いたその発酵生産に大きな期待が集まっている2,3)。グリコール酸の発酵生産は主に、(1) 糖などを出発物質として生じる代謝中間体グリオキシル酸の還元を介する経路1,4)、または (2) エチレングリコールを出発物質としてグリコールアルデヒドの酸化を介する経路によって行われる5,6)。(1) については、大腸菌や酵母などを用いた研究が行われているが、生産量が10-20 g/Lと低い1,4)。一方、(2) については酢酸菌Gluconobacter oxydans 621H株を用いることで、220 g/Lの生産量が達成されている5,6)。
図1 グリコール酸発酵生産とグリオキシル酸還元酵素
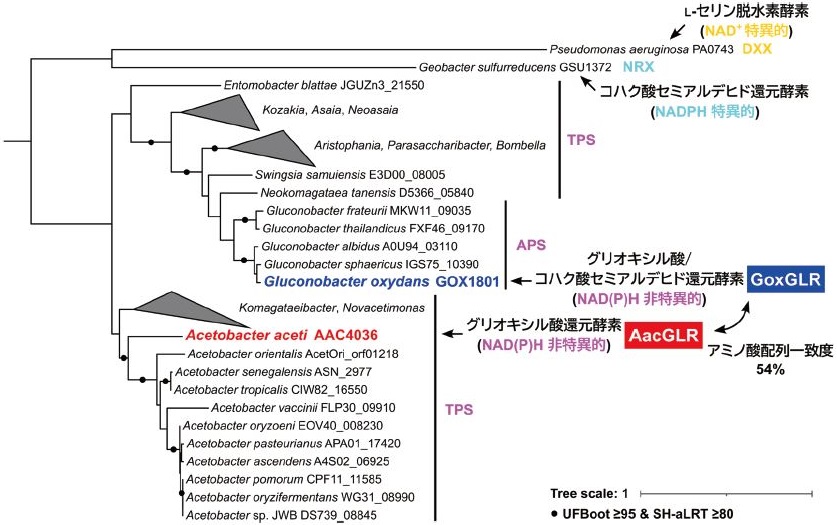
グリオキシル酸還元酵素 (GLR) は (1) の還元的経路の鍵酵素であるほか、グリコール酸の酸化も触媒することから、(2) の酸化的経路における生産能低下の原因となりうる (図1)。我々は、酢酸菌Acetobacter aceti JCM 20276株とGluconobacter oxydans 621H株に由来するGLR (それぞれaac4036とgox1801の遺伝子産物であり、以下AacGLRとGoxGLRと略す) の基質特異性と補酵素特異性に着目した研究を進めており、特に、AacGLRがGoxGLRに比べてグリオキシル酸への基質特異性が極めて高いこと、両者ともにNAD(P)への特異性が低いことを明らかにしている7-9)(図2)。このことは、副反応がなく、細胞内のNAD(P)H量に影響されにくいグリコール酸発酵プロセスを可能にし、酵素・代謝工学を通じてグリコール酸生産能の向上に資すると考えている。本稿では、グリコール酸発酵生産の鍵となる酢酸菌株由来のGLRに着目し、その基質・補酵素特異性、およびその構造基盤に関する最新の知見を紹介したい。

2.酢酸菌由来GLRのユニークな基質・補酵素特異性
2-1 酢酸菌GLRホモログ間の基質特異性の違い
GLRはβ-ヒドロキシ酸脱水素酵素スーパーファミリー (β-HADs) という極めて大きなタンパク質スーパーファミリーに属している10-14)。β-HADsには、GLRだけでなく6-ホスホグルコン酸脱水素酵素やタルトロン酸セミアルデヒド還元酵素、L-セリン脱水素酵素なども属するほか、触媒化学の分野で重要なイミン還元酵素と構造類似性を示すなど、世界的に注目を集めている。バクテリア、アーキア、真核生物を問わず多くの生物において複数のβ-HADsが存在することを鑑みても、このような基質の多様性は、生物ごとの代謝との共進化の結果と解釈することができる。
酢酸菌科のゲノムにコードされるGLRは、β-HADsにおいて独立した系統群を形成しており、酢酸菌特有の代謝機能を有していると予想された (図2)。GoxGLRは2015年に好気性細菌由来の初のコハク酸セミアルデヒド還元酵素として同定されていたが、グリオキシル酸に対する基質特異性については調べられていなかった15)。そこで、GoxGLRならびに、我々が独自にゲノム配列の決定に成功したA. aceti JCM 20276株16)由来のAacGLRを用いて、それらのグリオキシル酸還元活性について調べることとした7,8)。ちなみに両者のアミノ酸配列一致度は54%であることを先に述べておく。
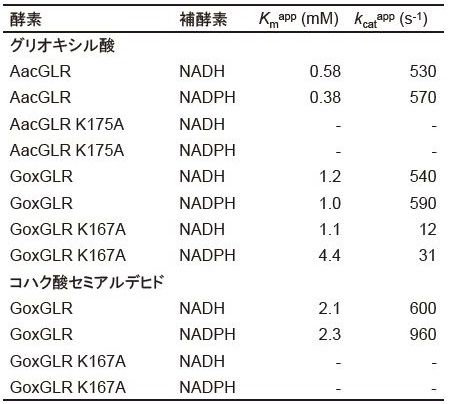
AacGLRとGoxGLRは、大腸菌BL21(DE3)を宿主としてpColdIIベクターを用いてIPTGと低温による発現誘導を行い、Hisタグ付き組換えタンパク質として発現させ、金属アフィニティー樹脂を用いて精製したものを用いた7,8)。AacGLRはいずれの温度・pHでもコハク酸セミアルデヒドに対する還元活性を示さず、グリオキシル酸に対してのみ還元活性を示した7)。最適温度は45℃であり、最適pHは4.0 (100 mM クエン酸緩衝液) であった。0.15 mMのNADHまたはNADPH存在下におけるグリオキシル酸に対するみかけのKm値はそれぞれ0.58 mMと0.38 mMであり、みかけのkcat値は530 s-1と570 s-1であった。これはNAD(P)Hへの特異性が低いことを示している (表1)。
表1 各酵素活性の反応動力学定数
一方で、GoxGLRはグリオキシル酸およびコハク酸セミアルデヒドの両方に対して高い還元活性を示した8)。どちらの基質に対しても最適温度は30℃であり、最適pHは6.5 (40 mM リン酸緩衝液) であった。pH 4.0とpH 6.5の両方の条件でAacGLRとGoxGLRの活性測定を行ったが、どちらのpHにおいてもGoxGLRはグリオキシル酸とコハク酸セミアルデヒドの両方に還元活性を示したが、AacGLRはグリオキシル酸にのみ還元活性を示した。このことは、その高い配列相同性にもかかわらず、GoxGLRとAacGLRでは基質特異性が異なることを明確に示している。さらに、0.15 mMのNADHまたはNADPH存在下におけるGoxGLRのグリオキシル酸に対するみかけのKm値はそれぞれ1.2 mMと1.0 mMであり、みかけのkcat値は540 s-1と590 s-1であった (表1)。一方で、コハク酸セミアルデヒドに対するみかけのKm値はそれぞれ2.1 mMと2.3 mMであり、みかけのkcat値は600 s-1と960 s-1であった。以上の結果、GoxGLRはグリオキシル酸とコハク酸セミアルデヒドの両方に対して同程度の高い還元活性を示すことが明らかとなり、AacGLRと同様にNAD(P)Hへの特異性が低いことを示している。
2-2 推定触媒残基の変異体における基質特異性の変化
AacGLRとGoxGLRは保存されたLys残基 (それぞれLys175とLys167) を有している。このLysは他のβ-HADsにおいて推定触媒残基として報告されており11,12)、GLRにおいても触媒活性に重要なアミノ酸残基である可能性があった。そこで、部位特異的変異導入によってAacGLR K175AとGoxGLR K167Aを作製し、それらの活性を調べた8)。
その結果、AacGLR K175Aはグリオキシル酸還元活性を完全に消失し、野生型酵素の活性測定に用いた100倍以上の酵素量を添加しても活性はほとんど見られなかった (表1)。このことは、AacGLRの触媒反応においてLys175が必須であることを示唆した。GoxGLR K167Aについてもコハク酸セミアルデヒド還元活性は検出されなかったが、グリオキシル酸還元活性は消失しなかった。GoxGLR K167Aのグリオキシル酸に対するみかけのKm値にはあまり大きな変化はなかったが、みかけのkcat値については20-45倍程度低下していた。GoxGLR K167Aで見られたユニークなこの挙動は、このLysを酸塩基触媒残基とするAacGLR K175Aや他のβ-HAD酵素における観察結果とは大きく異なっている。GoxGLR K167Aとグリオキシル酸との反応では、コハク酸セミアルデヒドとの反応とは異なり、別のアミノ酸残基や水が酸塩基触媒として機能する可能性がある。
以上の結果から、AacGLRとGoxGLRは両者ともに、NAD(P)に対する特異性が低い新たなタイプのグリオキシル酸還元酵素であることが分かった。さらに、AacGLRは厳密な基質特異性を有し触媒活性にLys残基を必須とすること、GoxGLRはAacGLRに比べてより広い基質特異性を示し、Lys残基への変異の効果が基質によって異なることが明らかになった。これらの発見は、酢酸菌株におけるGLRの生理機能だけでなく、β-HADsの構造-機能相関や分子進化においても新たな洞察を与えることが期待できる。
3.酢酸菌由来GLRが示す「ゆるい」補酵素特異性の構造基盤
3-1 AacGLR-NAD(P)H二者複合体のX線結晶構造
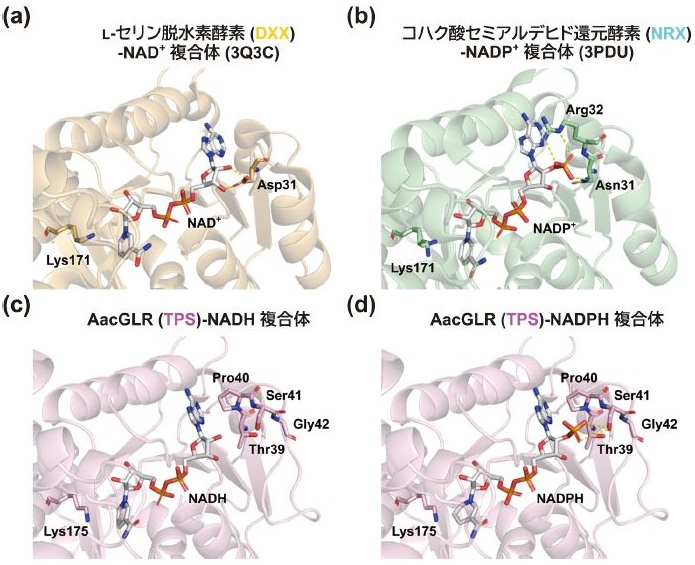
上述した酢酸菌株由来GLRのNAD(P)に対する特異性の低さはβ-HADsの中でも比較的珍しいものである。β-HADsのほとんどはNADかNADPのどちらかに対する比較的厳密な補酵素特異性を示す10-13)。この特異性は3つのアミノ酸からなる補酵素識別配列モチーフによって決定され、NAD依存型はDXXモチーフを、NADP依存型はNRXモチーフを持つ (図2)。DXXモチーフのAspの側鎖カルボキシ基はNADの2′-および3′-ヒドロキシ基と水素結合を可能にし、NADPの2′-リン酸基と静電的に反発することで、NADへの特異性を示す12)(図3a)。一方、NRXモチーフのAsnとArg残基はNADPの2'-リン酸基とそれぞれ水素結合とイオン性の相互作用によって、NADPへの親和性を上げると考えられている13)(図3b)。
図3 従来のβ-HAD酵素による補酵素結合様式と酢酸菌GLRによる新奇結合様式
AacGLRとGoxGLRは、NAD/NADPの識別に用いられる従来のDXX/NRXモチーフの代わりに、それぞれTPSモチーフとAPSモチーフというユニークなアミノ酸配列を持つ (図2)。したがって、この特徴的な配列モチーフがNADとNADPの両方を利用するために必要なのではないか?と考え、その構造基盤を明らかにすることを試みた9)。
AacGLRについて、単独、NADPH複合体、およびNADH複合体のX線結晶構造をそれぞれ1.60 Å、1.68 Å、1.60 Åの分解能で決定した。AacGLRは他のβ-HAD酵素と同様に四量体構造をしており、NAD(P)H結合部位も同様であった。NAD(P)Hのニコチンアミド環と推定触媒残基であるLys175残基との距離は3.6 Å以内であり、反対側には基質結合ポケットらしき空洞が見られたが、基質結合様式については推定することは困難であった。
NAD(P)Hの結合に伴う全体的な構造変化はほとんど見られなかったが (Cα原子のrmsd値は0.15 Å以内)、TPSモチーフ (Thr39-Pro40-Ser41) を含むフレキシブルな長いループ領域 (Arg43-Thr54) の電子密度がNAD(P)H複合体でのみ観察され、モデルを構築することができた (図3c、3d)。これはNAD(P)H結合によってこのループ領域のコンフォメーションが安定化することを示唆した。この長いループ構造は他のβ-HAD酵素には見られないものであり、Thr39-Pro40-Ser41およびGly42がβ-ターン様の構造を形成していることもユニークであった。このβ-ターン様構造はNAD(P)Hのアデニン部分とNADPHの2'-リン酸基を収容し、NAD(P)H結合時にわずかな構造変化を示すことが分かった。さらに注目すべきは、Ser41側鎖のヒドロキシ基がNADPHの2'-リン酸基の2個の酸素原子と相互作用していることであり、このような相互作用も従来のDXX/NRXモチーフを持つ古典的なβ-HAD酵素には見られないものであった。
3-2 「ゆるい」補酵素特異性を決める新戦略
NAD(P)Hの認識におけるTPSモチーフの役割を調べるため、部位特異的変異導入によって、NADPHの2'-リン酸との水素結合を破壊する変異体S41AとS41Dを作製した。さらに、NRXモチーフを模倣したT39N変異体とP40R変異体、およびDXXモチーフを模倣したT39D変異体を作製し、これら4つの変異体についてグリオキシル酸還元活性へのNAD(P)H依存性を調べた9)。
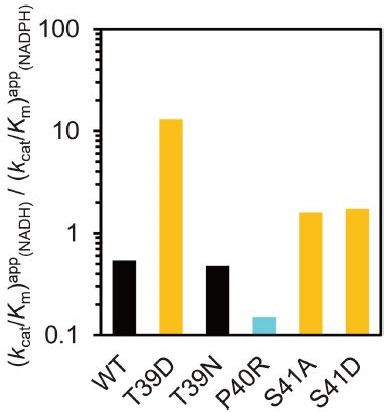
S41AおよびS41D変異体では、NADHに対するみかけのkcat/Km値とNADPHに対するみかけのkcat/Km値の比がそれぞれ1.6および1.7であり、野生型の値である0.54より高く、NADHをより好むように変化したことを示した (図4)。これらの結果は、Ser41とNADPHの2'-リン酸基との間の水素結合がNADPHの適切な収容に寄与している可能性を示唆した。一方、T39NとP40R変異体では、NADHに対するみかけのkcat/Km値とNADPHに対するみかけのkcat/Km値の比がそれぞれ0.48および0.15であり、NADHよりもNADPHに対して高い特異性を示した (図4)。これは、野生型の値よりも1.1倍および3.6倍高い値であり、特にP40R変異体においてArgによって正電荷が導入されたことで、NADPHに対する特異性が高まったと考えられる。しかしながら、これらの変異の効果は限定的であり、他のβ-HAD酵素に見られるほど特異性を上げるには至らなかった。
図4 各AacGLR変異体酵素におけるNADHとNADPHに対する特異性定数の比率
一方で、T39D変異体はNADPHに対して著しい親和性の低下を示し、0.23 mM NADPHの存在下でも初速度は直線的に増大し、Km値の決定はできなかった。NADHに対するみかけのkcat/Km値とNADPHに対するみかけのkcat/Km値の比は13であり (図4)、T39D変異体のNADPHに対するみかけのkcat/Km値は野生型よりも110倍低かったことから、NADPHに対する活性が著しく失われていることが分かった。これらの結果は、β-ターン様構造を形成するThr39の位置に負電荷を帯びたAspを導入したことでAacGLRをNADH依存的酵素に変換したことを示しており、Asp39のカルボキシ基がNADPHの2'-リン酸基との間に静電的な反発を生じたためだと考えることができる。
β-HAD酵素において、このような「ゆるい」NAD(P)H特異性はDRXモチーフを持つ6-ホスホグルコン酸デヒドロゲナーゼにおいて主に研究されてきた17)。この戦略は、NADPの2'-リン酸基とのArgの有利な相互作用、NADPの2'-リン酸基へのAspの反発、NADの2'-ヒドロキシ基との有利な相互作用の間のトレードオフから生じると予想されている。本研究で初めて明らかになったAacGLRのTPSモチーフのように、電荷を持たない補酵素識別モチーフを持つβ-HAD酵素は、GoxGLRを含めて酢酸菌科に高度に保存されている。さらに、AacGLRとたった20%のアミノ酸配列一致度しか示さないβ-HAD酵素である植物病原性真菌Ustilago maydis由来のタルトロン酸セミアルデヒド還元酵素Tsr1は、VYNモチーフを持ち、「ゆるい」NAD(P)H特異性を示すことが知られている18)。したがって、このような電荷を持たない補酵素識別モチーフによる戦略は、多様なβ-HAD酵素で使われていると考えられる。β-HAD酵素のNAD(P)H特異性は4つの戦略 (正電荷、負電荷、正負両電荷、非電荷) それぞれに適した構造基盤により成し遂げられており、それぞれの生物の代謝要求に応じてNAD(P)H特異性を変化させるように適応進化したのであろう。
4.おわりに
本稿では、グリコール酸発酵生産の鍵酵素である酢酸菌株由来GLRのユニークな基質特異性と補酵素特異性の構造基盤について紹介した。GLRはこれまで見過ごされてきた酵素であり、酢酸菌株由来のGLRが代謝工学を通じてグリコール酸発酵生産の鍵酵素になることを期待している。
残された最も大きな謎の1つは、やはり、非常によく似たホモログであるAacGLRとGoxGLRの基質特異性の違いがなぜ生じるのか?であろう。現在、AacGLRだけでなくGoxGLRについてもX線結晶構造解析を進めており、比較的近いうちにその巧みなメカニズムについてご紹介できる日が来ることを願っている。また、なぜ酢酸菌のGLRがこのようにユニークな基質・補酵素特異性を示すのか?についても大きな謎である。酢酸菌におけるGLRの役割は不明であるが、グリオキシル酸経路は酢酸菌の環境応答において重要な働きを担うことが示唆されている19,20)。絶対的に酸素に依存し、かつ呼吸基質が変動する環境で生育する酢酸菌において、GLRは重要な機能を有するに違いないと著者らは考えており、GLRの研究はグリコール酸の発酵生産のみならず、食酢醸造の分野においても大きな可能性を秘めていると言ってよいだろう。
GLRが属するβ-HAD酵素は、いわばやり尽くされた酵素であり、ごく見慣れた山と言えるかもしれない。しかしながら、いざ登ってみると、これまで報告されていたものとは異なる道、そして風景が見えたように思う。微生物の膨大なゲノム情報が溢れる時代を迎え、こと酵素の研究に関しては、どこに新奇性を見出して研究テーマを選択するのか悩ましい時代になってきた。そんな時ふと目の前にある登り尽くされた山に登ってみるのもまた一興なのではないだろうか。
謝辞
本稿の執筆にあたりお世話になりました、立命館大学生命科学部の松村浩由教授、戸部隆太講師 (現東北大学大学院農学研究科准教授)、吉澤拓也講師 (現中外製薬株式会社)、青野陸助教、越智杏奈助教、Toma Rani Majumder氏、Jakkaphan Kumsab氏、京都大学化学研究所の栗原達夫教授、豊橋技術科学大学大学院工学研究科の広瀬侑准教授、大阪工業大学工学部の大森勇門准教授をはじめとする共同研究者の皆様に厚く御礼申し上げます。
文献
1) Senatore, V. G., Milanesi, R., Masotti, F., Maestroni, L., Pagliari, S., Cannavacciuolo, C., Campone, L., Serra, I., Branduardi, P.: FEMS Yeast Res., foae024 (2024).
2) Choi, S. Y., Park, S. J., Kim, W. J., Yang, J. E., Lee, H., Shin, J., Lee, S. Y.: Nat. Biotechnol., 34, 435 (2016).
3) Koerner, J., Horvath, D., Herrmann, V. L., MacKerracher, A., Gander, B., Yagita, H., Rohayem, J., Groettrup, M.: Nat. Commun., 12, 2935 (2021).
4) Zhu, T., Yao, D., Li, D., Xu, H., Jia, S., Bi, C., Cai, J., Zhu, X., Zhang, X.: Biotechnol. Bioeng., 118, 4699 (2021).
5) Hua, X., Zhou, X., Xu, Y.: Bioprocess Biosyst. Eng., 41, 1555 (2018).
6) Yu, Q., Liu, X., Lv, Y., Zhou, X., Xu, B., Wang, H., Xu, Y.: Chem. Eng. J., 452, 138994 (2023).
7) Kumsab, J., Tobe, R., Kurihara, T., Hirose, Y., Omori, T., Mihara, H.: Biosci. Biotechnol. Biochem., 84, 2303 (2020).
8) Majumder, T. R., Inoue, M., Aono, R., Ochi, A., Mihara, H.: Biosci. Biotechnol. Biochem., zbae081 (2024).
9) Majumder, T. R., Yoshizawa, T., Inoue, M., Aono, R., Matsumura, H., Mihara, H.: submitted (2024).
10) Hawes, J. W., Harper, E. T., Crabb, D. W., Harris, R. A.: FEBS Lett., 389, 263 (1996).
11) Njau, R. K., Herndon, C. A., Hawes, J. W.: Chem. Biol. Interact., 130, 785 (2001).
12) Tchigvintsev, A., Singer, A., Brown, G., Flick, R., Evdokimova, E., Tan, K., Gonzalez, C. F., Savchenko, A., Yakunin, A. F.: J. Biol. Chem., 287, 1874 (2012).
13) Zhang, Y., Zheng, Y., Qin, L., Wang, S., Buchko, G. W., Garavito, R. M.: Biochime, 104, 61 (2014).
14) Montgomery, S. L., Pushpanath, A., Heath, R. S., Marshall, J. R., Klemstein, U., Galman, J. L., Woodlock, D., Bisagni, S., Taylor, C. J., Mangas-Sanchez, J., Ramsden, J. I., Dominguez, B., Turner. N. J.: Sci. Adv., 6, eaay9320 (2020).
15) Meyer, M., Schweiger, P., Deppenmeier, U.: Appl. Microbiol. Biotechnol., 99, 3929 (2015).
16) Hirose, Y., Kumsab, J., Tobe, R., Mihara, H.: Microbiol. Resour. Announc., 9, e00962-20 (2020).
17) Maturana, P., Tobar-Calfucoy, E., Fuentealba, M., Roversi, P., Garratt, R., Cabrera, R.: Wellcome Open Res., 6, 48 (2021).
18) Liu, Y., Koh, C. M., Sun, L., Ji, L.: PLOS ONE, 6, e16438 (2011).
19) Sakurai, K., Yamazaki, S., Ishii, M., Igarashi, Y., Arai, H.: J. Biosci. Bioeng., 115, 32 (2013).
20) Ahn, S., Jung, J., Jang, I. A., Madsen, E. L., Park, W.: J. Biol. Chem., 291, 11928 (2017).