【トピックス】
生化学研究に資するメタボロミクスの現状と課題
津川裕司、轟 善勝、西田孝三
東京農工大学工学研究院
1.はじめに
メタボロミクス研究は、生体内代謝物の総体 (メタボローム) を捉えることで、生命現象の理解や疾患の事前診断や予後予測マーカー開発に貢献してきた。本稿では、メタボロミクスという言葉が使われ始めて25年ほどの月日が流れた現在において、メタボロミクスの技術が生化学研究にどのように利活用されうるのかについて述べる。そして、メタボロミクスで可能なところと、まだ研究段階であることを明確にすることで、読者の生化学研究にメタボロミクスを取り入れるきっかけになれば幸いである。
2.メタボロミクスの歴史と現在
メタボロミクスという言葉が初めて使われたのは1998年のOliver S.G. et al.の論文だと言われている1)。メタボロミクスの研究は、細胞内に含まれる代謝物の多様性をいかにして網羅的に検出・同定するかが最初の課題となった。代謝物の網羅的検出には、ガスクロマトグラフィー (GC) や液体クロマトグラフィー (LC) などにより生体試料に含まれる化合物を分離し、それらを質量分析 (MS) で検出する手法が広く利用されている。現在では、メタボローム解析において「ゴールドスタンダード」と呼ばれるプロトコールがいくつか提唱されており、最近では「分析化学」の新手法が考案される論文は減少傾向にある。具体的には、香気成分の分析や短鎖脂肪酸を含む低沸点代謝物の分析はガスクロマトグラフィー質量分析 (GC-MS)、イオン性の代謝物はキャピラリー電気泳動質量分析 (CE-MS)、そして脂質分子を含む幅広い代謝物の分析は液体クロマトグラフィー質量分析 (LC-MS) が用いられている2)。一方近年では、機械学習を含むメタボロミクスデータサイエンスに関する研究論文数が急上昇しており、新規化学構造や代謝経路の発見、およびメタボローム解析を切口とした研究により新たな生命現象が次々と明らかになっている3)。
3.メタボロミクスと生化学研究
メタボロミクスは低分子化合物を対象にする学問であることから、生化学研究と関係が深い。筆者らは、これまでのメタボロミクス研究史において、いくつかの重要な研究があると考えている。1つは、Oliver Fiehnらが2000年に発表した植物機能ゲノミクスにおけるメタボロミクスの可能性を示した研究である4)。この論文では、表現型として差異の見られない遺伝子欠損株においても明確な代謝変化が存在し、遺伝子機能を捉える上で代謝 (メタボローム) という階層を捉える意義が明確に示されている。また、2009年に報告されたオンコメタボライトの同定は、メタボロミクス研究の火付け役となった5)。この論文では、癌細胞ではイソクエン酸デヒドロゲナーゼのミスセンス変異が存在し、この変異によって産生される代謝物であるD-2-ヒドロキシグルタル酸 (D2HG) が、がんの進展に寄与することが示されている。その後の研究により、D2HGはα-ケトグルタル酸デヒドロゲナーゼ酵素群との競合的な阻害を介して低酸素誘導因子-1 α (HIF-1 α) の安定化やヒストンの脱メチル化の抑制が行われることが明らかにされている6)。また近年、腸内細菌が特異的に産生する短鎖脂肪酸や二次胆汁酸などの代謝物と腸管免疫、および腸内細菌の種をまたいだメタボリックリレーの理解に資する研究が相次いでおり、生物をまたいだ代謝コミュニケーションを理解することの重要性が認識されつつある7)。上記以外にも、メタボロミクスは様々な研究において重要な生化学的知見をもたらしている。一方、いま何ができて、何が実際にできないのか、それらを整理することは重要なことのように思う。そこで以降では、メタボローム解析においても「代謝パスウェイ解析」に焦点を当て、現状の課題を整理したい。
4.メタボロミクスによる生化学ネットワークの理解
質量分析を用いたメタボローム解析は、原核生物、植物、菌類からヒトに至るまで、様々な生物における代謝プロセスを理解するために用いられる。中でも、分析対象を定めないノンターゲット解析によるアプローチでは、できるだけ多くの代謝物を一度に検出し、その中から有意に変動する代謝物を同定するというプロセスを経る。しかし、そのような大規模かつ複雑なデータセットの生物学的解釈はメタボローム解析の出力結果 (すなわち、サンプル名×代謝物名×発現量のデータ行列) のみでは困難である。その課題に対処するアプローチの1つとして、生化学 (既知の基質および反応生成物) のネットワーク (以降では代謝パスウェイと呼ぶ) へメタボローム情報を投影するデータ統合・可視化 (パスウェイマッピング) がある。これによりメタボローム解析結果に対して、酵素反応に関する事前知識に基づいた解釈を与えることが可能となる。一方、今のメタボロミクス技術では、既知の生化学反応 (たとえば解糖系、クエン酸回路、ペントースリン酸回路) の何パーセントに情報をマッピングできるだろうか?
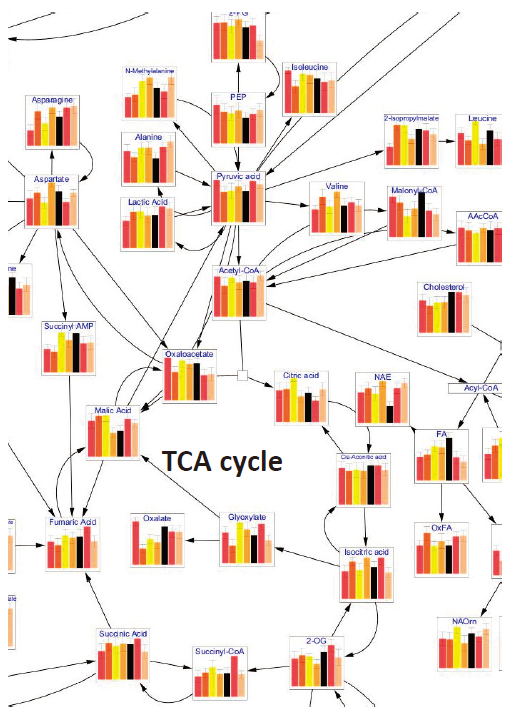
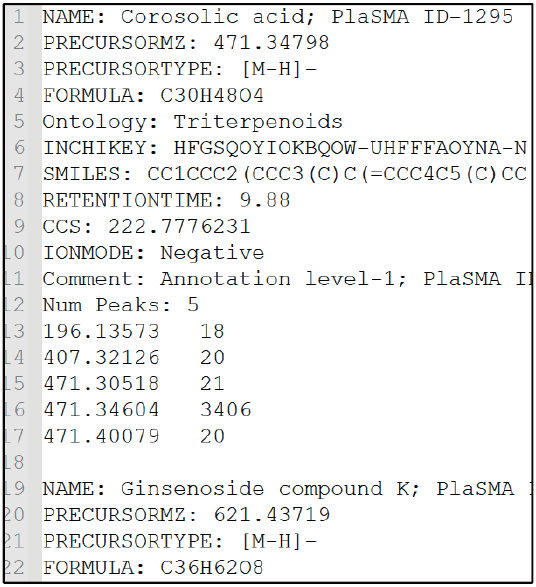
基本的なメタボロミクス研究ワークフローにおいて図1のようなパスウェイ投影は、質量分析によって検出されたピーク情報に代謝物情報を付与 (アノテーション) した後に行われる。アノテーションには、スペクトルライブラリーと呼ばれる化学構造特異的なマススペクトルパターンを集積したデータベースが用いられる (図2)。一方、ヒト、マウス、酵母、シロイヌナズナ、そして大腸菌といった生物種のゲノムスケール代謝モデルデータベースに対し、このようなスペクトルデータベースのカバー率は40%程度である8)。さらに質量分析では、細胞中に存在する代謝物すべてを検出できるわけではない。細胞内の多様な代謝物濃度のダイナミックレンジは (脂質分子のみに着目したとしても) 108を超える一方、質量分析装置のダイナミックレンジは104程度である9)。さらに、メタボロームの対象である低分子の物性は多様であり、クロマトグラフィーやイオン化法ごとに検出できる成分対象が異なる。そのため、通常の (KEGGのような) 代謝パスウェイデータベースにマッピングできるものは、1つの計測手法のみでは10%に満たない。もちろん、着目する代謝パスウェイに焦点を絞ったターゲット解析を用いれば、その特定の代謝パスウェイを占める代謝物アノテーションのカバレッジは上昇する。実際、最新のターゲット分析系を用いれば、少なくとも解糖系・ペントースリン酸経路・クエン酸回路・核酸代謝・アミノ酸代謝・脂質代謝といった主要代謝パスウェイ構成代謝物はおおよそ捉えることができる。しかしながら、それは決して1つの計測手法で完結するものではなく、複数の手法を必要とする。そのため、 着目する代謝経路の物性および濃度範囲を調査し、適切な代謝物抽出のための前処理や、クロマトグラフィー、およびイオン化法・質量分析計測手法の検討を行わなれなければならない。また、現実的なことであるが、メタボローム解析をするための設備投資はとても費用がかかる。現在、そのような社会ニーズに合わせて色々な企業がメタボローム受託サービスを開始しており、上述したように自分の目的に沿った代謝物プロファイリングを提供している会社に外注することが、1つ現実的な手法であるように思う。
図1 メタボローム解析で得られた結果を代謝マップ投影した一例
図2 マススペクトルライブラリーの一例
5.ノンターゲット解析の魅力
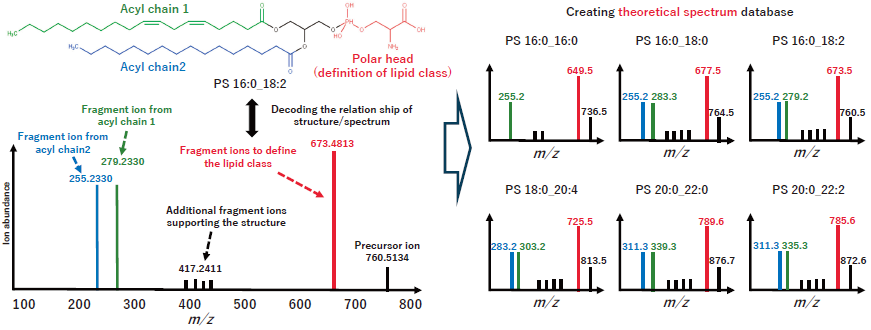
前項には、ノンターゲット解析の網羅性は、まだまだ生化学ネットワークの全容を捉えることができていないことを述べた。一方、当然魅力もある。それは、既存の代謝パスウェイに掲載されていない代謝物も発見できる点にある。もちろん、完全に新規構造を提案できる場合は少ないが、少なくとも着目する生物種にとっては新しい知見である、というケースは多く存在する。また現在、マススペクトルの機械学習法に基づいた未知スペクトルの構造推定研究も広く行われており、前述したマススペクトルライブラリーに登録の無い代謝物構造も捉えることが可能な時代となっている。たとえば筆者らの脂質メタボローム解析 (リピドミクス) では、標準品に由来するマススペクトルライブラリーを必要とせず、真に網羅的な脂質代謝物プロファイリングが可能になってきた。これは、1つの脂質クラス内で生成されるタンデムマススペクトル (MS/MS) が体系化できることに起因する (図3)10)。
![]()
そのため、ある脂質クラスとMS/MSの関連性が明らかになれば、その脂質クラスに含まれる脂質分子種のMS/MSライブラリーを網羅的に生成することが可能となる。現在、国際的な脂質データベースであるLIPIDMAPS11)に登録されている脂質分子種は合計で5万種ほどであるが、ホスファチジルコリンを脂質クラスの1つと数えた場合、脂質クラスの登録数は300ほどである。脂質の構造多様性は、このような極性基と多様性と、1つの脂質クラスに存在する脂肪酸側鎖の多様性に起因する。一方、上述したように、1つの脂質クラスのマスフラグメンテーションパターンは、脂肪酸側鎖の組成によってあまり影響を受けないことから、脂質クラス1つ1つについて標準品を揃え、脂質構造とMS/MSの関連性を紐解くことができれば、現在知られている脂質分子種のほとんどを捉えることができると言える。実際、筆者らの以前の研究では、9種類の藻類から1000分子種を超える脂質分子を捉えることに成功した。また、脂質プロファイルの類似度に基づき藻類分類を行ったところ、taxonomyによる古典的な分類結果とよく一致する結果を得た12)。このことは、藻類の細胞膜を形成する脂質分子の観点から、藻類の形質 (ひいては進化) を解き明かすことが可能であることを示唆している。
また筆者らは近年、ノンターゲットリピドミクスの更なる発展により、生物試料から8000以上の脂質分子構造を捉えることに成功している13)。その中には、上述したLIPIDMAPSデータベースに登録の無い、新規の構造も複数存在する。現在は、このようなノンターゲットリピドミクスにより明らかになった脂質多様性の生物学的重要性について明らかにするための研究を行っている。つまり、新たな代謝物がどのような細胞・遺伝子・タンパク質によって作り出され、それが作れなくなった時にいったい何が起こるのか、そのような新たな科学的問いを創成できるのがノンターゲット解析の最大の魅力であり、これにより新たなバイオロジーが開拓されるといっても過言ではない。
6. ノンターゲット解析から得られる新たな生化学的知見の創出
上述したように、ノンターゲット解析では、 (1) 代謝パスウェイに存在する既知代謝物をすべて検出できるわけではない一方、 (2) 代謝パスウェイに存在しない代謝物を検出することが可能である。つまり、メタボロミクスでプロファイリング可能な分子種と、生化学的知見の包含関係で重なるもの以外の部分がとても多く、最近では (2) の代謝物情報がノンターゲット解析により増えてきているのが現状である。このような新しい分子を、既知の生化学ネットワークに当てはめて考えることはとても難しい反面、生化学研究においてはその分子も含めたオミクスネットワークを可視化し、生命現象を記述したく思う。
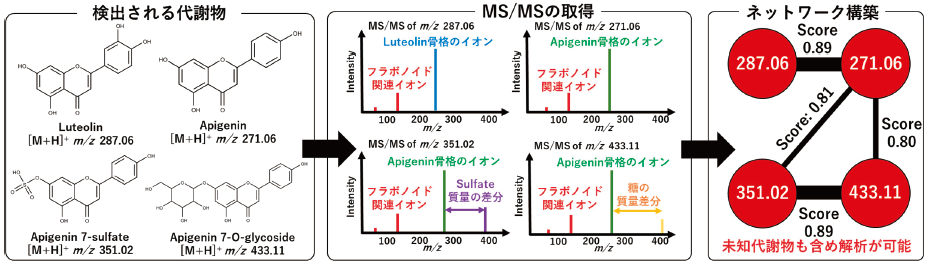
そのためのアプローチとして、マススペクトルの類似度に基づいた分子ネットワーク法が広く用いられる14)(図4)。この手法は、構造の類似するものは類似したMS/MSスペクトルを生成するという仮説に基づく。そして、分子量の異なる分子が産生するMS/MSスペクトルの類似度を評価する計算法がいくつか考案され、類似度が高いMS/MSを持つ代謝物を線 (エッジ) で繋ぐ。この手法では、既知の生化学ネットワークの知識とは全く異なった代謝物の関連性が表現されることもあるが、上述したようなメタボロミクスでしか捉えられない代謝物情報もネットワーク上で可視化できるメリットがある。また、KEGGのような既存の代謝パスウェイとして関連付けられる代謝物を異なるエッジプロパティにより結合させることで、生化学ネットワークとMS/MSスペクトルネットワークを同時に可視化することもできる15)。この手法のもう1つの利点としては、未知のMS/MSスペクトルの構造が、既知代謝物のMS/MSスペクトルと関連付けられることで帰納法的に新規構造推定を行うことができる点にある。現在メタボロミクスで利用可能なインフォマティクスツールおよびデータベースによって、質量分析によって検出されるイオン (ノード) およびその関連性 (エッジ) に様々なアノテーション情報を付与することが可能であり、ソーシャルネットワーキングシステムで採用されるようなコミュニティ検出手法を適用することでヒトでは到達できない生化学の新たな発見が生み出される可能性がある。
図4 マススペクトル類似度に基づいた分子ネットワーク法の概念図
7.ノンターゲット解析とセミ・ノンターゲット解析
メタボロミクスの技術開発により、生化学ネットワークの知識を超えた情報が複数生み出されることは記載した。一方筆者らは、メタボロミクスの1つの課題としては分析化学的なダイナミックレンジ (代謝物の検出可能範囲) の狭さにあると考えている。今のインフォマティクスおよびデータベースの知見があれば、質量分析でイオンとして検出され、そのMS/MSスペクトルが取得されていれば、そのイオンに対して何らかのアノテーション情報が付与される。一方、検出限界以下もしくはMS/MSが取得されていない場合、アノテーションのつけようがない。代謝パスウェイにおける反応中間体の多くが非検出であるという事実は、インフォマティクスの問題というよりも質量分析装置の感度の問題である。そのため、着目する代謝物構造特異的に濃縮する手法や、誘導体化など、生物試料に対する前処理が重要になってくる。前処理における特定代謝物の濃縮と、メタボロミクスによって検出できる網羅性はトレードオフの関係にある。つまり、メタボロミクスという言葉1つとっても、着目する代謝パスウェイによって前処理・分析手法・データ解析手法は異なる。そして現在、代謝物物性ごとに網羅性を担保したセミ・ノンターゲット解析のアプローチが次々に開発されている。たとえば、脂質メタボローム解析におけるホスホイノシチド (PIPs) はその1例であるように思う16)。リン酸基が多くイノシトール環に存在するPIPsは、分析用カラムやLC配管表面に存在する金属性官能基と吸着し、ピークがブロードになってしまうため通常検出できない。このため、リン酸基をメチル化することで疎水性を上げ、カラムにしっかりと保持させる (これはつまり、ピークをシャープにすることにつながる) ことで、高感度の検出を可能にしている。
おわりに
本稿は、メタボロミクスと生化学というテーマで、メタボロミクスで用いられる技術について考察した。インフォマティクス技術やデータベースが進歩したことにより、ノンターゲット解析により得られるピーク情報に付与できる代謝物情報の網羅性が向上した。そして現在、分析系のダイナミックレンジの限界により、最終的には装置の検出感度の壁が存在することを述べた。低分子の化学的物性は多様である一方、着目する代謝パスウェイ上の代謝物に適した前処理・濃縮を行うことで、目的代謝物群を高感度に捉えることが可能になる。また、このような前処理と上述したノンターゲット解析の手法を組み合わせることで、予期しなかった新たな代謝中間体や新規物質が見つかるかもしれない。新しい分子を発見し、新しい代謝の世界を開拓するためには、分析化学や情報科学を含む工学技術の研究開発が必要不可欠であり、筆者らの研究室においても、生化学における新たな発見に貢献できるように日々精進していきたいと思う。
参考文献
1) Oliver, S. G.: Trends Biotechnol, 16, 447 (1998).
2) Naz, S., Moreira dos Santos, D. C., Garcia, A., Barbas, C.: Bioanalysis, 6, 1657 (2014).
3) Alseekh, S., Aharoni, A., Brotman, Y., Contrepois, K., D'Auria, J., Ewald, J., Ewald, J. C., Fraser, P. D., Giavalisco, P., Hall, R. D., Heinemann, M., Link, H., Luo, J., Neumann, S., Nielsen, J., Perez de Souza, L., Saito, K., Sauer, U., Schroeder, F. C., Schuster, S., Siuzdak, G., Skirycz, A., Sumner, L. W., Snyder, M. P., Tang, H., Tohge, T., Wang, Y., Wen, W., Wu, S., Xu, G., Zamboni, N., Fernie, A. R.: Nat Methods, 18, 747 (2021).
4) Fiehn, O., Kopka, J., Dörmann, P., Altmann, T., Trethewey, R. N., Willmitzer, L.: Nat Biotechnol, 18, 1157 (2000).
5) Dang, L., White, D. W., Gross, S., Bennett, B. D., Bittinger, M. A., Driggers, E. M., Fantin, Jang, H. G., Jin, S., Keenan, M. C., Marks, K. M., Prins, R. M., Ward, P. S., Yen, K. E., Liau, L. M., Rabinowitz, J. D., Cantley, L. C., Thompson, C. B., Vander Heiden, M. G., Su S. M.: Nature, 462, 739 (2009).
6) Xu, W., Yang, H., Liu, Y., Yang, Y., Wang, P., Kim, S.-H., Ito, S., Yang, C., Wang, P., Xiao, M.-T., Liu, L.-L., Jiang, W., Liu, J., Bin Wang, J.-Z., Frye, S., Zhang, Y., Xu, Y., Lei, Q., Guan, K.-L., Zhao, S., Xiong, Y.: Cancer Cell, 19, 17 (2011).
7) Bauermeister, A., Mannochio-Russo, H., Costa-Lotufo, L. V., Jarmusch, A. K., Dorrestein, P. C.: Nat Rev Microbiol, 20, 143 (2022).
8) Frainay, C., Schymanski, E. L., Neumann, S., Merlet, B., Salek, R. M., Jourdan, F., Yanes, O.: Metabolites, 8, 15 (2018).
9) Burla, B., Arita, M., Arita, M., Bendt, A. K., Cazenave-Gassiot, A., Dennis, E. A., Ekroos, K., Han, X., Ikeda, K., Liebisch, G., Lin, M. K., Loh, T. P., Meikle, P. J., Orešič, M., Quehenberger, O., Shevchenko, A., Torta, F., Wakelam, M. J. O., Wheelock, C. E., Wenk, M. R.: J Lipid Res, 59, 2001 (2018).
10) Kind, T., Liu, K.-H., Lee, D. Y., DeFelice, B., Meissen, J. K., Fiehn, O.: Nat Methods, 10, 755 (2013).
11) Sud, M., Fahy, E., Cotter, D., Brown, A., Dennis, E. A., Glass, C. K., Merrill Jr, A. H., Murphy, R. C., Raetz, C. R. H., Russell, D. W., Subramaniam, S.: Nucleic Acids Res, 35, D527 (2007).
12) Tsugawa, H., Cajka, T., Kind, T., Ma, Y., Higgins, B., Ikeda, K., Kanazawa, M., VanderGheynst, J., Fiehn, O., Arita, M.: Nat Methods, 12, 523 (2015).
13) Tsugawa, H., Ikeda, K., Takahashi, M., Satoh, A., Mori, Y., Uchino, H., Okahashi, N., Yamada, Y., Tada, I., Bonini, P., Higashi, Y., Okazaki, Y., Zhou, Z., Zhu, Z.-J., Koelmel, J., Cajka, T., Fiehn, O., Saito, K., Arita, M., Arita, M.: Nature Biotechnology, 38, 1159 (2020).
14) Nguyen, D. D., Wu, C.-H., Moree, W. J., Lamsa, A., Medema, M. H., Zhao, X., Gavilan, R. G., Aparicio, M., Atencio, L., Jackson, C., Ballesteros, J., Sanchez, J., Watrous, J. D., Phelan, V. V., Wiel, C., Kersten, R. D., Mehnaz, S., Mot, R., Shank, E. A., Charusanti, P., Nagarajan, H., Duggan, B. M., Moore, B. S., Bandeira, N., Palsson, B., Pogliano, K., Gutiérrez, M., Dorrestein, P. C.: P Natl Acad Sci USA, 110, E2611 (2013).
15) Zhou, Z. W., Luo, M., Zhang, H., Yin, Y., Cai, Y., Zhu, Z.-J.: Nat Commun, 13, 6656 (2022).
16) Morioka, S., Nakanishi, H., Yamamoto, T., Hasegawa, J., Tokuda, E., Hikita, T., Sakihara, T., Kugii, Y., Oneyama, C., Yamazaki, M., Suzuki, A., Sasaki, J., Sasaki, T.: Nat Commun, 13, 83 (2022).