【追悼学術記事】
上田宏先生が生み出した分子機械たち
大室有紀
株式会社島津製作所
2022年12月23日に急逝された東京工業大学上田宏教授は、タンパク質工学分野における第一人者で、タンパク質機能を利用・改変した数々の巧みな分子機械を創出されてきました。それらの分子機械の中から、代表的な技術であるオープンサンドイッチ免疫測定法・FlimPIA・quenchbody・パトロール酵母について、紹介させていただきます。
1.オープンサンドイッチ法
1-1 オープンサンドイッチ免疫測定法1-38)
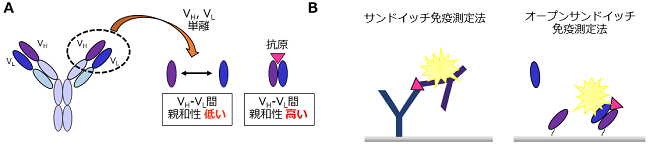
抗体は、各2本のH鎖とL鎖からY字型の構造を取っている。上田先生は、抗体からH鎖の抗原結合部位であるVH・L鎖の抗原結合部位であるVLを単離するというユニークな手法をとられた。そして、抗原が存在しない時、VH-VL間の親和性は非常に低いのに対し、抗原存在時ではVH-VL間の親和性が著しく上昇することで、VH-抗原-VLの三者複合体が形成される現象を見出された (図1B)。この現象を利用して開発されたのが、「オープンサンドイッチ免疫測定法」である。
図1 オープンサンドイッチ法
それまでの免疫測定では、主に2種類の抗体を使うサンドイッチ免疫測定法が利用されていた。これは抗原に結合する2種類の抗体を使用し、抗原を2つの抗体で挟み込むようにして検出する (図1B)。サンドイッチ免疫測定法に対して、オープンサンドイッチ免疫測定法の画期的な点は、分子量1000以下の低分子が検出可能なことである。オープンサンドイッチ免疫測定法よって、ホルモン様物質ビスフェノールA・タンパク質のチロシンリン酸化・セリンリン酸化・カビ毒ゼアラレノン・骨疾患マーカーペプチド・糖質ステロイド・甲状腺ホルモン等、様々な低分子が測定された。
1-2 オープンサンドイッチ法の細胞増殖制御技術への応用39-47)
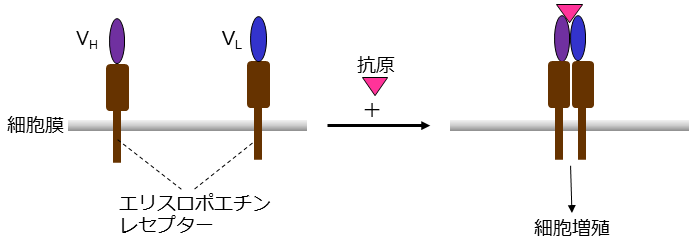
オープンサンドイッチ法の利用は免疫測定にとどまらない。その一例として、VH、VLを各々、細胞増殖因子エリスロポエチン等の受容体における細胞膜-細胞質に存在する構造と融合させたキメラ受容体による細胞増殖制御技術がある。抗原を添加した時、VHとVLが相互作用することにより、受容体が二量体になり、細胞増殖のための細胞内シグナル伝達が誘起される (図2)。実例の1つとして、IL-3依存的に増殖するBa/F3細胞にこのキメラ受容体を発現させた結果、IL-3非存在下において細胞はほぼ増殖しなかったが、抗原を添加すると添加濃度依存的な細胞増殖が観察された。本法は、細胞機能を人工的に制御できる画期的な手法である。
図2 オープンサンドイッチ法の細胞増殖制御技術への応用
2.FlimPIA48-55)
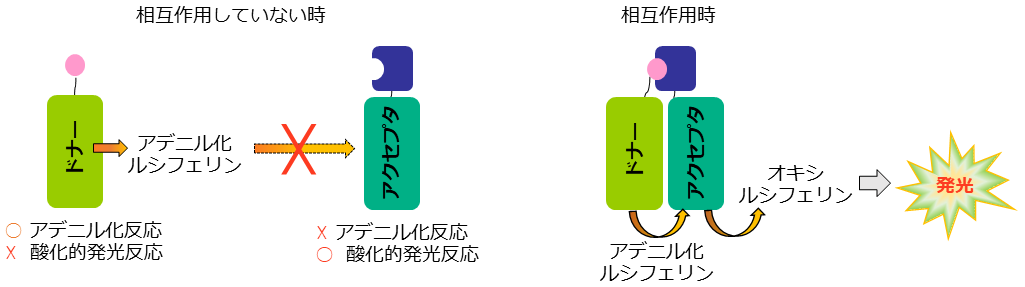
抗体工学分野において著名な上田先生であるが、長年、発光酵素、特にホタル由来発光酵素の研究を行っておられた。ホタル由来発光酵素の発光反応は、基質ルシフェリンをアデニル化するアデニル化反応と、アデニル化ルシフェリンを酸化してオキシルシフェリンを産生し、発光を起こす酸化的発光反応の、2つの半反応に大別できる。ホタル由来発光酵素はN末ドメインとC末ドメインから構成され、面白いことに、アデニル化反応後にN末ドメインが約140°回転し、酸化的発光反応が起きる。先生は、タンパク質工学によってホタル由来発光酵素を改変して、この2半反応をコントロールすることに長く興味を持たれていた。その知見の集積によって創出されたのが、タンパク質間相互作用検出系「FlimPIA (Firefly luminescent intermediate Proteinprotein Interaction Assay)」である。
FlimPIAのために、アデニル化反応はほぼ正常だが、酸化的発光反応が非常に遅いホタル由来発光酵素変異体「ドナー」と、逆に、アデニル化反応が非常に遅く、酸化的発光反応はほぼ正常な変異体「アクセプタ」を作製した。ドナー・アクセプタに相互作用タンパク質を各々融合させると、相互作用時、ドナーとアクセプタが近接するので、ドナーが産生したアデニル化ルシフェリンをアクセプタが利用して酸化的発光反応が起き、発光が観察される (図3)。
図3 FlimPIA
本法は、迅速・簡便なタンパク質間相互作用であるだけでなく、他の汎用的な相互作用検出系である蛍光共鳴エネルギー移動・PCA (Protein-fragment Complementation Assay) と比較して、これらの方法では測定できない大きなタンパク質の相互作用検出が可能である、検出限界濃度が低い、といったメリットを持つ 優れた手法である。
3.Quenchbody56-77)
Quenchbodyは、オープンサンドイッチ法を発展させる過程で、実験結果を丁寧に検証する上田先生の日頃の姿勢から生まれた、まさにセレンディピティによる開発だと言える。
VH・VLに各々赤色蛍光色素・緑色蛍光色素を修飾することにより、蛍光共鳴エネルギー移動による検出を行っていた。通常、蛍光共鳴エネルギー移動においては、赤色蛍光のエネルギーがすべて緑色蛍光に移動することはないので、赤色蛍光の減少度の方が緑色蛍光の上昇率が大きい。しかし、先生は、この測定では、赤色蛍光の減少度より緑色蛍光の上昇率の方が大きいことに気がつかれた。そこで、この点について検証を行うため、VHだけを赤色蛍光色素で標識して非標識のVL断片と混合したところ、蛍光強度が抗原濃度依存的に増大する現象が観察された。先生はこの現象について、抗原非結合時には蛍光色素からVH・VL内の抗原結合部位付近のトリプトファン (Trp) 残基への光誘起電子移動による蛍光消耗 (quench) が起き、抗原結合により、蛍光色素がTrp残基から離れて蛍光が回復するのではないかと考えた。このことはVH・VL内のTrpを変異させた時には、quenchが起こらなかったことから証明された。このように、先生の考察力・深い知識とセレンディピティが重なり、抗原に結合していない時は消光状態で、抗原に結合すると光る抗体「quenchbody」が生み出された。さらに、すべての抗体においてquenchを起こすTrp残基が保存されていることから、quenchbodyのメカニズムに汎用性があること、適用可能な抗体の形態は、VH/VLだけでなく、scFv・Fab・VHH・全長抗体に適用できることが明らかとなった。
本法によって、骨疾患マーカーペプチド・ビメンチンのセリンリン酸化・インフルエンザウイルスのタンパク質ヘマグルチニン・ステロイドホルモン・農薬・細胞表面がんマーカー・SARS Cov2・細胞内p53等について、高感度、かつ迅速な検出がされた。この技術を基盤に、2022年6月に東工大発ベンチャー「HikariQ Health」を立ち上げ、今こそ社会実装に挑むという段階であった。
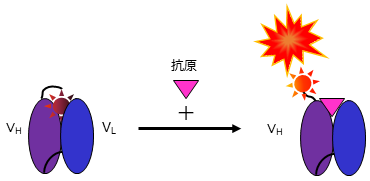
図4 quenchbody
4.パトロール酵母78)
最近、上田先生は、細胞壁を持つため丈夫で、増殖が早い酵母を利用することで、安価であり、かつ細胞外から入るシグナルを細胞内で増幅することによって高感度検出ができるバイオセンサー「パトロール酵母」を開発された。
まず、2つの融合タンパク質、カフェインを認識するVHH-エリスロポエチン受容体の膜貫通領域-ユビキチンN末断片とVHH-エリスロポエチン受容体の膜貫通領域-ユビキチンC末断片-転写因子LexA-VP16を酵母に発現させた。VHHは細胞外に位置し、ユビキチン断片と転写因子は細胞内に位置する。細胞内には、転写因子LexAVP16が結合するプロモーターにβ-ガラクトシダーゼをコードする遺伝子を繋げることで、LexA-VP16が結合時にβ-ガラクトシダーゼが発現する。2つのVHHがカフェインを挟み込むように結合することにより、細胞内でユビキチンの2断片が近接し、元の構造に戻ることにより、ユビキチンのC末断片から転写因子が切り離される。遊離した転写因子がプロモーターに結合することで、発現したβ-ガラクトシダーゼのシグナルを検出することにより、カフェイン量を測定した (図5A)。この方法によって、コーラ中のカフェインを検出することに成功した。
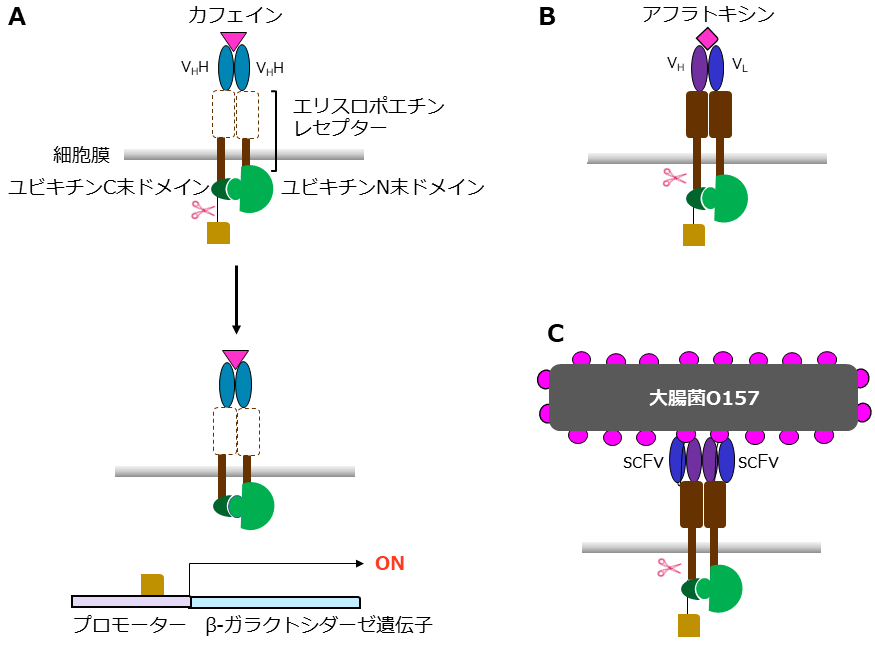
図5 パトロール酵母
次に、カビ毒アフラトキシンの検出をアフラトキシンに結合するVH・VLを利用して、アフラトキシンの検出を行った。VH-エリスロポエチン受容体-ユビキチンC末断片-転写因子・VL-エリスロポエチン受容体-ユビキチンN末断片を酵母に発現させた。アフラトキシン結合時、VH-アフラトキシン-VLの三者複合体ができることにより、β-ガラクトシダーゼのシグナルが検出された (図5B)。さらに、大腸菌O157の細胞壁表面抗原を認識する一本鎖抗体を利用して、大腸菌O157の検出を行った。一本鎖抗体-エリスロポエチン受容体-ユビキチンC末断片-転写因子・一本鎖抗体-エリスロポエチン受容体-ユビキチンN末断片を酵母に発現させた。大腸菌O157の細胞表層には抗原が密に存在するので、一本鎖抗体が抗原に結合することにより2プローブが近接する確率が高くなるため、近接時にβ-ガラクトシダーゼのシグナルが検出された (図5C)。
本法は、上記のように様々なサイズの3種類の標的を検出でき、さらに、VHH・ VH/VL・一本鎖抗体といった様々な形態の抗体を利用できることから、汎用性の高い技術である。
5.おわりに
誌面の関係上、ここに書けなかったすばらしい分子機械がまだたくさんあります。長い間、共同研究者・ポスドク・助教として、上田先生と研究を行い、たくさん勉強させていただきました。拙い文章ですが、感謝を込めて精一杯、書かせていただきました。この追悼学術記事を弔事として捧げます。
代表文献
1) Ueda, H. et al.: Nature Biotechnol., 14, 1714-1718 (1996).
2) Suzuki, C. et al.: J. Immunol. Methods, 22, 171-184 (1999).
3) Ueda, H. et al.: Biotechniques, 27, 738-742 (1999).
4) Arai, R. et al.: Protein Eng. Des. Sel., 13, 369-376 (2000).
5) Suzuki, C. et al.: Anal. Biochem., 286, 238-246 (2000).
6) Arai, R. et al.: Anal. Biochem., 289, 77-81 (2001).
7) Ueda, H.: J. Biosci. Bioeng., 94, 614-619 (2002).
8) Yokozaki T. et al.: Anal Chem., 74, 2500-2504 (2002).
9) Ueda, H. et al.: J. Immunol. Methods, 279, 209-218 (2003).
10) Aburatani, T. et al.: Anal. Chem., 75, 4057-4064 (2003).
11) 上田 宏: 生化学, 76, 670-674 (2004).
12) Sasajima, Y. et al.: Biotechnol. Prog., 22, 968-973 (2006).
13) Suzuki, T. et al.: Anal. Sci., 23, 65-70 (2007).
14) Lim, S. L. et al.: Anal. Chem., 79, 6193-6200 (2007).
15) 上田 宏: 薬学雑誌, 127, 71-80 (2007).
16) Dong, J. H. et al.: Anal. Biochem., 386, 36-44 (2009).
17) Ihara, M. et al.: Anal. Chem., 81, 8298-8304 (2009).
18) Sakata, T. et al.: Anal Chem., 81, 7532-7537 (2009).
19) Ihara, M. et al.: Lab Chip, 10, 92-100 (2010).
20) Iwai, H. et al.: Protein Eng. Des. Sel., 23, 85-93 (2010).
21) 大室 (松山) 有紀他: 細胞周期フロンティア (共立出版) , 11-17 (2010).
22) Sasajima, Y. et al.: J. Immunol. Methods, 30, 57-63 (2010).
23) Islam, K. N. et al.: Bioanalysis, 2, 1683-1687 (2010).
24) Islam, K. N. et al.: Anal. Chem., 83, 1008-1014 (2011).
25) Dong, J. et al.: J. Immunol. Methods, 30, 1-7 (2012).
26) Minami, K. et al.: Bioconjug. Chem., 23, 1463-1469 (2012).
27) Liu, X. et al.: Anal. Sci., 28, 861-867 (2012).
28) Dong, J. et al.: PLoS One, 8 (4):e61158 (2013).
29) Hara, Y. et al.: Anal Chim. Acta, 2, 107-113 (2013).
30) Hasan, S. et al.: Anal. Sci., 29, 871-876 (2013).
31) Chung, C. I. et al.: Anal. Chem., 87, 3513-3519 (2015).
32) Chung, C. I. et al.: J. Biosci. Bioeng., 123, 272-276 (2017).
33) Dong, J. et al.: Analyst, 142, 787-793 (2017).
34) Ohmuro-Matsuyama, Y., Ueda, H.: Anal. Chem., 90, 3001-3004 (2018).
35) Su, J. et al.: J. Biosci. Bioeng., 128, 677-682 (2019).
36) Kanai, Y. et al.: ACS Sens., 5, 24-28 (2020).
37) Harada, Y. et al.: Anal. Sci., 37, 455-459 (2021).
38) Okuyama, H. et al.: Anal. Chem., 18, 7210-7219 (2021).
39) Kawahara, M. et al.: Cytotechnology, 52, 171-179 (2006).
40) Kawahara, M. et al.: J. Biotechnol., 133, 154-161 (2008).
41) Liu, W. et al.: Biotechnol. Bioeng., 101, 975-984 (2008).
42) Sogo, T. et al.: J. Immunol. Methods, 337, 16-23 (2008).
43) Sogo, T. et al.: Cytokine, 46, 127-136 (2009).
44) Tanaka, Y. et al.: Biotechnol. Prog., 25, 1138-4115 (2009).
45) Kawahara, M. et al.: Cytokine, 55, 402-408 (2010).
46) Kaneko, E. et al.: J. Biosci. Bioeng., 113, 641-646 (2012).
47) Tone, Y. et al.: Hum. Gene. Ther. Methods, 24, 141-150 (2013).
48) Ohmuro-Matsuyama, Y. et al.: Anal. Chem., 85, 7935-7940 (2013).
49) Ohmuro-Matsuyama, Y. et al.: Anal. Chem., 86, 2013-2018 (2014).
50) Kurihara, M. et al.: Biotechnol. J., 11, 91-99 (2016).
51) Ohmuro-Matsuyama, Y., Ueda, H.: Methods Mol. Biol., 1461, 131-142 (2016).
52) Ohmuro-Matsuyama, Y., Ueda, H.: Methods Mol. Biol., 1596, 119-130 (2017).
53) Ohmuro-Matsuyama, Y. et al.: Luminescence, 33, 125-130 (2018).
54) Ohmuro-Matsuyama, Y. et al.: Anal. Biochem., 563, 61-66 (2018).
55) Ohmuro-Matsuyama, Y. et al.: Front. Bioeng. Biotechnol., 9, 778120 (2021).
56) Abe, R. et al.: J. Am. Chem. Soc., 133, 17386-17394 (2011).
57) Jeong, H. J. et al.: Biosens. Bioelectron., 40, 17-23 (2013).
58) Abe, R. et al.: Sci. Rep., 4, 4640 (2014).
59) Jeong, H. J., Ueda, H.: Sensors (Basel), 14, 13285-13297 (2014).
60) 上田 宏: 現代化学, 518, 32-36 (2014).
61) Ueda, H., Dong, J.: Biochim. Biophys. Acta - Proteins and Proteomics, 1844, 1951-1959 (2014).
62) Ohashi, H. et al.: Bioconj. Chem., 27, 2248-2253 (2016).
63) Jeong, H. J. et al.: Anal. Chem., 89, 10783-10789 (2017).
64) Dong, J. et al.: Biotechnol. Bioeng., 117, 1259-1269 (2020).
65) Dong, J. et al.: Biosens. Bioelectron., 165, 112425 (2020).
66) Inoue, A. et al.: ACS Sens., 5, 3457-3464 (2020).
67) Dong, J., Ueda, H.: Sensors (Basel), 21(4), 1223 (2021).
68) Takahashi, R. et al.: Anal. Chem., 93, 7571-7578 (2021).
69) Yasuda, T. et al.: Chem. Commun., (Camb) 57, 8206-8209 (2021).
70) Ning, X. et al.: Sensors (Basel), 21(15):4993 (2021).
71) Inoue, A. et al.: Sci. Rep., 11(1):22590 (2021).
72) Li, H. et al.: ACS Omega, 6, 31009-31016 (2021).
73) Liang, J. et al.: J. Steroid Biochem. Mol. Biol., 221:106118 (2021).
74) Dai, Y. et al.: Chem. Sci., 13, 9739-9748 (2022).
75) Li, H. et al.: Sci. Rep., 12(1), 15496 (2022).
76) Zhu, B. et al.: Analyst, 147, 4971-4979 (2022).
77) Ueda, H. et al.: Trends Cell Biol., In press.
78) Su, J. et al.: Biosens. Bioelectron., 219:114793 (2022).