【トピックス】
抗生物質生産菌が持つ自己耐性酵素の機能解明
佐藤道大、渡辺賢二
静岡県大薬
1.はじめに
これまでに植物や微生物などの天然資源から獲得されてきた低分子化合物は数十万にも上る。その中には、抗生物質をはじめとする生物活性を有する化合物も含まれ、我々人間の生活になくてはならないものも多い。この抗生物質は、ほかの生物の生育を抑制する作用を持つ化合物の総称であるが、なぜ生産菌に対しては、その作用が及ばないのだろうか。この問いに対してはこれまで多くの研究がなされ、生物が有する多様な耐性機構の存在が明らかにされている。現在我々は、その耐性機構の一つを利用した「標的分子指向型ゲノムマイニング」を基盤とする物質探索研究を行っている。最近、このゲノムマイニングにより見出したある抗生物質の生合成研究から、その標的分子、抗生物質耐性酵素およびその耐性機構についてその詳細を明らかにすることができた。これまでの標的分子探索研究とは異なるアプローチの本研究は、生物活性物質の作用機序を解明する新しい方法論となるだろう。本総説では、抗生物質生産菌における耐性機構について、これまでの研究結果を含めて概説する。
2.生物活性物質および生産菌における自己耐性機構
微生物が抗生物質を生産することがわかったのは、アレクサンダー・フレミングによるアオカビにおけるペニシリンの存在を明らかにしたことが端緒となったとされている。それから100余年、抗生物質だけではなく、あらゆる生理・生物活性を指標とした天然物探索研究が盛んになり、これまでに数十万もの天然物が見出されてきた。獲得されてきた化合物は、医農薬品をはじめとし我々人類のQOLを向上させるためになくてはならないものとして利用されている。
これら生物活性物質、特に抗菌活性や抗がん活性などの抗生物質は、必須のハウスキーピング酵素の阻害または不活性化を介して、競合する生物の成長を止める、または制限するために生産されると考えられている。しかし、その標的分子が生産菌にも保存されており、かつそれが生育に必須な酵素である場合、その天然物は生産菌自身にも毒性を示してしまうおそれがある。したがって、自己耐性システムは、生合成された天然物に抵抗性を付与するために産生菌に存在しなければならない。この自己耐性システムのメカニズムについてはいくつか知られており、たとえば積極的に細胞外空間に代謝物を輸送する排出ポンプ、細胞内に残っている高活性の天然物を隔離または代謝するための抵抗性タンパク質、および天然物の阻害を回避するために生産菌のハウスキーピグ酵素を修飾する酵素、などがあげられる (図1)1,2)。微生物が自己耐性のために採用しているもう一つの戦略は、機能的に等価な自己耐性酵素 (Self-Resistance Enzyme, SRE) を保有することである3,4)。SREは標的となるハウスキーピング酵素と配列的に非常に類似しているが、活性を維持しながらも天然物による阻害に対して酵素を非感受性にする変異を含んでいる (図1)4,5)。このSREの配列は、標的となるハウスキーピング酵素と類似しているため、in silico解析によって SREの機能を容易に予測することが可能である。興味深いことに多くのSREは天然物の生合成遺伝子クラスター (Biosynthetic Gene Cluster, BGC) 中にクラスタリングされていることが知られており、天然物生産が生合成遺伝子と転写的に共制御されている。それにより、天然物が細胞内に蓄積し始めるときにはSREが確実に存在することになる。このSREを利用した微生物の作戦は、細菌および真菌類の両方で 天然物生産微生物によって広く用いられている3-5)。
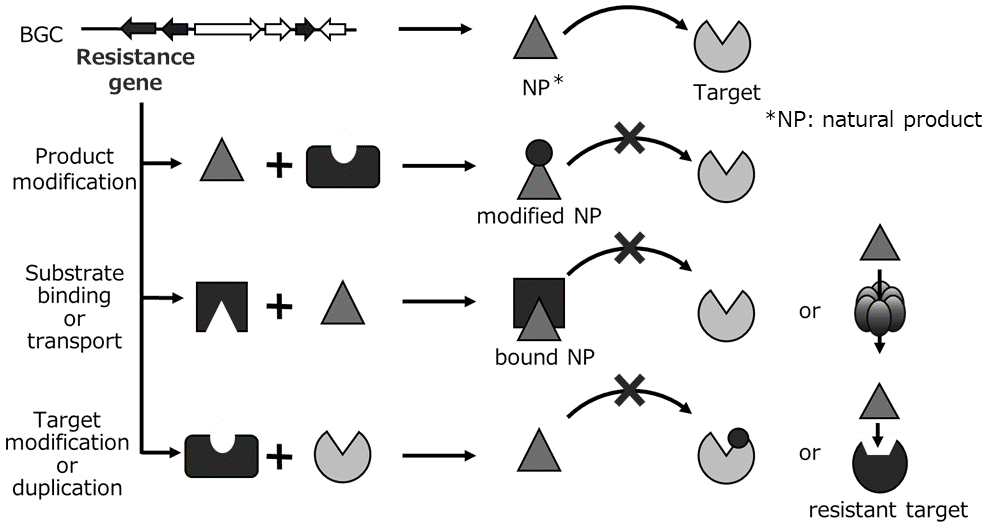
図1 生合成遺伝子クラスター (BGC) に存在する自己耐性メカニズム
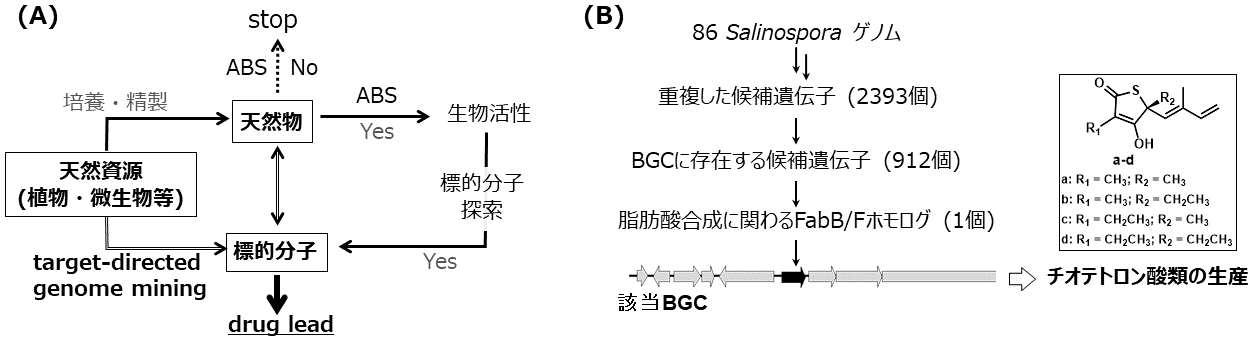
3.標的分子探索
SREによる自己耐性システムは、生産される天然物の標的分子を表していることから、SREを含むBGCを探索することで、目的の標的分子を阻害する化合物を得ることが可能となる。これは標的分子指向型ゲノムマイニング (target-directed genome mining)と呼ばれる。創薬研究において活性を指標としたスクリーニング (Activity Based Screening, ABS) では、化合物を獲得した後、標的分子の探索を明らかにする必要がある。一方で標的分子指向型ゲノムマイニングでは、ゲノム情報から標的分子が明らかになっているため、化合物を獲得することで一挙にドラッグリード化合物の獲得へつながる (図2A)。この概念を実証した初めてのケースが、2015年に報告されたMooreらによるチオテトロン酸類の発見である (図2B)6)。Mooreらは、標的分子をコードする抗生物質耐性遺伝子と未知BGCを相関させることで、目的とする生物活性を有する経路特異的な天然物の獲得を目指した。SREとしては、ゲノム中に2コピー存在するハウスキーピング遺伝子を対象に、かつBGC内に存在している条件で絞り込んだ。絞り込んだSREのうち、脂肪酸合成酵素阻害活性に着目し、チオテトロン酸BGCを見出した。チオラクトマイシン類が脂肪酸合成酵素阻害活性を有することは30年以上前に報告されていたが、この研究報告は、生合成と自己耐性システムを相関させる標的分子指向型ゲノムマイニング戦略を実証した初めての例となった。
![]()
自己耐性酵素をコードする遺伝子は、細菌の天然物BGCだけでなく、真菌類においても存在している。たとえばロバスタチン (HMG-CoA還元酵素を標的とする) やミコフェノール酸 (IMPDHを標的とする) の遺伝子クラスターに、それぞれ化合物に対して非感受性のHMG-CoA還元酵素とイノシン一リン酸デヒドロゲナーゼ (IMPDH) の存在が知られている3)。最近では、Tangらによって糸状菌における標的分子指向型ゲノムマイニングから、植物生育抑制活性を有するアスプテル酸が見出された。さらにそのBGC内に存在する、耐性遺伝子astDの機能解明を進め、AstDの作用機序を明らかにした7)。これらの研究は、微生物における標的分子指向型ゲノムマイニングの実用性を明確に示すものとなった。
我々は最近、FR901483 (1) という化合物の生合成研究を行ってきた。本化合物は、糸状菌培養液から単離された化合物であり、複雑な含窒素三環性骨格を有する (図3)8)。本化合物のBGCには、生合成には関与しないと考えられる酵素phosphoribosylpyrophosphate amidotransferase (PPAT) をコードする遺伝子が存在していた。このことから、PPATは1に対するSREであること、すなわち1の標的分子はPPATであると考えられた (図3)。これは、Aspergillus nidulansを用いた1の全生合成を試みた際、PPATをコードする遺伝子frzKを共発現しなければ1が生産されないことからも支持された9)。すなわち、1は異種発現宿主であるA. nidulansにとって有毒であり、SREを発現することでそれを無毒化していると考えられた。
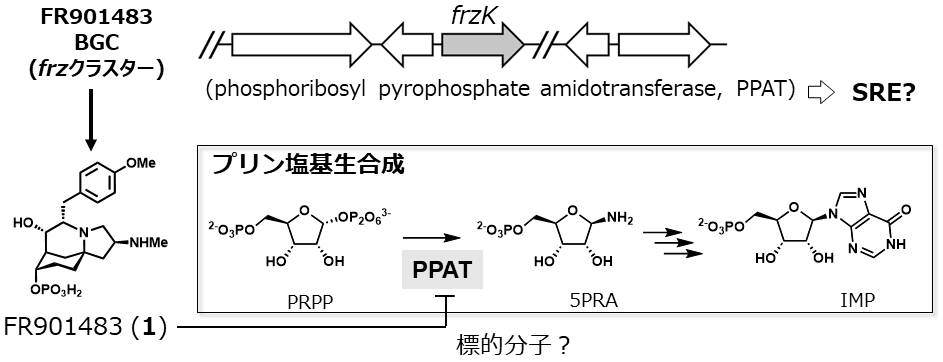
図3 FR901483 (1) 生合成遺伝子クラスターのSRE
4. FR901483の標的分子の同定および生産菌の自己耐性機構の解明
FR901483 (1) は糸状菌Cladobotryum属真菌より、免疫抑制活性を指標に獲得された化合物である。この免疫活性は、現在医療の現場で使用されているサイクロスポリンやタクロリムスなどのIL-2産生阻害活性を有する化合物とはその作用機序が異なることが示唆されているが、その標的分子をはじめとする作用機序は解明されていない8)。先述したように我々は本化合物の生合成研究の過程で、そのBGC内に1の耐性酵素が存在することを見出した (図3)。興味深いことに、本糸状菌ゲノム中には、PPATホモログタンパク質はこのFrzKのみしか存在しておらず、FrzKはSREでありかつ本菌における唯一のPPATであった。PPATは核酸合成の初発を担う重要な一次代謝酵素であり、ホスホリボシル二リン酸 (PRPP) とグルタミンを基質に加水分解およびアミノ基転移酵素の両機能によりホスホリボシルアミン (5PRA) とグルタミン酸を生成する (図3)。
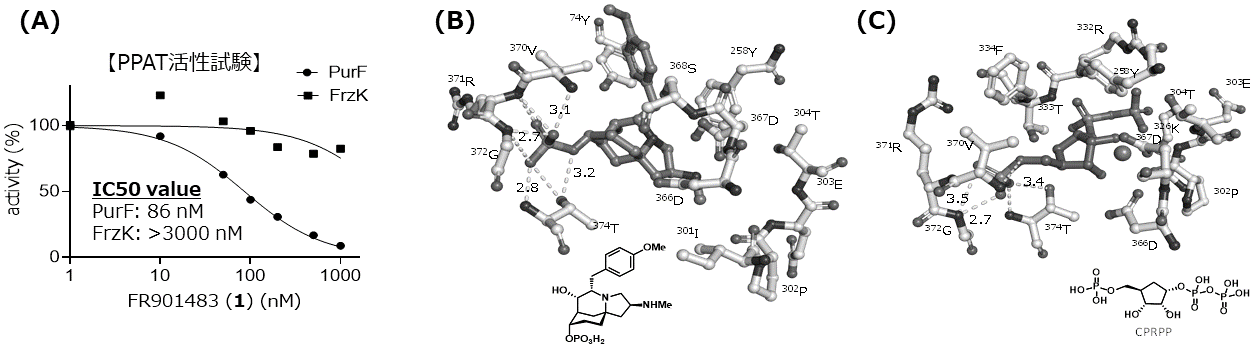
はじめに、1の標的分子がPPATであるかを確かめるために、大腸菌由来PPAT (PurF) を用いて1によるその阻害活性試験を行った。その結果、PurFの活性は濃度依存的に1により阻害されることが明らかとなった。その阻害活性は強く、IC50値はnMオーダーであった。このことから、1の標的分子はPPATであることが示された。一方で、FrzKの活性は1による阻害をほとんど受けないことから、SREであることが確認された (図4A)。次に1の作用機序を明らかにするために、PurFとの共結晶を作製した。得られた共結晶をX線結晶構造解析に供し、PurF-1複合体の結晶構造を明らかにした。この結晶構造から、1はPurFの本来の基質であるPRPP結合部位に結合していることが示された。1のリン酸基はPRPPのモノリン酸と同じく、V370~T374と水素結合を形成しており、1の脱リン酸体においては阻害活性が消失することからも、このリン酸基の存在が活性に重要であることが示唆された (図4BC)。さて、それではどのようにFrzKは1に対する自己耐性能を獲得したのだろうか。続いて1に対する阻害回避メカニズムの解明を目指した。
![]()
PPATはプリン塩基の生合成に関わる酵素であり、原核生物から真核生物まで普遍的に存在し、その活性部位のアミノ酸残基は高度に保存されている。我々は活性部位周辺のアミノ酸残基に1の結合を妨げる要因があると考え、いくつかの生物種におけるPPATのアミノ酸配列のアライメントを調べた。すなわち、ほかの生物種のPPATで保存されており、FrzKのみ保存されていないアミノ酸残基を探索した (図5A)。これらのアミノ酸は、PPATの酵素活性には関与しないが、1の結合に関わるアミノ酸残基であると考えられた。また、並行してFrzK-1のモデリング構造を構築し、PurF-1の共結晶構造との相違点を探索した。
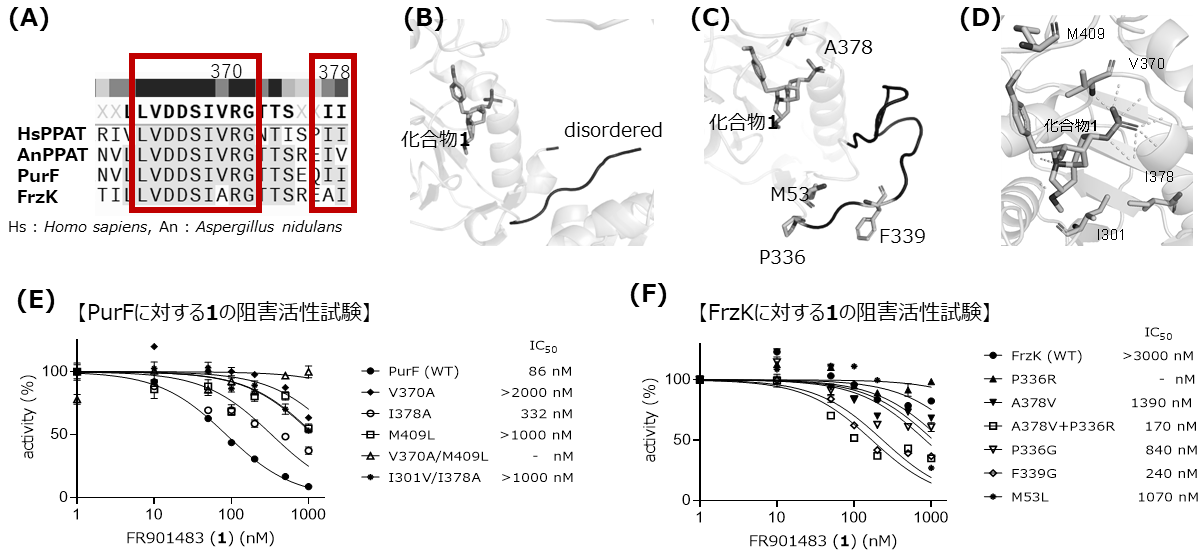

PurFとFrzKは、相同性も高く、3次元構造も高い相同性を有していたが、活性部位の下部に存在するループ構造を構成するアミノ酸残基に違いが見られた (図5BC)。このことから、ループ構造は自由度を持ったコンフォメーションを取ることが明らかとなった。一方で、FrzK-1複合体では、F339やP336の存在によりそのループ構造が固定したコンフォメーションに形成されていることが示唆された。すなわち、FrzKへの1の結合は、ループ構造が障壁となることで妨げられており、PurFにおいては、その障壁がないことから1の活性部位への侵入が可能となると考えられた。これら二つの仮説を検証するため、PurFおよびFrzKのそれぞれに対して、結合に関与すると考えられるアミノ酸残基に変異を導入し、1によるPPAT阻害活性を測定した。
PurFの変異体に関しては、それぞれ、V370A 、I378A、M409L、V370A/M409LおよびI301V/I378Aとなるよう変異を導入した結果、1に対して耐性を持つことが確認された (図5E)。PurF-1複合体結晶構造より、V370A変異体では1との水素結合が消失することで、1との結合親和性が低下したと考えられた。またM409L変異体においては、近傍に存在する1のチロシン残基由来の4-メトキシフェニル基との相互作用の消失が、I378A変異体に関しては、I301との相互作用の消失が影響していると示唆された (図5D)。一方でFrzKにおいては、A378V/P336R、F339GおよびM53L変異体が1から阻害を受ける変異体であることが確認された (図5F)。FrzKのA378はPurFのV370に対応し、1のチロシン残基由来の4-メトキシフェニル基の近くに存在しているアミノ酸である。A378V+P336Rの変異体においては、バリンに置換されたことにより1との水素結合が形成され、結合親和性が高まったため、阻害を受けると考えられた (図5CD)。またF339はFrzKの活性部位下部のループ構造を構成するアミノ酸の一つであり、嵩高いフェニルアラニンをグリシンに置換することで、ループ構造の自由度が大きくなり、FrzK活性部位内に1が取り込まれやすくなったと考えられた。さらに活性部位下部のループ構造付近に位置しているM53に関しても、ロイシンに置換することによってループ構造の自由度が増加したことが要因であると考えられた (図5CF)10)。
5.おわりに
以上のように、我々は標的分子指向型ゲノムマイニングを基盤として、免疫抑制活性を有するFR901483の標的分子を同定し、さらに生産菌の耐性機構について明らかにすることに成功した。通常生合成遺伝子クラスターに存在するSREはゲノム中に2コピー以上存在することが多いが3)、興味深いことに本化合物に対するSREは、生産菌のゲノム中に1コピーしか存在していなかった。標的分子指向型ゲノムマイニングにおいて、重複した遺伝子で絞り込みを行ったMooreらの手法を用いると、今回のような1コピーしか存在しないSREは見出すことはできないため、その対象はより広く選択しなければならない。また、今回見出したPPATホモログタンパク質のFrzKは、アミノ酸配列を大きく変えること無く、構造中のループ構造や化合物の結合に関わるアミノ酸の相互作用をうまく利用した耐性メカニズムを有していることが明らかとなった10)。
最近、グルタミン由来の窒素をDNAの合成に利用する代謝系 (窒素シフト) が、悪性のがん細胞において亢進していることが示された。さらに、がん細胞に発現する約1200種の全ヒト代謝酵素のうち、最も発現量が増加していたPPATがその主因となっていることが明らかとなった11)。このことから、PPATはがん細胞の悪性化に必須の因子であり、治療上有望な標的であると考えられた。しかしながらこれまで有効なPPAT阻害剤は報告されておらず、今回我々が明らかにしたPPAT阻害剤FR901483は、創薬研究において重要な発見となるであろう。標的分子指向型ゲノムマイニングは、化合物と標的が一挙に獲得できる方法で、今回のように新しい標的とその活性化合物が見出すことが可能である。本メソッドを利用した標的分子-化合物探索研究の今後の展開に期待する。
文献
1) Galm, U., Hager, M. H., Van Lanen, S. G., Ju, J., Thorson, J. S., Shen, B.: Chem. Rev., 105, 739 (2005).
2) Weisblum, B.: Antimicrob. Agents Chemother., 39, 577 (1995).
3) O’Neill, E. C., Schorn, M., Larson, C. B., Millan-Aguinaga, N.: Crit. Rev. Microbiol., 45, 255 (2019).
4) Yan, Y., Liu, N., Tang, Y.: Nat. Prod. Rep., 37, 879 (2020).
5) Almabruk, K. H., Dinh, L. K., Philmus, B.: ACS Chem. Biol., 13, 1426 (2018).
6) Tang, X., Li, J., Millan-Aguinaga, N., Zhang, J. J., O’ Neill, E. C., Ugalde, J. A., Jensen, P. R., Mantovani, S. M., Moore, B. S.: ACS Chem. Biol., 10, 2841 (2015).
7) Yan, Y., Liu, Q., Zang, X., Yuan, S., Bat-Erdene, U., Nguyen, C., Gan, J., Zhou, J., Jacobsen, S. E., Tang, Y.: Nature, 559, 415 (2018).
8) Sakamoto, K., Tsujii, E., Abe, F., Nakanishi, T., Yamashita, M., Shigematsu, N., Izumi, S., Okuhara, M.: J. Antibiot., 49, 37 (1996).
9) Zhang, Z., Tamura, Y., Tang, M., Qiao, T., Sato, M., Otsu, Y., Sasamura, S., Taniguchi, M., Watanabe, K., Tang, Y.: J. Am. Chem. Soc., 143, 132 (2021).
10) Sato, M., Sakano, S., Nakahara, M., Tamura, Y., Hara, K., Otsu, Y., Sasamura, S., Taniguchi, M., Hashimoto, H., Tang, Y., Watanabe, K.: J. Am. Chem. Soc., submitted.
11) Kodama, M., Oshikawa, K., Shimizu, H., Yoshioka, S., Takahashi, M., Izumi, Y., Bamba, T., Tateishi, C., Tomonaga, T., Matsumoto, M., Nakayama, K. I.: Nat. Commun., 11, 1320 (2020).