【トピックス】
国産ゲノム編集システム「CRISPR-Cas3」の社会実装
小堀峻吾、平井昭光
C4U 株式会社 研究開発部技術研究センター
1.はじめに
ゲノム編集は、ZFN (Zinc-Finger Nucleases) やTALEN (Transcription Activator-Like Effector Nucleases) などの人工制限酵素またはCRISPR-Cas9 (Clustered Regularly Interspaced Short Palindromic Repeats-CRISPR associated proteins 9) のような人工ヌクレアーゼで標的とするDNAの塩基配列を切断することにより、生物のゲノムの特定の箇所を編集する技術である。特に、第三世代のゲノム編集技術であるCRISPR-Cas9 (Cas9) は、汎用性や精度が高く、いかなる遺伝子に対しても迅速に良好な効率が容易に得られることから、多様な技術基盤に適用されている。Cas9の基礎研究者であるエマニュエル・シャルパンティエ氏 (独マックス・プランク感染生物学研究所長) とジェニファー・ダウドナ氏 (米カリフォルニア大バークレー校教授) にノーベル化学賞が授与されたことは記憶に新しい。
国内でのCas9を利用した高等生物の品種改良では、ミオスタチン遺伝子をノックアウトすることにより肉厚マダイ1)や、GABA合成を阻害する遺伝子の破壊2)によるGABA高含有トマトが知られている。微生物や細胞を利用したアミノ酸やバイオ医薬品などの工業的生産において、宿主細胞の成長と目的物質の生産がトレードオフの関係では、ゲノム編集技術とシステム生物学、合成生物学、代謝工学の知見を生かして競合する代謝回路を破壊する方法3)により課題の解決が図られている。
Cas9の他、CRISPR-Casシステムには様々な種類があり、本稿で紹介するCRISPR-Cas3 (Cas3) もその一つである。Cas3は、Cas9にない特徴があり、Cas9と同様に様々な産業分野に適用な技術として期待されている。本稿では、Cas3の特徴とともに、産業応用への可能性について紹介する。
2.Cas3の原理と特徴
2-1 ゲノム編集の原理
CRISPRは原核生物の獲得免疫系機構であり、構成要素によりクラス1とクラス2に分類される。さらに、クラス1はType I, III, IVに、クラス2はType II、V、VIに細分類される。Cas9はクラス2 Type IIに、Cas3はクラス1 Type Iに属している。クラス1 Type IはさらにAからFの6つのサブタイプに区分される。Type I-Eには大腸菌も属しており、Cas3システムを保持している。
Type I-Eは、ヌクレアーゼ活性とヘリカーゼ活性を有するCas3タンパク質と、CRISPR-RNA (crRNA) を内在するCascade (CRISPR-associated complex for antiviral defense (Cascade) と呼ばれるタンパク質からなる複合体 (リボヌクレオプロテイン:RNP) の2つのユニットから構成されている (図1)。Cas3によるゲノム編集は、Cascadeがターゲットとなる配列を認識するとCas3タンパク質がリクルートされ、ゲノム編集に係る反応が開始される。crRNAが標的配列認識するための相補的配列は32塩基分である。CRISPR-Casシステムの活性は、標的配列直上に位置するプロトスペーサー隣接モデル (Protospacer Adjacent Motif:PAM) に依存する。PAMは3塩基から構成され、Cas3では6種類のPAM (AAG、TAG、AAC、GAG、AGG、ATG) で強い活性を発揮する。最も一般的なType II CRISPRシステムでは化膿性レンサ球菌由来のCas9ヌクレアーゼが使用され、1種類のPAM (NGG) を認識する。Cas3の標的配列認識はCas9と同様に塩基の相補性を利用しているため、crRNA設計の自由度は非常に高く、自由自在に標的配列を選定することが可能である。
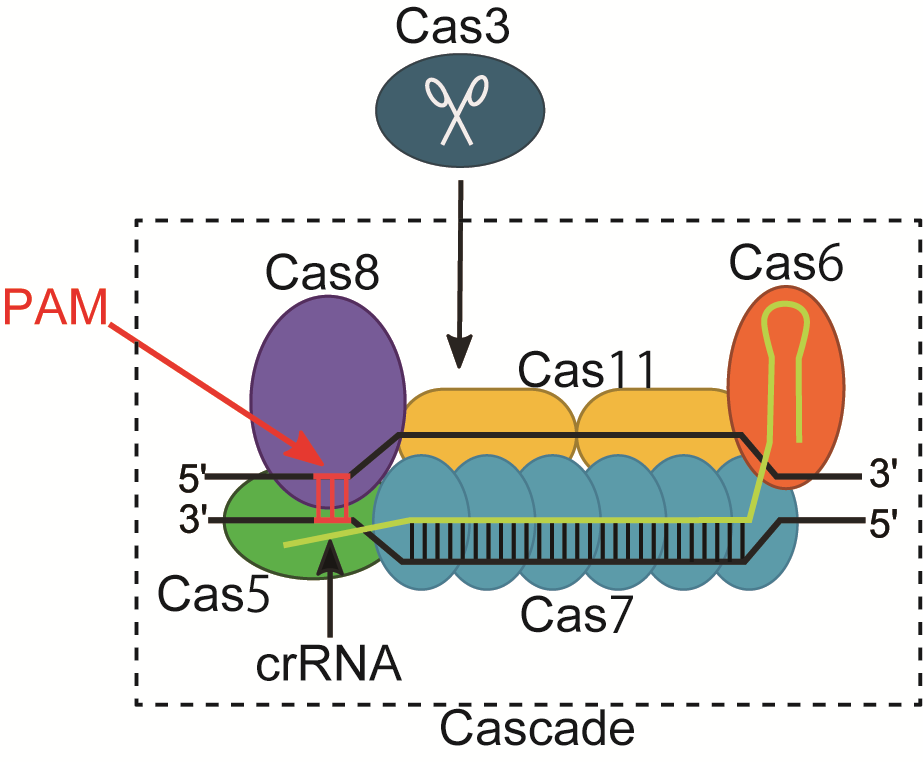
図1 Type I-E Cas3の構成
近年、クラス1Type Iに属するCRISPRのゲノム編集への応用例が報告され始め4-6)、森坂らはType I-E Cas3の真核細胞内でのゲノム編集活性や機能をはじめ、様々な特徴を明らかにしている4)。次項では当該報告から、Cas3のゲノム編集の特徴を解説する。
2-2 ゲノム編集の特徴
2-2-1 大規模欠失
Cas3の大きな特徴は、大規模な欠失領域とオフターゲット効果の低さである。Cas9の点による二本鎖切断に対し、Cas3は大きく削るような二本鎖切断 (大規模欠失) を可能とする。ヒト細胞のEMX1およびCCR5遺伝子のノックアウトによるゲノム編集により、PAM配列から上流に数百bp~数十kbpの領域欠失が報告されている (図2)4)。この切断活性は、Cas3タンパク質がもつヌクレアーゼ活性とヘリカーゼ活性によるもので、二本鎖をほどく反応と標的配列特異なDNAの分解、非特異的に1本鎖を分解するコラテラル活性の組み合わせによるものと考えられている7)。ノックインも可能であり、ドナー遺伝子のゲノムへの挿入も報告されている4)。
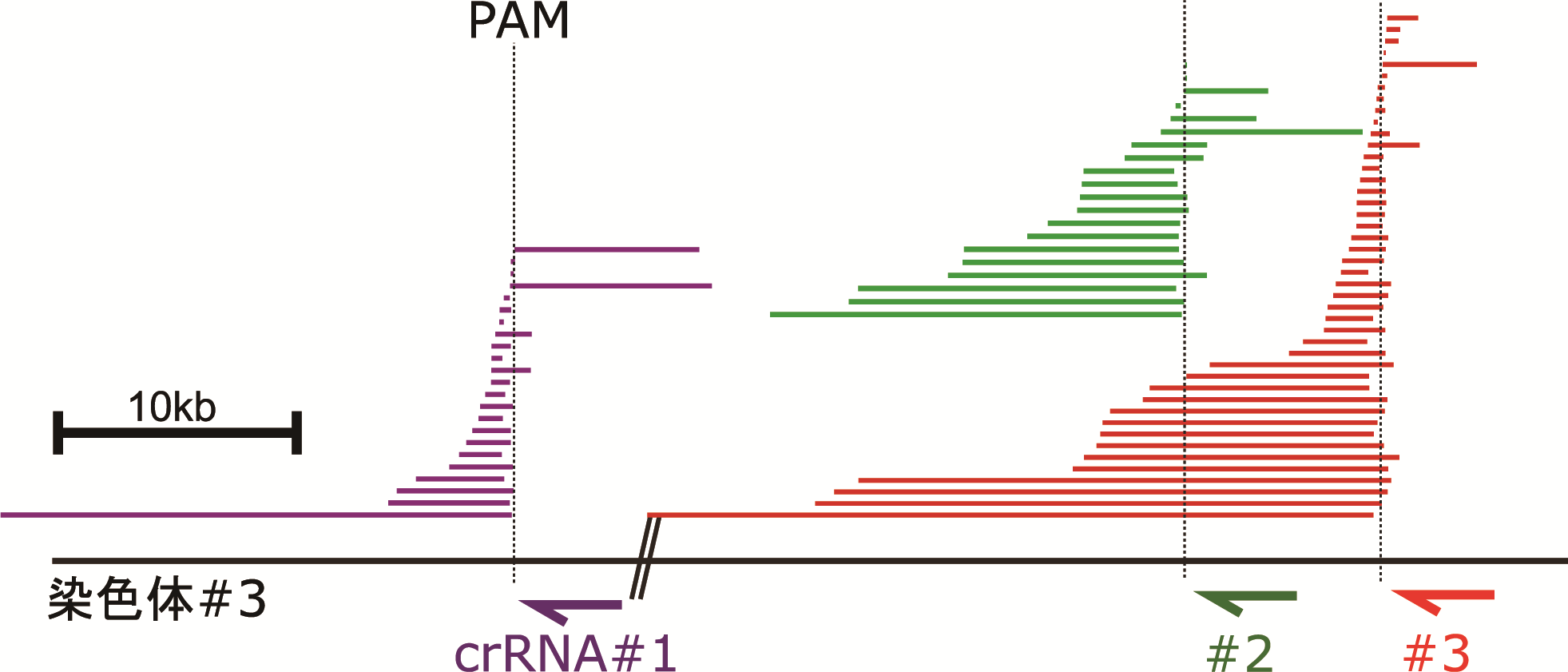
![]()
Cas3の特徴である大規模欠失能は、遺伝子のノックアウトや染色体の欠失への応用が期待されている。Cas9による遺伝子のノックアウトは開始コドンの削除や読み取り枠のフレームシフトによって引き起こされる。フレームシフトによるノックアウトは標的遺伝子の発現レベルが低下するもののタンパク質が発現する現象が報告されており8)、不完全なタンパク質発現9)による生体に及ぼす影響が懸念される。Cas3の大規模な欠失能は、標的遺伝子をコードするエクソン全体の除去、遺伝子の塩基配列のリピート領域または特定の機能部位が判別困難なノンコーディング領域等の除去することを可能とする効率的なノックアウト方法として力強い技術となる。
2-2-2 オフターゲット効果
オフターゲット効果とは、標的配列以外の配列が切断されることである。Cas9ではオフターゲット効果が様々な細胞や標的遺伝子で確認されており、実用化に向けた安全性の課題として取り上げられている10)。一方、ヒト細胞に対するCas9を用いたゲノム編集で観察されたオフターゲット効果が、Cas3では観察されなかったことが報告されている4)。Cas3のcrRNAで標的配列を認識する32塩基のうち27塩基分が効果的に標的配列を認識できることが明らかにされており、Cas9の20塩基分に比べ7塩基分長い。この認識塩基数の違いが標的配列への高い特異性に寄与していると考えられている。Cas3は高い特異性を有する、より安全性の高いゲノム編集技術であると考えられる。
2-3 診断技術への応用
Cas3の診断技術への応用も期待されている。海外ではSHERLOCK11)やDETECTR12)と呼ばれるCRISPR診断システムが存在し、それぞれCas13aとCas12aタンパク質の核酸認識機能を活用して標的配列を有する核酸の検出を可能としている。吉見らはCas3が一本鎖DNAを切断する活性 (コラテラル活性) を有することを発見し、新たな核酸検出技術であるCas3-Operated Nucleic Acid detectioN (CONAN) 法を開発した (図3)13)。CONAN法はウイルスや細菌などの核酸を迅速、簡便かつ高感度で検出することができ、一塩基の違いを識別可能な非常に高い特異性を有する反応系である。
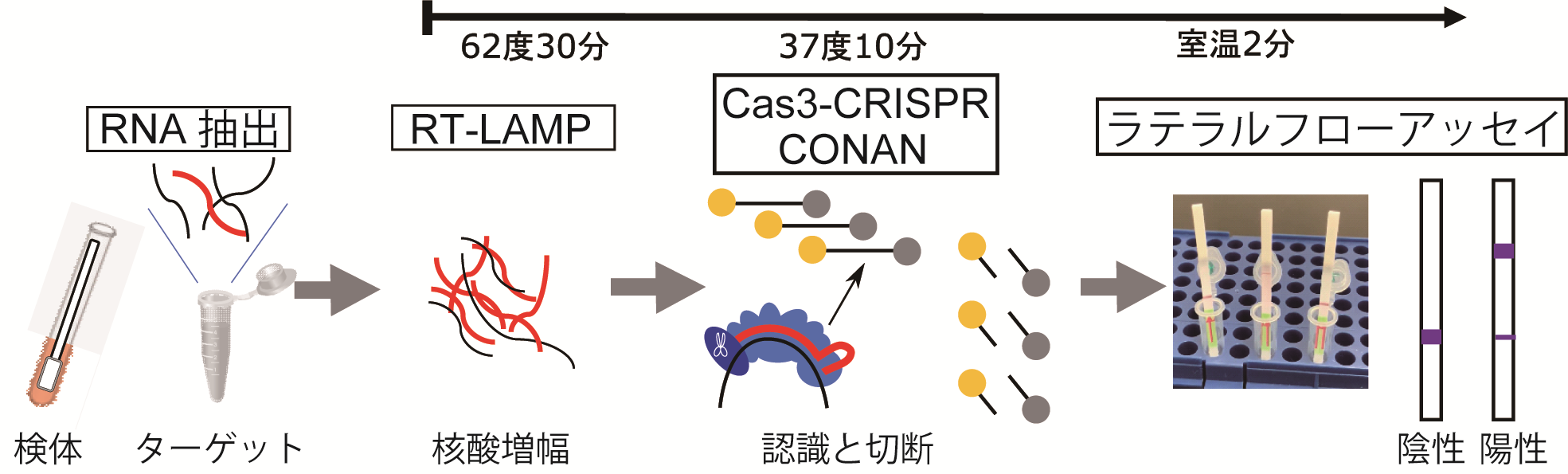

CONAN法は、新型コロナウイルス感染症の診断技術として実用化研究も進められており、一塩基の違いを識別できることから、次々に変異するウイルスの識別も可能である。感染症の診断技術に留まることなく、農作物や畜産物などの品種判別や品質管理を目的とした検査技術としての応用も期待されている。
3.Cas3の社会実装
著者が所属するC4U株式会社 (当社) は、新規ゲノム編集技術であるCas3システムの社会実装を目指し、医療分野・ヘルスケア (根本治療)、農畜水産業 (品種改良)、工業 (製造プロセス改良)、環境・エネルギー (バイオ燃料) など、スマートセルによる新産業の創生に向けた基盤技術開発を進めている。
当社では、医療・ヘルスケア分野での技術開発に注力している。ゲノム編集した細胞によるex vivo遺伝子治療、ウイルスベクターやナノ粒子を利用してCas3システムを直接体内に投与するin vivo遺伝子治療、ゲノム編集により免疫拒絶反応を低減したユニバーサル細胞の作製技術の開発が進んでいる。また、迅速、簡便、高感度を実現するCRISPR診断技術であるCONAN法を用いた新型コロナウイルスの核酸検出検査の実用化は実現間近である。
産業分野では、ゲノム編集を用いた鶏卵バイオリアクターによるタンパク質生産が実用化され、食品検査技術確立に向けたCONAN法の応用研究なども開始されている。その他、様々な産業分野での共同研究により、社会実装に向けた活動が進展している。
当社では、標準的な治療法が存在しない単一遺伝子疾患に対する根本的な治療法や医療・ヘルスケア分野での画期的な技術開発を目指し、産業分野や社会の課題を解決するために基盤技術を更に進化させ、社会実装に向けた研究開発を進めていく。
4.おわりに
Cas3はCas9と異なるゲノム編集技術の特徴を有するとともに、事業的にも有利な点があると考えられている。Cas9の基本特許を巡っては、ブロード研究所とカリフォルニア大学の特許係争が継続しており、解決の道筋は未だ不透明な状況である。一方、Cas3はCas9の基本特許から完全に独立して権利化されており、Freedom to Operate (FTO) が確保されている。事業的視点からCas3はCas9と比較して、大きな利点を有していると考えられる。
当社の社名であるC4Uは、多くの研究者をはじめ産業界で広くCas3を活用いただきたいという発明者の思いから、”CRISPR for You”を由来に命名された。Cas3に興味を覚える際は、是非ともお声がけいただきたい。国産ゲノム編集技術CRISPR-Cas3の一日も早い社会実装に向け、力強いご支援をいただきたい。CRISPR for You!
謝辞
本稿執筆に際し、東京大学医科学研究所の真下知士教授および吉見一人講師より多大なご支援を頂きました。心より感謝申し上げます。
文献
1) Kishimoto, K., Washio, Y., Yoshiura, Y., Toyoda, A., Ueno, T., Fukuyama, H., Kato, K., Kinoshita, M.: Aquaculture, 495, 415 (2018).
2) Nonaka, S., Arai, C., Takayama, M., Matsukura, C., Ezura, H.: Sci. Rep., 7, 7057 (2017).
3) Zhao, D., Zhu, X., Zhou, H., Sun, N., Wang, T., Bi, C., Zhang, X.: Metabo. Eng., 63, 148 (2021).
4) Morisaka, H., Yoshimi, K., Okuzaki, Y., Gee, P., Kunihiro, Y., Sonpho, E., Xu, H., Sasakawa, N., Naito, Y., Nakada, S., Yamamoto, T., Sano, S., Hotta, A., Takeda, J., Mashimo, T.: Nat. Commun., 10, 5302 (2019).
5) Osakabe, K., Wada, N., Murakami, E., Miyashita, N., Osakabe, Y.: Nucleic Acids Res., 49, 11, 6347 (2021).
6) Tan, R., Krueger, R. K., Gramelspacher, M. J., Zhou, X., Xiao, Y., Ke, A., Hou, Z., Zhang, Y.: Mol. Cell (2022).
7) Yoshimi, K., Takeshita, K., Kodera, N., Shibumura, S., Yamauchi, Y., Omatsu, M., Kunihiro, Y., Yamamoto, M., Mashimo, T.: bioRxiv, 2021.07.18.452824 (2021).
8) Smits, A. H., Ziebell, F., Joberty, G., Zinn, N., Mueller, W. F., Clauder-Münster, S., Eberhard, D., Fälth Savitski, M., Grandi, P., Jakob, P., Michon, A.-M., Sun, H., Tessmer, K., Bürckstümmer, T., Bantscheff, M., Steinmetz, L. M., Drewes, G., Huber, W.: Nat. Methods, 16, 1087 (2019).
9) Tuladhar, R., Yeu, Y., Tyler Piazza, J., Tan, Z., Rene Clemenceau, J., Wu, X., Barrett, Q., Herbert, J., Mathews, D. H., Kim, J., Hyun Hwang, T., Lum, L.: Nat. Commun., 10, 4056 (2019).
10) Zhang, X.-H., Tee, L. Y., Wang, X.-G., Huang, Q.-S., Yang, S.-H.: Mol. Ther. Nucleic Acids, 4, e264 (2015).
11) Kellner, M. J., Koob, J. G., Gootenberg, J. S., Abudayyeh, O. O., Zhang, F.: Nat. Protoc., 14, 2986 (2019).
12) Chen, J. S., Ma, E., Harrington, L. B., Da Costa, M., Tian, X., Palefsky, J. M., Doudna, J. A.: Science, 360, 436 (2018).
13) Yoshimi, K., Takeshita, K., Yamayoshi, S., Shibumura, S., Yamauchi, Y., Yamamoto, M., Yotsuyanagi, H., Kawaoka, Y., Mashimo, T.: iScience, 103830 (2022).