【トピックス】
微生物資源の活用に向けたノンターゲットメタボロミクス
岡橋伸幸
阪大院・情報
1.はじめに
微生物は高等生物が持たない特有の代謝経路を有し、希少化合物や新規化学反応を触媒する酵素などを見出す資源として活用が期待されている1-3)。本邦でも、タクロリムス、イベルメクチンなどの生理活性物質が微生物から発見され、医薬品として利用されてきた。持続可能な社会の実現が求められる昨今、健康増進、農地改善、排水処理、エネルギーなどの社会課題の改善に向けて、微生物の代謝能力の活用が一層求められている。同時に、様々な難培養微生物の単離技術の開発4)や、それらを集積するバイオバンクなどの活動も活発化しており5)、単離できる微生物資源は増加している。このような状況で、次に立ち現れてくる課題は、蓄積された微生物の中から希少な代謝産物を産生する菌株をいかにスクリーニングするかである。どのような活用が期待できるか見当のつかない新規微生物に対して、その有用性を見極めるのは非常に難しい。このように事前の仮説がない場合、一通りの代謝産物を網羅的に解析するノンターゲットメタボロミクスが有効である6)。本稿では、ノンターゲットメタボロミクスを取り巻く現状と、未知代謝物を効率よく見出す情報解析技術を解説する。
2.ノンターゲットメタボロミクス
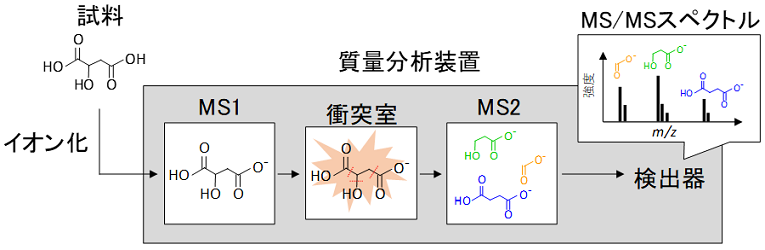
代謝物の総体を計測する学問体系をメタボロミクスと呼ぶ。メタボロミクスでは、感度良く多成分の一斉分析が可能な質量分析法 (mass spectrometry, MS) が汎用される。クロマトグラフィーと組み合わせて利用されることが多く、カラムにて物性の違いで分離した成分を順次質量分析装置に導入し、イオン化することで質量電荷比(m/z)を計測する (図1)。近年では質量分析装置の精密質量精度が格段に向上し、計測対象の分子式をある程度絞り込むことが可能となっている6)。ただし、対象物質の構造 (化学結合) を解析する核磁気共鳴法とは異なり、質量分析装置が計測するのはイオンの質量である。対象物質の構造を推定するためには、計測したイオンを質量分析装置内で開裂させ、その断片イオンの質量電荷比をスキャンして得たMS/MSスペクトル (より正確にはプロダクトイオンスペクトル) を取得する (図1)。この時の断片イオンのパターンは元の構造に特有なので、パズルのピースのように断片イオンを組み立てることで元の構造を推定することができる。この開裂前後のイオンを100 msec程度で高速スキャンすることで、1ランの分析から約5000 ~ 10000にものぼるMS/MSスペクトルを得ることができる。このようにあらかじめ標的を定めることなく、網羅的な解析を行う手法を、従来の標的限定型のターゲットメタボロミクスと対比して、ノンターゲットメタボロミクスと呼ぶ。この方法を駆使することで、想定していない新たな代謝物の発見が期待できる。

3.代謝物の同定
ノンターゲットメタボロミクスで最も難しいのは代謝物の同定である6,7)。同定とは、得られたMS/MSスペクトルを標準品などのMS/MSスペクトルと比較し、その同一性を確認することでMS/MSスペクトルと代謝物を紐づける作業のことである。一般に標準品が入手可能な代謝物は、それらのMS/MSスペクトルがMassBank8)などの公共データベースに収められており、それと比較することで代謝物の同定が可能である。さらに、実測MS/MSスペクトルをもとに、代謝物の炭素数や不飽和度などのありえるバリエーションを拡張したin silico MS/MSスペクトルライブラリの構築が行われ、ヒト、マウス、植物などのサンプルから8000分子種ほどが同定できるようになった9)。同定作業自体も、各分子に特徴的なスペクトルの有無をスコアリングすることで半自動で実施できるようになっており9)、従来手間と時間のかかっていたノンターゲット解析の代謝物同定は飛躍的に進歩している。それでも、現状1ランの分析で検出できるイオンのうち同定できるのは、ヒト血漿のような成分のよく調べられている検体でも2割程度にとどまる10)。微生物など哺乳類とは異なる代謝経路を有する生物種のMS/MSスペクトルライブラリは整備が十分でなく同定率はさらに低下する11)。一説には、環境中の化合物のうち既存のデータベース情報で同定できるのは1.8%とまで言われている12)。ゆえに、菌株のノンターゲットメタボロームデータを取得しても、その多くが読み解けないデータになってしまい、活用が難しいというのが現状である。
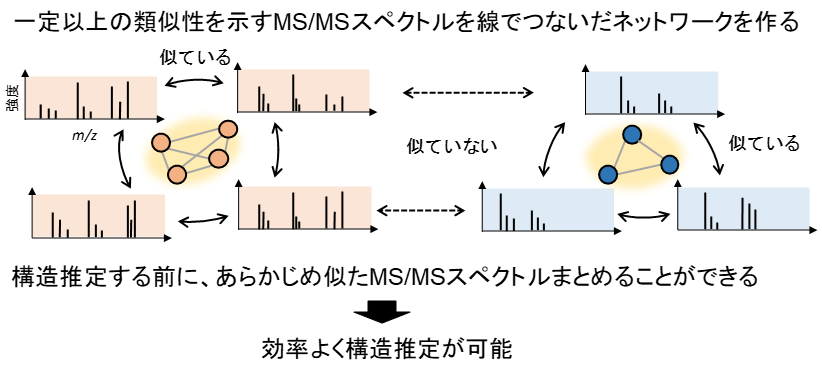
しかし、自動では同定できなかったMS/MSスペクトルの中にこそ、まだ人類の知らない新たな化合物が眠っている可能性が高く、膨大な未知データから解読する価値のあるデータを簡便に見出す方法論が必要である。この要求に応えるべく、近年、天然物化学の分野で、Molecular spectrum networkingという情報科学的な手法が開発された13)。この方法は、近しい構造の物質は互いに似たMS/MSスペクトルを示すという性質を利用し、MS/MSスペクトルの類似度に基づく分子構造ネットワークを作成する技術である (図2)。これを使えば、未同定なりにも互いに似た構造を持つと推測されるMS/MSスペクトルを簡便に見出すことができる。

4.腸内細菌叢の解析事例
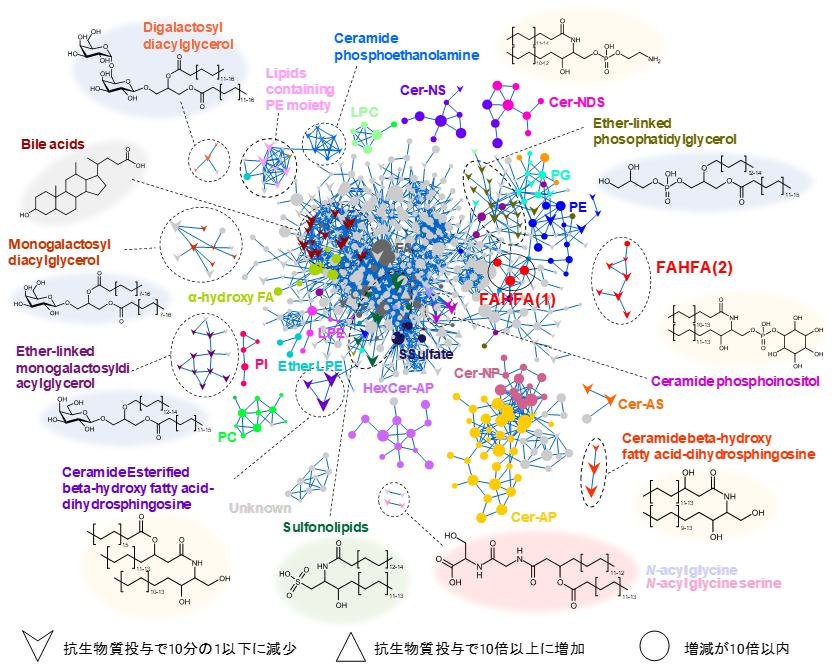
ここでは、ノンターゲットメタボロミクスとMolecular spectrum networkingを組み合わせて、マウス腸内に共生する細菌叢の脂質解析に取り組んだ、我々の最近の研究成果を紹介したい11)。腸内細菌の脂質は、宿主にないユニークな構造を有し、宿主の疾患や生体恒常性の制御に関わることから、生理活性物質の宝庫である。例えば、菌体表層を覆い、宿主と接する脂質膜成分は、宿主のパターン認識受容体を介して自然免疫応答を引き起こすことが知られており14)、宿主免疫機能の活性化やワクチンの効果を強めるアジュバントとしての活用が期待される。そこで、それらの全体像をとらえるべく、野生型マウスの糞便の総脂質画分を抽出し、液体クロマトグラフィー-四重極飛行時間型質量分析装置 (LC-QTOF/MS) を用いてノンターゲットメタボロミクスを実施した。10818個のイオンが観測され、そのうち既存のライブラリで同定できたのは全イオンの内の7.9% (855種) と、多くの未同定化合物が存在することが分かった。これらの未同定分子の構造を効率よく推定するべく、Molecular spectrum networkingを適用し、MS/MSスペクトルネットワークを作成した (図3)。図3の各点が計測されたイオンを表し、互いに似た構造を持つと推定されるイオン同士が線でつながれたクラスターを形成している。ここからさらに腸内細菌によって産生される分子を絞り込むために、抗生物質カクテルを投与したマウスの糞便を同様に分析した。抗生物質投与群で減少したイオンは腸内細菌によって産生されていた可能性を示唆する。そのようなイオンを図3では下向きの矢印で表記した。これより、図3の破線で囲ったクラスターに属する代謝物は、抗生物質投与で量が減少したことから腸内細菌由来の代謝物であることが強く示唆され、優先的に構造解析するべき対象であると絞り込むことができた。これらのクラスターに含まれるMS/MSスペクトルから構造を推定したところ、エーテル結合をもつグリセロ脂質、極性頭部にホスホエタノールアミンやホスホイノシトールなどの修飾を有するセラミド、脂肪酸のβ位にヒドロキシ基があるセラミド、炭素―硫黄原子が直接結合したスルホノリピド、グリシンやセリンで抱合された脂肪酸代謝物など、哺乳類には見られない多様な構造の脂質分子群の存在が明らかとなった。

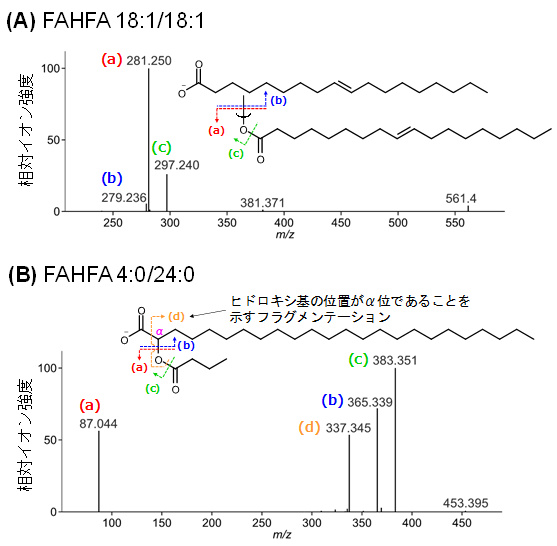
さらに興味深いことに、互いに類似の構造を有しながら、2つのクラスターで抗生物質投与による量的変動の傾向が異なる脂質Fatty Acid esters of Hydroxyl Fatty Acid (FAHFA)が見出された (図3、FAHFA(1)と(2))。FAHFAは脂肪酸とヒドロキシ脂肪酸がエステル結合した脂質の総称であり、先行研究では宿主によって長鎖脂肪酸同士がエステル結合した分子が産生されると報告されていた15)。この既報と一致して、FAHFA(1)は長鎖同士のエステル結合分子と推定され (図4A)、抗生物質投与群での顕著な現象は見られなかった。一方、FAHFA(2)は、抗生物質投与で20分の1以下に減少しており、腸内細菌叢によって産生されている可能性が強く示唆され。そこで、MS/MSスペクトルから構造を読み解くと、FAHFA(2) は極長鎖ヒドロキシ脂肪酸 (炭素数20 ~ 24) に短鎖脂肪酸 (炭素数2 ~ 5) がエステル結合した構造であると推定された (図4B)。さらに、FAHFA(2)のMS/MSスペクトルからは、FAHFA(1)にはみられない、ヒドロキシ基の位置が極長鎖脂肪酸のα位であることを示す特徴的な断片イオンが観測された (図4B)。実際にFAHFA(2)に相当する化合物を有機合成し、LC-QTOF/MS解析で同一の保持時間およびMS/MSスペクトルを示したことから推定構造が正しいことを実証した。このユニークな構造の脂質は既存データベースに収載がなかったことから、我々はこの新規脂質群をAcyl Alpha-Hydroxy Fatty Acid (AAHFA) と名付けた11)。このようにMolecular spectrum networkingは、膨大なノンターゲットメタボロームデータから似通った構造を持つイオン群を分類することができ、効率よく新たな分子を見出すのに威力を発揮する。本解析に必要な装置のセッティングや前処理法、Molecular spectrum networkingの実施方法などは筆者らの原著論文中で詳しく解説しているので、興味があればぜひトライしていただきたい16)。

5.おわりに
本稿では、ノンターゲットメタボロミクスにおける代謝物同定の現状と、未知代謝物を効率よく見出すための情報科学的な解析法を概説した。ノンターゲットメタボロミクスは未知代謝物をとらえるポテンシャルを有しており、多成分の同時計測が可能であることから菌株を特徴づけるための一次スクリーニングとして威力を発揮する。Molecular spectrum networkingは植物、藻類、微生物などの産生する生理活性物質の探索への適用も進んでおり17)、脂質以外の多様な分子に利用可能である。今後は、このような大規模メタボロームデータの蓄積が加速されると見込まれ、大規模データを読み解く技術の開発が一層重要になる。また、メタボロームデータとゲノムデータやプロテオームデータなど他の階層のオミクスデータの統合ができれば、新規代謝物の産生酵素遺伝子の特定も可能になると期待される18)。これに向けて、筆者は、菌株のゲノム情報と代謝物情報を高度に組み合わせて解析する方法の開発にも着手している。微生物資源の活用で持続可能な社会の実現をリードするべく、これからも計測と情報解析という立場から新たな発見に貢献できれば幸いである。また、興味深い代謝物を産生する可能性のある菌株を持っているが、化合物が絞り込めていないといった方から共同研究のお誘いをいただけると技術開発を生業とする者として嬉しい限りである。
謝辞
本稿で紹介した研究11)は前職の理化学研究所生命医科学研究センターの有田誠先生、池田和貴先生、津川裕司先生のご指導の下、当時大学院生の安田柊博士が中心となって行ったものである。この場を借りて厚く御礼申し上げます。
文献
1) Pham, J. V., Yilma, M. A., Feliz, A., Majid, M. T., Maffetone, N., Walker, J. R., Kim, E., Cho, H. J., Reynolds, J. M., Song, M. C., Park, S. R., Yoon, Y. J.: Front. Microbiol., 10, 1 (2019).
2) Harvey, A. L., Edrada-Ebel, R., Quinn, R. J.: Nat. Rev. Drug Discov., 14, 111 (2015).
3) Challinor, V. L., Bode, H. B.: Ann. N. Y. Acad. Sci., 1354, 82 (2015).
4) Bodor, A., Bounedjoum, N., Vincze, G. E., Erdeiné Kis, Á., Laczi, K., Bende, G., Szilágyi, Á., Kovács, T., Perei, K., Rákhely, G.: Rev. Environ. Sci. Biotechnol., 19, 1 (2020).
5) 永井利郎、一木 (植原) 珠樹、澤田宏之、佐藤豊三、青木孝之、山﨑福容、竹谷 勝、埋橋志穂美、廣岡裕吏、富岡啓介: 日本食品科学工学会誌, 60, 257 (2013).
6) Schrimpe-Rutledge, A. C., Codreanu, S. G., Sherrod, S. D., McLean, J. A.: J. Am. Soc. Mass Spectrom., 27, 1897 (2016).
7) Sindelar, M., Patti, G. J.: J. Am. Chem. Soc., 142, 9097 (2020).
8) Horai, H., Arita, M., Kanaya, S., Nihei, Y., Ikeda, T., Suwa, K., Ojima, Y., Tanaka, K., Tanaka, S., Aoshima, K., Oda, Y., Kakazu, Y., Kusano, M., Tohge, T., Matsuda, F., Sawada, Y., Hirai, M. Y., Nakanishi, H., Ikeda, K., Akimoto, N., Maoka, T., Takahashi, H., Ara, T., Sakurai, N., Suzuki, H., Shibata, D., Neumann, S., Iida, T., Tanaka, K., Funatsu, K., Matsuura, F., Soga, T., Taguchi, R., Saito, K., Nishioka, T.: J. Mass Spectrom., 45, 703 (2010).
9) Tsugawa, H., Ikeda, K., Takahashi, M., Satoh, A., Mori, Y., Uchino, H., Okahashi, N., Yamada, Y., Tada, I., Bonini, P., Higashi, Y., Okazaki, Y., Zhou, Z., Zhu, Z.-J., Koelmel, J., Cajka, T., Fiehn, O., Saito, K., Arita, M., Arita, M.: Nat. Biotechnol., 38, 1 (2020).
10) Riquelme, G., Zabalegui, N., Marchi, P., Jones, C. M., Monge, M. E.: Metabolites, 10, 1 (2020).
11) Yasuda, S., Okahashi, N., Tsugawa, H., Ogata, Y., Ikeda, K., Suda, W., Arai, H., Hattori, M., Arita, M.: iScience, 23, 1 (2020).
12) Da Silva, R. R., Dorrestein, P. C., Quinn, R. A.: Proc. Natl. Acad. Sci. USA, 112, 12549 (2015).
13) Aron, A. T., Gentry, E. C., McPhail, K. L., Nothias, L. F., Nothias-Esposito, M., Bouslimani, A., Petras, D., Gauglitz, J. M., Sikora, N., Vargas, F., van der Hooft, J. J. J., Ernst, M., Kang, K. B., Aceves, C. M., Caraballo-Rodríguez, A. M., Koester, I., Weldon, K. C., Bertrand, S., Roullier, C., Sun, K., Tehan, R. M., Boya P, C. A., Christian, M. H., Gutiérrez, M., Ulloa, A. M., Tejeda Mora, J. A., Mojica-Flores, R., Lakey-Beitia, J., Vásquez-Chaves, V., Zhang, Y., Calderón, A. I., Tayler, N., Keyzers, R. A., Tugizimana, F., Ndlovu, N., Aksenov, A. A., Jarmusch, A. K., Schmid, R., Truman, A. W., Bandeira, N., Wang, M., Dorrestein, P. C.: Nat. Protoc., 15, 1954 (2020).
14) Miller, S. I., Ernst, R. K., Bader, M. W.: Nat. Rev. Microbiol., 3, 36 (2005).
15) Yore, M. M., Syed, I., Moraes-Vieira, P. M., Zhang, T., Herman, M. A., Homan, E. A., Patel, R. T., Lee, J., Chen, S., Peroni, O. D., Dhaneshwar, A. S., Hammarstedt, A., Smith, U., McGraw, T. E., Saghatelian, A., Kahn, B. B.: Cell, 159, 318 (2014).
16) Okahashi, N., Ueda, M., Yasuda, S., Tsugawa, H., Arita, M.: STAR Protoc., 2, 1 (2021).
17) Fox Ramos, A. E., Evanno, L., Poupon, E., Champy, P., Beniddir, M. A.: Nat. Prod. Rep., 36, 7 (2019).
18) Panter, F., Bader, C. D., Müller, R.: Chem. Sci., 12, 5994 (2021).