【トピックス】
バクテリア由来1-amino-2-methylcyclopropanecarboxylic acid生合成に関わるSAM C - メチル化酵素
丸山千登勢、濱野吉十
福井県大院・生物資源
1.はじめに
バクテリアや糸状菌によって生産される薬理学的に重要なペプチド系抗生物質の多くは、非リボソーム型ペプチド合成酵素 (NRPS) と呼ばれる巨大なタンパク質複合体によって、非リボソーム的に合成される1-3)。非リボソーム型ペプチドは、その化学構造内に非タンパク性アミノ酸 (NPA) を含むものが多く4)、それは、構成するアミノ酸を決定するadenylationドメインが、基質として標準的なタンパク性アミノ酸だけでなく、NPAも認識できることに由来する5)。このNPAを取込むという性質は、ペプチド系天然物の構造多様性に大きく貢献しており、またペプチダーゼやプロテアーゼのようなアミ
ド加水分解酵素による分解を受けにくくし、結果として、ペプチド系化合物の生化学的安定性を向上することが知られている。さらにNPA含有ペプチドは、NPAの化学構造と物性に起因するユニークな生理活性を示すことも多い。実際にvancomycinやdaptomycin、cyclosporinのような、臨床上で重要な非リボソーム型ペプチド化合物がNPAを含むペプチドであることからも、NPA含有ペプチドの有用性と新規化合物探索の重要性が感じられる。最近我々は、研究室で保有する放線菌株のゲノムマイニングによって、シクロプロパン環構造を有するNPA、1-amino-2-methylcyclopropanecarboxylic acid (MeACC、図1) を構成成分とするペプチド化合物の生合成遺伝子群を見出した。シクロプロパン環構造は、小さな環状分子の中に固有の歪みを持つことから、この骨格を有する化合物の生理活性の要因になることが知られている6,7)。最もシンプルなシクロプロパン含有NPAである1-aminocyclopropanecarboxylix acid (ACC、図1) は、植物ホルモンの一つであるethyleneの生合成前駆体としてよく知られており、生合成機構も明らかにされている。その一方で、モノメチル化されたACCアナログであるMeACCの生合成は未解明なままであった。本稿では、バクテリアにおけるシクロプロパン環生合成機構及び、そのメチル化に関わる新規radical SAMmethyltransferaseについて紹介する。


2. 二次代謝産物に含まれるシクロプロパン環含有NPAの生合成研究
最もシンプルなシクロプロパン含有NPAである1-aminocyclopropanecarboxylix acid (ACC) は、植物ホルモンの一つであるethyleneの生合成前駆体としてよく知られており、PLP依存型aminotransferase (ACCsynthase) が、SAMからACCと5′-methylthioadenosine (MTA) を生成することが判明している6,8)(図1A)。最近、微生物においても、二次代謝産物の化学構造内に存在するACCの生合成経路が解明された。その一つとして、放線菌が生産するguangnanmycinのACC生合成では、新規バクテリア型ACC synthase、GnmYが、植物由来ACC synthaseと同様に、PLP依存的な反応メカニズムによって、SAMからACCとMTAを形成することが報告された9)(図1A)。その一方で、次にシンプルなシクロプロパン含有NPAである1-amino-2-methylcyclopropanecarboxylic acid (MeACC、図1B) の生合成は未解明なままであった。norcoronamic acidとも呼ばれるMeACCは、SW-163C (図1B) とそのアナログ化合物に見出され、radical SAM proteinであるSwb7とPLP依存型aminotransferaseであるSwb6が協働してL-valineから生合成する経路が提唱された10)。しかしながら、Swb6のアミノ酸配列が、SAMからACCを生合成するGnmYに相同性を示すことから、SW-163CにおけるMeACCもまたSAMから生合成される可能性が示唆された。そこで本研究では、MeACCを化学構造内に有するQ6402A (図1B) の生合成遺伝子群について、MeACC生合成経路の解明を試みた。
2-1 MeACC生合成に関わる遺伝子群の探索
研究室で所有する放線菌ライブラリーのドラフトゲノムデータベースより、Q6402A生合成に関わることが予想される約31 kbpからなるゲノム断片を見出した (図2)。このゲノム断片をBACベクターにクローニングし11)、放線菌Streptomces lividans TK23を宿主とした異種発現実験を行ったところ、培養上清のHPLC-HR-ESIMS分析の結果、m/z 952.445[M+H]+を示す化合物を検出し、各種データベース検索の結果から、本化合物がStreptomyces sp. Q-6402によって生産される環状ペプチド化合物、Q6402A (図1B) であることが示唆された12)。本ゲノム断片に含まれる遺伝子群について機能予測を行ったところ、本遺伝子群には、SW-163C生合成において、MeACC形成に関わるradical SAM protein、Swb7と、PLP依存型aminotoransferase、Swb6のそれぞれに高い相同性を示す、orf29 (accession No. BCD33697) とorf30 (No. BCD.33698) が存在していた。またorf30は、guangnanmycin分子内のACC生合成を触媒するGnmYにも40%の相同性を示すことが判明した。そこで我々は、orf29及びorf30が、Q6402AにおけるMeACC生合成に関与していると予想し、これら遺伝子の機能解析を試みた。
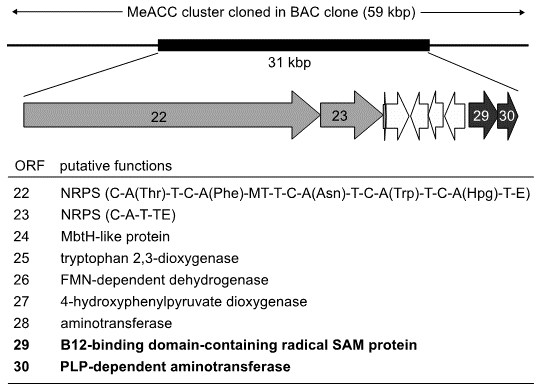
図2 Q6402A生合成遺伝子群
まずorf29とorf30遺伝子がQ6402Aの生合成に関与しているか調べるために、これらの遺伝子をBACベクター上で破壊し、異種発現用宿主S. ividans TK23に導入した。得られた形質転換体、TK23_MeACC_Δorf29及びTK23_MeACC_Δorf30を培養し、その生成物をHPLC-HR-ESI-MSで確認した。野生型遺伝子群を有するTK23_MeACCと比較して、orf29及びorf30の各破壊株はQ6402Aの生産性が完全に消失していた。このことから、orf29とorf30遺伝子が、Q6402A生合成に重要な役割を持つことが判明した。また興味深いことに、TK23_MeACC_Δorf29は、Q6402A (m/z 952.445[M+H]+) ではなく、新たな化合物 (m/z 938.428 [M+H]+) の生産を示した。この約14 Daの減少は、Q6402Aからメチル基がなくなった分子量に一致した。したがって、radical SAM methyltransferaseに相同性を示すOrf29が、Q6402Aの構造内にある2つのメチル基のうちいずれかに関与していることが予想された。加えて、NRPSをコードするorf22のモジュール内にはmethyltransferase (MT) ドメインが存在しており、このMTドメインがPhe分子のN-メチル化を触媒することが予想された。すなわち、orf29はMeACC分子内のメチル基の生合成に関与している可能性が強く示唆され、したがって、TK23_MeACC_Δorf29によって生産された化合物は、Q6402A内のMeACC分子からメチル基が無くなったdemethyl-Q6402Aであることが強く示唆された。
そこで我々は、Orf29によるメチル化反応がどのタイミングで触媒されるかを調べるために、TK23_MeACC_Δorf30を0.2% ACCを添加した培地で培養し、Q6402Aが生産されるか検証した。その結果、Q6402Aは生産されず、demethyl-Q6402Aのみが生産されることが判明した。この結果から、Orf29がACC分子、demethyl-Q6402A分子のいずれのシクロプロパン環も基質として認識しない可能性が考えられた。ACCもメチル化されないのであれば、いつOrf29によるメチル化反応が触媒されるのか、それを明らかにするために、orf29とorf30遺伝子の共発現実験を試みた。
2-2 大腸菌を宿主としたorf29及びorf30遺伝子の共発現実験
Orf29の推定アミノ酸配列からドメイン検索を行ったところ、B12結合ドメインと[4Fe-4S]結合ドメインを有することが示唆された。したがって、Orf29はmethylcobalaminをメチル基供与体とし、また活性発現には[4Fe-4S]クラスターの再構築が必須であると考えられた。そこで我々は、大腸菌の[4Fe-4S]クラスターの構築に関わるsufオペロン遺伝子を連結したプラスミド、pRKSUF017を埼玉大学、高橋康弘教授より分譲いただき13)、pKUSUF017を導入した大腸菌、EcSUFを宿主にorf29及びorf30遺伝子の共発現実験を試みた。共発現株、EcSUF_orf29_orf30を培養し、その培養上清をアミノタグAPDSで誘導体化後、HPLC-HR-ESI-MSにて分析したところ、orf29遺伝子だけを発現するEcSUF_orf29と比較して、わずかながらも共発現株特異的に、MeACCの生産が確認された。したがって、これら2つの酵素遺伝子は確かにMeACC生合成に関与することが判明した。さらに、EcSUF_orf29を0.2% ACCを添加した培地で培養した場合には、MeACCの生産は確認されず、この結果からもOrf29は、ACC分子内のシクロプロパン環をメチル化しないことが示唆された。
これまでの結果から、MeACCの生合成経路として、SAMがOrf30によってシクロプロパン化された後にOrf29によってメチル化される経路Aと、SAMがOrf29によってメチル化された後にOrf30によって環化される経路Bが考えられた (図3)。そこで次に、orf29及びorf30の詳細な機能解析ために、これら2つの組換え酵素を用いたin vitro反応を試みた。
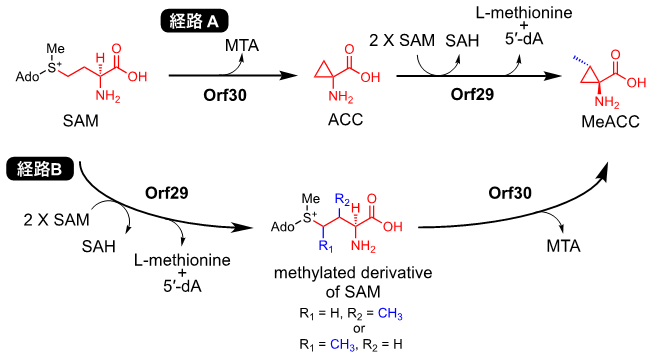
図3 MeACCの推定生合成経路
2-3 orf30組換え酵素 (rOrf30) を用いたin vitro反応
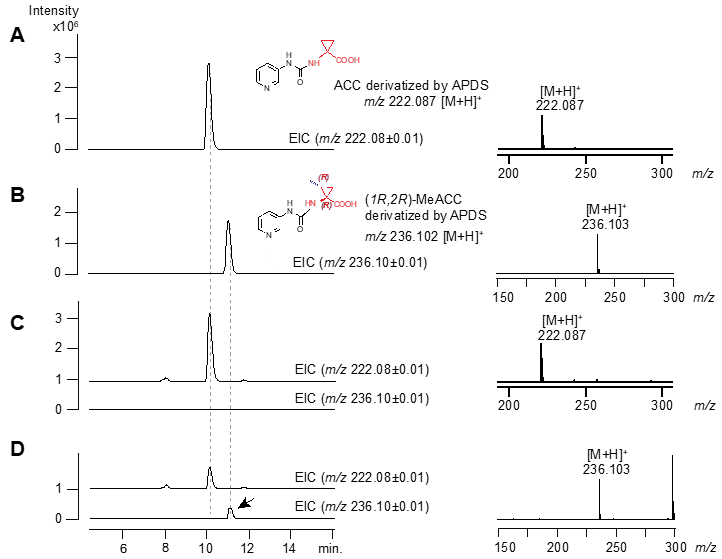
前述したようにOrf30は、バクテリア由来ACC synthaseとして報告されたGnmYに相同性 (identity 40%) を示した。したがって、Orf30もまたSAMからACCを生合成すると予想した。そこで大腸菌を宿主にC末端Hisタグ融合タンパクとしてOrf30組換え酵素 (rOrf30) を精製し、基質としてSAMを用いて酵素反応を行った。反応生成物をアミノタグAPDSにて誘導体化した後、HPLCHR-ESI-MSにて分析したところ、ACCに一致する化合物ピークが検出された (図4C)。熱失活酵素を用いた反応ではACCの生成は見られなかったこと、また反応副産物としてMTAの生成も確認されたことから、酵素反応依存的にSAMがACCへと変換されたことが判明した。このことからrOrf30はGnmYと同様の反応機構を有するバクテリア由来ACC synthaseであることを証明した。そこで次にrOrf30の酵素学的性質を調べたところ、rOrf30はSAMへの見かけのKmが1.09±0.33 mM、kcatは0.0049±0.00068 s-1であった。驚くべきことに、報告されているGnmYの見かけのKm 21.6±1.8µM、kcat0.79±0.02 s-1と比較すると、rOrf30のkcat値はGnmYよりも1,900倍も低いことが明らかになった9)。このことから、MeACC生合成において、SAMはrOrf30の本来の基質ではない可能性が示唆された。前述したように、EsSUF_orf29を0.2% ACCを含む培地で培養した場合に、MeACCは生成されず、Orf29はACCをメチル化しないことが示唆された。これらの結果から、MeACCのメチル基は、Orf30によって触媒されるシクロプロパン化ステップの前に導入される経路B (図3) と考えられ、すなわち、Orf29によってメチル化されたSAMが、Orf30の本来の基質であると予想した。そこで次に、この予想を証明するために、Orf29組換え酵素を使ったin vitro反応を試みた。

2-4 Orf29組換え酵素 (rOrf29) を用いたin vitro反応
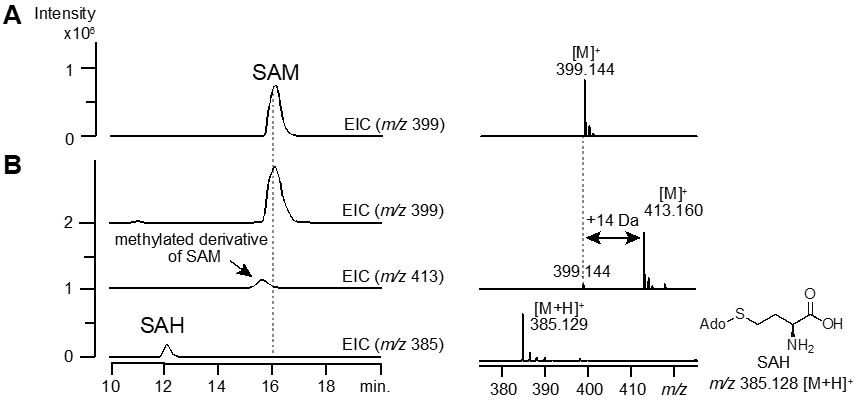
Orf29は、B12結合ドメインを有するradical SAMmethyltransferaseであることが予想されることから、活性型のOrf29組換え酵素、rOrf29を得るためには、組換え酵素発現の宿主には、[4Fe-4S]クラスターの再構築遺伝子群の他に、コバラミンの十分な取込みが必須であると考えられた。そこで、[4Fe-4S]クラスターの再構築に必要なsufオペロンを有する大腸菌宿主EcSufに、大腸菌由来コバラミン取込み遺伝子群、btuオペロンをプラスミドで導入した大腸菌EcSufBtuを構築し14,15)、rOrf29発現に用いた。pETベクターにorf29遺伝子を連結し、EcSufBtuに導入した発現用大腸菌、EcSufBtu_orf29を0.2mM FeSO4(NH4)2SO4と0.2mM L-Cysを含む最少培地で培養後、ITPG誘導によりrOrf29発現を行った。得られた菌体から、嫌気条件下 ([O2]<5 ppm) でrOrf29を精製し、UV-VIS測定によって還元型[4Fe+4S]+が再構築されたことを確認した。活性型rOrf29を用いて、SAMを基質に嫌気条件下、28℃、16時間の酵素反応を行い、反応生成物をHPLC-HR-ESI-MSで分析した。その結果、rOrf29を含まない酵素反応と比較して、SAM (m/z399.144 [M]+, calculated for 399.144 [M]+) から14 Daの分子量増加を示す化合物 (m/z 413.160 [M]+) の生産が確認され、SAMのメチル化誘導体であると考えられた (図5)。さらにこの生成物に加えて、SAHの生成も検出された (図5)。その一方で、ACCを基質にrOrf29の酵素反応を行ったところ、MeACCは生産されなかった。この結果は、これまでのorf30遺伝子破壊株を用いたACC添加培養実験 (2-2項)、及びorf29発現大腸菌を用いたACC添加培養実験 (2-3項) の結果に一致するものであり、rOrf29によるメチル化反応において、ACCを基質認識しないことをin vitroで証明することができた。さらに、rOrf29の触媒により生産されたSAMのメチル化誘導体が、rOrf30によって基質認識されるかを調べるために、rOrf29酵素反応液にrOrf30を加えて反応したところ、MeACCに一致する化合物ピークが検出された (図4-D)。この生成物の絶対立体配置を決定するために、改良Marfey法を用いて16)、評品である(1R,2R)-MeACC及び(1S,2R)-MeACCとの比較を行った結果、rOrf29及びrOrf30の連続反応による生産物は(1S,2S)-MeACCと決定された。

3.おわりに
本研究で明らかにしたorf29は、[4Fe-4S]クラスター結合ドメインとコバラミン結合ドメインを持つことから、radical SAM methyltransferaseにおけるクラスB methyltransferaseグループに分類される。クラスBmethyltransferaseグループは数多くのradical SAM酵素が含まれており、多くの二次代謝産物生合成に関わる遺伝子群から見出されている。しかしながら、数多く存在するクラスB methyltransferaseにおいて、SAMをメチル化するものは報告がない。したがって、Orf29は、SAMのL-Met分子のC-メチル化を触媒する新規radicalSAM methyltransferaseであることを明らかにした17)。rOrf29によって触媒されるSAMのメチル化部位を決定するために、rOrf29による反応生成物のNMR解析が必要であるが、反応生成物が十分に得られず、同定することはできなかった。しかしながら、rOrf29とrOrf30の連続反応によってMeACC生成が認められたことから、rOrf29がSAMのL-Met骨格をメチル化したことは明確である。今後、rOrf29のより詳細な機能解析を行いたいと考えている。
本研究で我々は、Q6402Aが有するMeACCが、Orf29の触媒によって生成されるSAMのメチル化誘導体がOrf30によってシクロプロパン環へと変換される経路B (図3) によって生合成されることを明らかにした。MeACCは非常に小さなNPAであるが、その生合成には、SAMによるラジカル転移反応と、SAMをメチル基供与体としたメチル化反応が、SAMを基質に触媒され、それが環状化合物へと変換されるという、なんとも贅沢にSAMを使ったメカニズムで生合成されることを明らかにした17)。その生合成に関わるOrf30について、GnmY、バクテリア由来PLP依存型aminotransferaseファミリーに属する酵素群、そして植物由来ACC synthaseとの系統樹解析を行ったところ、Orf30は、GnmYとも異なる、独立したクレードを形成することが判明した。このことから、Orf30はこれまでに報告されたACCのシクロプロパン環形成を触媒する酵素とは異なる、新規ACC synthaseであると考えられる。Orf30と同じクレードに属する機能未知のaminotransferase酵素遺伝子について、その近傍領域を探索したところ、これら酵素遺伝子は全て、Orf29に相同性を示すradical SAM methyltransferase遺伝子と隣り合わせていることが判明した。さらにその近傍には、NRPSをコードする遺伝子群が存在することも見出した。したがって、Orf29とOrf30のホモログ遺伝子を併せ持つ遺伝子群は、MeACCを構造内に有する化合物を生合成する可能性が示唆された。したがって、orf29及びorf30ホモログ酵素遺伝子の探索は、新たなMeACC含有ペプチド化合物の遺伝子群探索に有用であると考えられる。
謝辞
本稿で紹介した研究を行うにあたり、BACライブラリーの構築及びQ6402A生合成遺伝子群のクローニングはAIST、新家一男先生との共同研究にて行われました。また(1R,2R)-MeACC及び(1S,2R)-MeACC標準物質は、東北大学、土井隆行教授により分譲いただきました。[4Fe-4S]クラスター再構築系遺伝子群sufオペロンの発現では、埼玉大学、高橋康弘教授よりpRKSUF017をご提供いただきました。またorf29組換え酵素の発現及びin vitro反応は、東京工業大学、江口正教授、工藤史貴准教授との共同研究にて行いました。本研究を進めるにあたり、ご助言、ご協力を賜りました先生方に、この場を借りて厚く御礼申し上げます。
文献
1) Marahiel, M. A., Stachelhaus, T., Mootz, H. D.: Chem. Rev., 97, 2651 (1997).
2) Mootz, H. D., Schwarzer, D., Marahiel, M. A.: ChemBioChem., 3, 490 (2002).
3) Schwarzer, D., Finking, R., Marahiel, M. A.: Nat. Prod. Rep., 20, 275 (2003).
4) Walsh, C. T., O’Brien, R. V., Khosla, C.: Angew. Chem. Int. Ed. Engl., 52, 7098 (2013).
5) Kudo, F., Miyanaga, A., Eguchi, T.: J. Ind. Microbiol. Biotechnol., 46, 515 (2019).
6) Thibodeaux, C. J., Chang, W. C., Liu, H. W.: Chem. Rev., 112, 1681 (2012).
7) Wessjohann, L. A., Brandt, W., Thiemann, T.: Chem. Rev., 103, 1625 (2003).
8) Adams, D. O., Yang, S. F.: Proc. Natl. Acad. Sci. USA, 76, 170 (1979).
9) Xu, Z., Pan, G., Zhou, H., Shen, B.: J. Am. Chem. Soc., 140(49), 16957 (2018).
10) Watanabe, K., Hotta, K., Nakaya, M., Praseuth, A. P., Wang, C. C., Inada, D., Takahashi, K., Fukushi, E., Oguri, H., Oikawa, H.: J. Am. Chem. Soc., 131, 9347 (2009).
11) Komatsu, M., Komatsu, K., Koiwai, H., Yamada, Y., Kozone, I., Izumikawa, M., Hashimoto, J., Takagi, M., Omura, S., Shin-ya, K.: ACS. synthetic biology, 2, 384 (2013).
12) Hiramoto, M., Niwa, A., Miyake, A., Yamamoto, H., Takebayashi, Y., Nishikawa, T., Shibazaki, M., Nagai, K.: Tennen Yuki Kagobutsu Toronkai Koen Yoshishu, 35, 678 (1993).
13) Takahashi, Y., Tokumoto, U.: J. Biol. Chem., 277, 28380 (2002).
14) Lanz, N. D., Blaszczyk, A. J., McCarthy, E. L., Wang, B., Wang, R. X., Jones, B. S., Booker, S. J.: Biochemistry, 57, 1475 (2018).
15) Guzman, L. M., Belin, D., Carson, M. J., Beckwith, J.: J. Bacteriol., 177, 4121 (1995).
16) Fujii, K., Ikai, Y., Mayumi, T., Oka, H., Suzuki, M., Harada, K.: Anal. Chem., 69, 3346 (1997).
17) Maruyama, C., Chinone, Y., Sato, S., Kudo, F., Ohsawa, K., Kubota, J., Hashimoto, J., Kozone, I., Doi, T., Shin-ya, K., Eguchi, T., Hamano, Y.: biomolecules, 10(5), 775 (2020).