【トピックス】
酵母に見出したN-アセチルトランスフェラーゼMpr1の分子機能
那須野 亮、高木 博史
奈良先端大・バイオ、奈良先端大・バイオ
1.はじめに
N-アセチルトランスフェラーゼ (NAT) (EC 2.3.1.-) は、アシルトランスフェラーゼ (EC 2.3.-) の一種であり、アセチルCoA (AcCoA) をアセチル基供与体として用い、基質のアミノ基にアセチル基を転移する転移酵素である。これまでに数多くのNATが発見されており、アミノ酸やポリアミンをはじめとした低分子化合物からタンパク質などの高分子まで、様々な分子を基質とすることが報告されている一方、その生理機能も極めて多様である1)。
筆者らは、酵母Saccharomyces cerevisiaeにおけるプロリン代謝の研究過程で、プロリンの毒性アナログであるL-アゼチジン-2-カルボン酸 (AZC) をアセチル化する新規のNAT、Mpr1を発見した。またその後の解析から、Mpr1は酵母のアルギニン合成経路に寄与することが分かってきた。加えて、筆者らは、X線結晶構造解析と酵素学的解析によりMpr1の立体構造と触媒反応機構を明らかにした。さらに、立体構造情報をもとにした理論的な分子設計 (アミノ酸置換) により、熱安定性が向上したMpr1を設計・創製するとともに、これを酵母で発現させることによりアルギニン合成を強化することに成功した。本稿では、Mpr1の構造生物学的・酵素学的特徴や生理機能について、既知のNATとの類似点・相違点に触れながら概説する。また、最近明らかになってきたMpr1依存的なアルギニン合成経路の詳細について紹介する。
2.環状二級アミンをアセチル化する新規N-アセチルトランスフェラーゼMpr1
酵母S. cerevisiaeは、高等真核生物や病原性真菌のモデルとして重要であるのみならず、醸造や製パン、バイオエタノール製造を初めとした発酵産業において極めて重要な微生物である。そのため、酵母の発酵生産性を制限するようなストレス (高温、高浸透圧、高濃度エタノールなど) に対する酵母の応答を理解することは、基礎・応用の両面で重要である2)。筆者らは、植物や細菌が浸透圧ストレスに応答してプロリンを蓄積することに着目し3)、製パン関連ストレスである冷凍ストレスにおけるプロリンの効果を解析したところ、冷凍ストレスに伴う酵母の生存率低下を抑えることを見出した。そこで、酵母におけるプロリンの生理機能をさらに解析するため、プロリンの毒性アナログであるAZCを用いたスクリーニングにより、プロリンの細胞内含量が増加した酵母の取得を試みた。AZCは翻訳の際にプロリンと競合して新生タンパク質に取り込まれ、フォールディング異常のタンパク質を細胞内に生成することで毒性を発揮すると考えられており4,5)、細胞内プロリン濃度が上昇するとAZCの新生タンパク質への取り込みが競合的に抑制され、AZC毒性に耐性を示すと期待された。
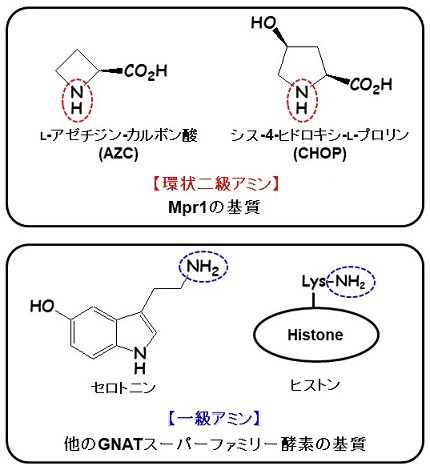
スクリーニングの結果、予想通りプロリン蓄積株を取得するとともに、新たにS. cerevisiae ∑1278b株から、AZCに耐性を示す遺伝子MPR1 (sigma 1278b gene for L-proline analogue resistance) を見出した6)。興味深いことに、MPR1の発現は細胞にAZC耐性を付与するものの、細胞内プロリン含量を増加させなかった。MPR1の翻訳産物 (Mpr1) は全長229残基のタンパク質であり、そのアミノ酸配列からNATと考えられた。そこで、大腸菌で発現した組換えMpr1を精製し、解析したところ、Mpr1はAZCに対する明確なアセチル化活性を示した7)。以上のことから、Mpr1はAZCをアセチル化することで新生タンパク質への取り込みを抑制し、細胞にAZC耐性を付与すると考えられた。MPR1は∑1278b株ゲノム上に2コピー (MPR1, MPR2) 存在し、両遺伝子間にはアミノ酸置換を伴う点変異が一ヵ所あるが (Mpr1:Gly85, Mpr2:Glu85)、これまでに機能的な違いは観察されていない6)。アミノ酸配列から、Mpr1はGcn5関連 N-アセチルトランスフェラーゼ (GNAT) スーパーファミリーに属すると考えられた6)。GNATスーパーファミリーに属する酵素には、低分子化合物からタンパク質まで、様々な基質をアセチル化する酵素が存在するが8)、これらはすべて一級アミンである。一方、Mpr1の基質であるAZCは環状の二級アミンであり、その後の解析により見出された別の基質であるシス-4-ヒドロキシ-L-プロリン (CHOP) も環状二級アミンである9)。環状二級アミンを基質とするNATはこれまでに全く報告されていないことから、Mpr1は極めて特殊な基質特異性を示す新規のNATであると考えられた (図1)。
![]()
3.Mpr1のユニークな構造生物学的・酵素学的特徴
Mpr1は、GNATスーパーファミリーに保存される配列モチーフを有しているものの8,10)、アミノ酸配列全体において相同性が高く、かつ立体構造が決定されているタンパク質は報告されていなかった。Mpr1が有する珍しい基質特異性と合わせて考えると、Mpr1が基質認識部位等を含め新規な立体構造を有し、ユニークな機構で酵素反応を触媒していることが予想された。そこで、X線結晶構造解析によりMpr1の立体構造の決定を試みたところ、ネイティブ状態の構造を1.9Åの分解能で、CHOPとの基質複合体構造を2.3Åの分解能で、それぞれ決定することに成功した11)。その結果、Mpr1は8本のβ-ストランドと6本のα-ヘリックスから成るフォールディングを有していることが分かった。また、Mpr1の会合状態を超遠心分離により解析したところ、Mpr1は基質の有無に関わらず溶液中で二量体を形成することが明らかとなった。これらのフォールディングや会合状態は、多くのGNATスーパーファミリータンパク質と類似したものであった11)。一方、Daliサーバー12)を用いてMpr1と立体構造が類似したタンパク質を探索したところ、Mpr1は真核生物よりも細菌のNATとよく似た立体構造を有することが明らかになった。
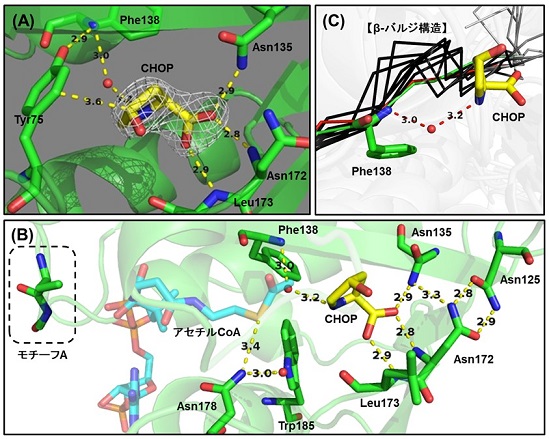
基質認識機構に着目すると、基質であるCHOPに相当する電子密度は、隣り合う2本のβ-ストランドが形成する裂け目状の構造に確認された11)。観察された電子密度をCHOP構造に帰属したところ、CHOPのカルボキシル基はAsn135の側鎖アミド基、Asn125とLeu173の主鎖アミド基と、またイミノ基はPhe138の主鎖アミド基と水分子を介して、それぞれ相互作用可能な位置に存在していた (図2A)。そこで、Asn135のアミノ酸置換型酵素を解析したところ、Asn135Alaは野生型の約20倍のKm値を示し、Asn135Asp型酵素は活性を示さなかった。さらに、種々のpH条件下でMpr1の活性を測定したところ、pHが酸性側にシフトするにつれて、Mpr1の基質に対するKm値が上昇した。以上のことから、Mpr1はAsn135とそれを含む領域により、基質のカルボキシレートアニオンを認識・結合し、酵素反応を行っていると考えられた。一方、基質の環状構造の認識については、Tyr75の側鎖がCHOPのγ位炭素原子と相互作用可能な位置に存在していたものの、厳密な認識機構は観察されなかった。環状二級アミンという特殊な構造の物質を基質とするにも関わらず、基質認識に厳密な様式が見られなかったことは、Mpr1の基質認識において、環状構造ではなくカルボキシル基の認識が重要であることを意味している。
続いて、Enterococcus faecium由来のアミノグリコシド-6’-N-アセチルトランスフェラーゼの立体構造 (PDB ID code : 1B87)13)とMpr1の立体構造を重ね合わせることにより、Mpr1-CHOP-AcCoAの三者複合体モデルを構築した。AcCoAとの複合体構造が決定されているタンパク質の中で最もMpr1と立体構造の相同性が高い1B87は、溶液中での会合状態もまたMpr1と同様に二量体である14)。Mpr1-CHOP-AcCoA三者複合体中のCHOP/AcCoA結合領域において、Mpr1と1B87の構造はよく重なっており、特にAcCoA周辺の構造は高い一致度を示した。GNATスーパーファミリーに属するタンパク質は、AからDまでの配列モチーフを有しており、そのうちモチーフAに存在するArg残基はAcCoAのピロリン酸部位と相互作用し、AcCoAの結合に寄与すると考えられている。Mpr1-CHOP-AcCoA三者複合体モデルにおいて、Mpr1のモチーフAのArg残基はAcCoAのピロリン酸と結合可能な位置に存在していた。以上のことから、構築した三者複合体モデルは概ね正しい構造であると考えられた (図2B)。
三者複合体モデルにおいて、Phe138の主鎖、およびAsn178とTrp185の側鎖が、AcCoAのカルボニル基と相互作用可能な位置に存在していた。GNATスーパーファミリーの多くの酵素において、以下のような触媒反応機構が報告されている。活性中心にある疎水性残基 (Mpr1ではPhe138) の主鎖が、隆起状のβ-バルジ構造によりオキシアニオンホールと呼ばれるポケットを形成する。オキシアニオンホールは、NAT反応における正四面中間体の酸素原子の負電荷を安定化することで、反応を触媒する。一方、活性中心付近の特定のTyr残基 (Mpr1ではTrp185に相当) は、側鎖のフェノール性水酸基によって、NAT反応後に生じるCoAチオレートアニオンをプロトン化することで、反応を進行させる8)。興味深いことに、Mpr1の触媒反応機構はこれらと全く異なるものであることが明らかとなった。まず、Mpr1のPhe135の主鎖はβ-バルジ構造を有していなかった (図2C)。また、CoAチオレートをプロトン化するTyr残基の位置に存在するTrp185は、Mpr1オルソログ間で保存されておらず、側鎖インドールのpKaを考慮すると、CoAチオレートをプロトン化することは難しいことから、触媒残基ではないと考えられた。一方、Mpr1のAsn178残基の側鎖アミド基はAcCoAの硫黄原子と相互作用可能な位置に存在すること、Asn178がMpr1オルソログ間で高度に保存されていることから、Asn178が反応を触媒する可能性が考えられた。そこで、精製組換え酵素を用いて解析したところ、Asn178Ala型酵素のkcat値は野生型のものと比べて著しく低下しており、Asn178Asp型酵素は活性を示さなかった。このことから、Asn178残基はNAT反応の生成物であるCoAチオレートアニオンを安定化することで反応を触媒すると考えられた。

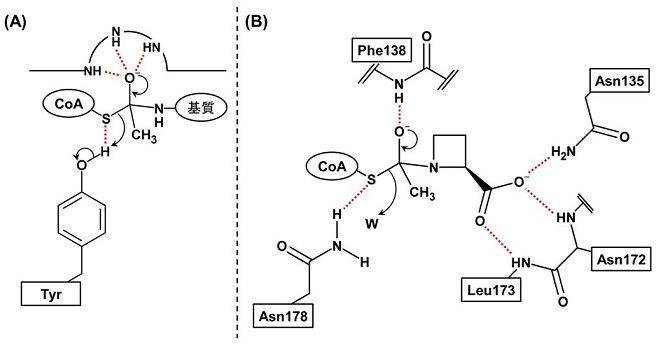
以上のことから、Mpr1の基質認識・反応触媒機構は以下のように考えられる11)(図3)。すなわち、Mpr1はAsn135を含む領域で基質のカルボキシレートを認識・認識結合する。続いて、基質イミノ基からAcCoAのカルボニル炭素に求核攻撃が起こり、生じた正四面中間体の酸素原子の負電荷をPhe138の主鎖が形成するオキシアニオンホールにより安定化し、反応を触媒する。但し、Mpr1はβ-バルジを持たないことから、オキシアニオンホールの形成は不十分であると考えられる。さらに、正四面中間体の分解によりアセチル化された基質とともに生じるCoAチオレートアニオンをAsn178の側鎖アミドが安定化することにより、反応が進行する。Mpr1は、環状二級アミンという特殊な構造の分子を基質とするにもかかわらず、基質認識そのものは環状構造を厳密に認識する様式ではなかった。しかし、Asn残基により反応を触媒するGNAT酵素はこれまで報告されておらず、Mpr1は、その基質特異性や活性中心の局所構造のみならず、反応機構においてもユニークなものを有することが明らかとなった。

Mpr1と既知のGNATタンパク質の基質特異性の差異を立体構造・反応機構から比較すると、基質認識ではなく触媒反応機構にこそ、Mpr1のユニークな基質特異性を説明しうる可能性が見えてくる。Mpr1は環状二級アミンを認識する厳密な機構を有さない一方、触媒反応に必要なオキシアニオンホールの形成に重要なβ-バルジを持っていない。従って、Mpr1による触媒反応においてオキシアニオンホールの形成は、他のGNATタンパク質に比べて不十分で、これによる触媒効率も低いと考えられる。一般に、二級アミンは一級アミンよりも求核性が高い。さらに、環状二級アミンは嵩高い置換基が固定され、求核攻撃を行う窒素原子上の非共有電子対の周辺に立体障害が少ない。従って、環状二級アミンは一級アミンに比べて極めて反応性の高い基質であると考えられる。Mpr1がβ-バルジを持たないこと、そのために恐らく触媒効率が低いであろうことは、反応性の高い環状二級アミンを基質として扱う上で、適度な反応速度を維持するために進化的に保存されてきた仕組みではないだろうか?
4.立体構造に基づいたMpr1の理論的分子設計
後で詳述するように、Mpr1は酵母のアルギニン合成を促進する。アルギニンは発酵速度やストレス耐性を向上させることが報告されており15-17)、アルギニンを高生産・高蓄積する株は産業応用上、有用であると考えられる。そこで筆者らは、Mpr1の立体構造・触媒反応機構の情報をもとに、機能を向上させたMpr1を理論的に分子設計し、酵母でこれを発現させることでアルギニン合成を強化した株を構築することを試みた18)。下記のように、タンパク質の熱安定性と細胞内存在量は相関すると予想されることから、ここでは、熱安定性を向上させたMpr1の構築に取り組んだ。
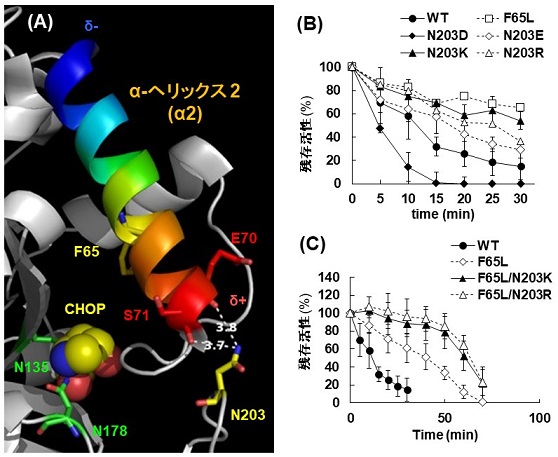
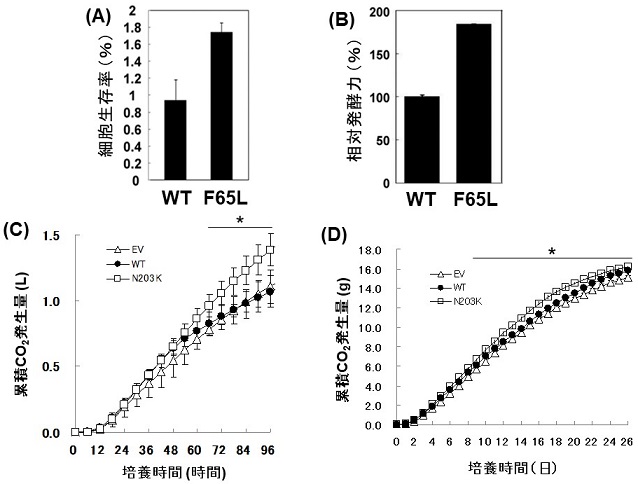
これまでに筆者らは、MPR1へのランダム変異導入により熱安定性の向上したPhe65Leu型Mpr1を取得している (図4A)19)。Phe65は2番目のα-ヘリックス (α2) 上に存在すること、また同じくα2上のAsn66残基の変異が熱安定性を低下させることから (未発表データ)、α2がMpr1の立体構造安定性に重要であると考えた。そこで、α2の双極子に起因する電荷を中和することで、α2自身を安定化することを試みた。立体構造を見ると、α2のC末端にあるGlu70とSer71の主鎖カルボニル酸素が、Asn203の側鎖アミドによって安定化されていることが考えられた。同様の方式でα2 をさらに安定化させるため、Asn203の位置に塩基性アミノ酸を導入し、α2双極子の負電荷を中和することを試みた (図4A)。精製組換え酵素を用いた解析の結果、Asn203Lys型およびAns203Arg型Mpr1は野生型酵素に比べて極めて高い熱安定性を示した18)(図4B)。一方、Asn203Asp型Mpr1は反対に熱安定性を低下させたことから、α2双極子の中和による安定化は、Mpr1の立体構造安定性に極めて重要であることが分かった。Asn203Lys、Asn203ArgにPhe65Leuを組み合わせたMpr1は、さらに高い熱安定性を示した (図4C)。構築した熱安定型Mpr1を酵母で発現させアルギニン合成能を検証した結果、熱安定型Mpr1を発現した株は野生型発現株に比べて高いアルギニン合成能を示した。さらに、熱安定型Mpr1発現株は、野生型Mpr1発現株に比べて有意に高い発酵力を示した (未発表データ)。
熱安定型Mpr1が酵母のアルギニン合成能を強化する機構は明確には分かっていない。一般的に、タンパク質の熱安定性とプロテアーゼによる分解に対する耐性は、ある程度相関すると言われている。そのため、熱安定型Mpr1は細胞内でのタンパク質ターンオーバーの際、プロテアーゼに耐性を示し、結果的に細胞内Mpr1量が増加したことで、アルギニン合成能の向上につながったと考えられる。

5.Mpr1依存的なアルギニン合成機構
2項で述べたように、Mpr1はAZC、CHOPと言った環状二級アミンを基質とする。AZCはスズランを含む一部の植物で合成される一方20,21)、CHOPは根粒菌由来の酸化酵素によりプロリンを基質として合成されるが22)、酵母細胞内でこれらの物質は検出されていない。また、スズラン周辺の土壌から単離されたPseudomonas属細菌はAZCを窒素源として資化することが報告されているが23)、一方、酵母はMPR1遺伝子の有無に関わらず、AZCを窒素源とすることはできない (未発表データ)。このように、Mpr1の細胞内基質および生理機能は未だ明らかにはなっていない。
筆者らは、Mpr1の生理機能を明らかにする目的で、酵母のアルギニン合成に着目して解析を行った。酵母のアルギニン合成は以下のように行われる。出発物質であるGluに対し、Arg2、Arg6、Arg5、Arg8、Arg7がそれぞれ触媒するアセチル化、リン酸化、還元、アミノ基転移、脱アセチル化反応によりオルニチンが合成された後、数段階の反応を経てアルギニンが合成される (図5A)。筆者らは、Mpr1とアルギニン合成の関連性を解析するために、遺伝子破壊株や変異株のアルギニン非含有培地における生育を評価した。その結果、Δarg2株のアルギニン非含有培地における生育は著しく低下したが、これはMPR1の過剰発現により回復した。一方、Arg6とArg5は同一遺伝子 (ARG5,6) 上にコードされるが、Δarg5,6株が示したアルギニン要求性は、MPR1の過剰発現により相補されなかった。さらに、Arg6の活性残基を置換したArg6失活株のアルギニン要求性は、MPR1の過剰発現によって相補されなかった。また、Δarg2のアルギニン要求性は、N-アセチル-グルタミン酸 (NAG) を培地に添加することで回復した。以上のことから、Mpr1は何らかの機構でNAGを供給することにより、アルギニン合成に寄与すると考えられた。
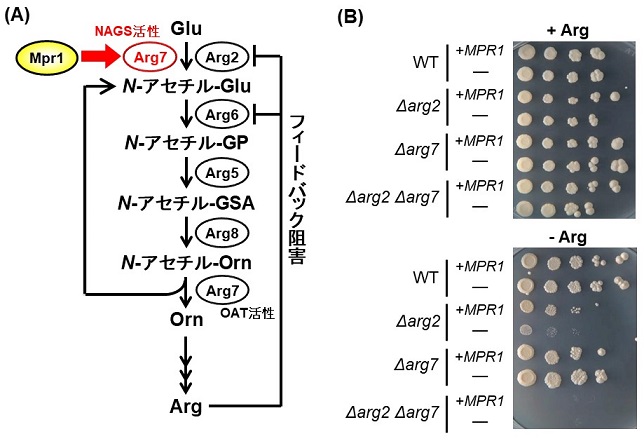

酵母においてNAGは、AcCoAをアセチル基供与体とするNAG合成酵素 (NAGS) 活性を持つArg2と、N-アセチルオルニチンをアセチル基供与体とするオルニチンアセチルトランスフェラーゼ (OAT) 活性を持つArg7によって合成される。OAT反応によるNAG合成は新規合成ではないが、Arg7はNAGS活性をも有しているため24)、両酵素が酵母内におけるNAG新規合成を担っていると言える。そこで、Δarg2株およびΔarg7株のアルギニン要求性を解析したところ、Δarg2株のアルギニン要求性はMPR1の過剰発現により回復したのに対し、Δarg7株の部分的なアルギニン要求性はMPR1の過剰発現によって回復しなかった (図5B)。さらに、MPR1がArg7タンパク質量に及ぼす影響をウェスタンブロットにより解析したところ、MPR1過剰発現によりArg7タンパク質量が増加することが明らかとなった。以上のことから、Mpr1はArg7のタンパク質量を何らかの機構で増加させ、そのNAGS活性によりNAGを供給することで、アルギニン合成に寄与していると考えられた。しかし依然として、Mpr1依存的なアルギニン合成経路には未解明な部分が多く残されており、Mpr1によるArg7量増加の分子機構を含め、さらなる解析を行っている。
6.バイオテクノロジーや発酵産業におけるMpr1の応用研究
Mpr1はAZCを無毒化する上で、極めて強力・高活性であり特異的な酵素である。筆者らはこれに着目し、MPR1とAZCを組み合わせることで、微生物や植物における新たな選択マーカーとして、形質転換に利用できることを示した25-27)。一方、Mpr1の基質の一つであるCHOPには抗がん作用があることが知られており、細胞膜透過性や細胞内寿命を制御するため、様々なCHOP誘導体が抗がん剤候補として合成・解析されている28-30)。CHOPのアセチル化体であるN-アセチル-CHOPも同様に抗がん剤としての可能性を有すると考えられるが、水酸基を有するCHOPを化学合成により選択的にN-アセチル化することは容易ではない。そこで筆者らは、Mpr1によるCHOPのN-アセチル化に着目し、Mpr1を発現させた大腸菌を用いることで、発酵法によるN-アセチル-CHOPの生産に成功した9,31)。
筆者らは、野生型および高機能型のMpr1を応用することで、産業酵母に有用な特性を付与する研究も行ってきた。これまでに、熱安定型Phe65Leu-Mpr1をパン酵母で発現させることにより、製パン関連ストレスである冷凍や乾燥に対する耐性や、これらのストレス曝露後の発酵力が向上した (図6A, 6B)32,33)。また最近、清酒酵母で熱安定型Asn203Lys-Mpr1を発現させることで、清酒酵母の発酵力が上昇することを見出した (図6C, 6D)34)。さらに、この株を用いて日本酒を製造したところ、カプロン酸エチルのような香気成分、また日本酒の味に大きく影響するリンゴ酸やピルビン酸などの有機酸の含量が、著しく変化することを見出した。これは、バリエーション豊かな日本酒を製造するという点において、極めて重要な結果といえる。

7.おわりに
本稿で触れた通り、Mpr1は極めてユニークな構造生物学的・酵素学的特徴を持つ新規のNATである。また、最近明らかになりつつあるアルギニン合成経路への寄与についても、同様の機能を持つタンパク質・酵素は発見されておらず、独特の生理機能をも有することが分かってきた。一方、Mpr1の有用機能はこれまでに様々な分野・手法で応用されているが、その発現機構については未解明な部分も多い。新規なアルギニン合成経路への関与を含めMpr1の生理機能をより詳細に明らかにすることで、さらなる産業応用が期待できる。これからも基礎・応用の両面から、このユニークな酵素の研究を進めていきたい。
謝辞
本研究の一部は、公益財団法人野田産業科学研究所の2016年度「奨励研究助成」(那須野 亮)、科学研究費補助金の2019年度「挑戦的研究 (萌芽) 19K22282」(高木 博史) の助成を受けましたことを記して深謝いたします。
文献
1) Evans, D. A.: Pharmacol. Ther., 42, 157 (1989).
2) Perez-Gallardo, R. V., Briones, L. S., Diaz-Perez, A. L., Gutierrez, S., Rodriguez-Zavala, J. S., Campos-Garcia, J.: FEMS Yeast Res., 13, 804 (2013).
3) 高木博史: 蛋白質核酸酵素, 53, 249 (2008).
4) Baum, B. J., Johnson, L. S., Franzblau, C., Troxler, F.: J. Biol. Chem., 250, 1464 (1975).
5) Bessonov, K., Bamm, V. V., Harauz, G.: Phytochemistry, 71, 502 (2010).
6) Takagi, H., Shichiri, M., Takemura, M., Mohri, M., Nakamori, S.: J. Bacteriol., 182, 4249 (2000).
7) Shichiri, M., Hoshikawa, C., Nakamori, S., Takagi, H.: J. Biol. Chem., 276, 41998 (2001).
8) Vetting, M. W., S de Carvalho, L. P., Yu, M., Hegde, S. S., Magnet, S., Roderick, S. L., Blanchard, J. S.: Arch. Biochem. Biophys., 433, 212 (2005).
9) Hoa, B. T., Hibi, T., Nasuno, R., Matsuo, G., Sasano, Y., Takagi, H.: J. Biosci. Bioeng., 114, 160 (2012).
10) Kotani, T., Takagi, H.: FEMS Yeast Res., 8, 607 (2008).
11) Nasuno, R., Hirano, Y., Itoh, T., Hakoshima, T., Hibi, T., Takagi, H.: Proc. Natl. Acad. Sci. U.S.A., 110, 11821 (2013).
12) Holm, L., Rosenstrom, P.: Nucleic. Acids. Res., 38, W545 (2010).
13) Wybenga-Groot, L. E., Draker, K., Wright, G. D., Berghuis, A. M.: Structure, 7, 497 (1999).
14) Burk, D. L., Ghuman, N., Wybenga-groot, L. E., Berghuis, A. M.: Protein Sci., 12, 12, 426 (2003).
15) Thomas, K. C., Ingledew, W. M.: Can. J. Microbiol., 38, 626 (1992).
16) Gutierrez, A., Chiva, R., Sancho, M., Beltran, G., Arroyo-Lopez, F. N., Guillamon, J. M.: Food Microbiol., 31, 25 (2012).
17) Nishimura, A., Kotani, T., Sasano, Y., Takagi, H.: FEMS Yeast Res.,; 10, 687 (2010).
18) Nasuno, R., Hirase, S., Norifune, S., Watanabe, D., Takagi, H.: J. Biochem., 159, 271 (2015).
19) Iinoya, K., Kotani, T., Sasano, Y., Takagi, H.: Biotechnol. Bioeng., 103, 341 (2009).
20) Fowden, L.: Biochem. J., 64, 323 (1956).
21) Fowden, L., Bryant, M.: Biochem. J., 71, 210 (1959).
22) Hara, R., Kino, K.: Biochem. Biophys. Res. Commun., 379, 882 (2009).
23) Gross, C., Felsheim, R., Wackett, L. P.: J. Bacteriol., 190, 4859 (2008).
24) Crabeel, M., Abadjieva, A., Hilven, P., Desimpelaere, J., Soetens, O.: Eur. J. Biochem., 250, 232 (1997).
25) Ogawa‐Mitsuhashi, K., Sagane, K., Kuromitsu, J., Takagi, H., Tsukahara, K.: Yeast, 26, 587 (2009).
26) Zhang, X. H., Takagi, H., Widholm, J. M.: Plant Cell Rep., 22, 615 (2004).
27) Tsai, F. Y., Zhang, X. H., Ulanov, A., Widholm, M. J.: J. Exp. Bot., 61, 2561 (2010).
28) Uitto, J., Hoffman, H., Prockop, D. J.: Science, 190, 1202 (1975).
29) Simon, P. M., Pachence, J., Belinka, B., Poiani, G. J., Lu, S. E., Tozzi, C. A., Riley, D. J.: Pulm. Pharmacol. Ther., 19, 242 (2006).
30) Sturm, D., Maletzki, C., Braun, D., Emmrich, J.: Int. J. Colorectal Dis., 25, 921 (2010).
31) Bach, T. M., Hara, R., Kino, K., Ohtsu, I., Yoshida, N., Takagi, H.: Appl. Microbiol. Biotechnol., 97, 247 (2013).
32) Sasano, Y., Haitani, Y., Hashida, K., Ohtsu, I., Shima, J., Takagi, H.: Microb. Cell Fact., 11, 40 (2012).
33) Sasano, Y., Takahashi, S., Shima, J., Takagi, H.: Int. J. Food Microbiol., 138, 181 (2010).
34) Ohashi, M., Nasuno, R., Watanabe, D., Takagi, H.: J. Ind. Microbiol. Biotechnol., 46, 1039 (2019).