【トピックス】
イミン還元酵素を利用したキラルアミン合成へのアプローチ
満倉浩一、吉田豊和
岐阜大・工
1.はじめに
キラルアミンは、天然アルカロイド、低あるいは中分子医薬品や農薬の骨格に含まれていることが多く、有用物質の一つに挙げられる。近年、アメリカ食品医薬品局 (FDA) によって承認された医薬品の解析から新規化学物質の約40%が、一つあるいはそれ以上のキラルアミン骨格を含むことが報告された1)。このような状況下、医薬品や農薬合成におけるキラルアミンの需要は今後も高いと予想されるため、効率的合成法の開発や新規合成ルートの探索は、依然として興味深い研究テーマと言えるだろう。最近、生体触媒を利用したキラルアミン合成において、アミノ基転移酵素2,3)やアミン脱水素酵素4,5)のようなアミン生成酵素群を補完する選択肢として、イミン還元酵素が注目されている。
これまでに天然化合物の炭素-窒素二重結合 (イミン) を立体選択的に還元する酵素がEC1.5.1に属するNAD(P)H依存酸化還元酵素として報告されているが、これらの酵素は基質特異性が高く、非天然のキラルアミン合成の利用には適していなかった6)。そこで、筆者らは非天然イミン2-メチル-1-ピロリン (2-MPN) を用いた酵素活性スクリーニングによって、土壌放線菌から2種類の新奇イミン還元酵素、(R)-選択性を示すStreptomyces sp. GF3587由来イミン還元酵素(R-IRED)と(S)-選択性を示すStreptomyces sp. GF3546由来(S-IRED)を見出し7)、それらの特徴を明らかにしてきた8,9)。本稿では、IREDの発見から10年間に報告された知見を整理し、IREDの特徴、種類、キラルアミン合成への応用の観点から解説する。
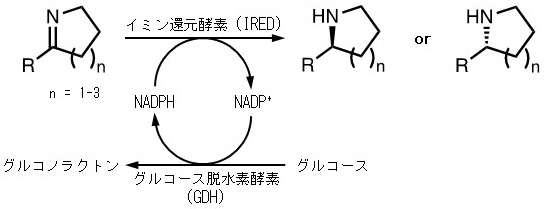
図1 イミン還元酵素とグルコース脱水素酵素を用いたキラル環状アミン合成
2.効率的なキラル環状アミン生産に適した組換え体の作製
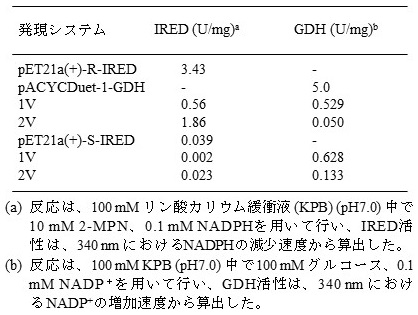
GF3587由来R-IREDおよびGF3546由来S-IREDはともにホモダイマー構造をとる酵素であり、還元反応では補酵素としてNADPHを要求し、中性条件下でアミン生成がよく進行する特性をもつ。プロキラルな環状イミンから光学活性環状アミンを安価で効率よく生産するためには、NADPH再生系を組み込んだ組換え体を作製する必要がある (図1)。そこで、IRED遺伝子に加えてBacillus subtilis由来グルコース脱水素酵素 (GDH) 遺伝子を組み込んだ組換え体の作製を検討した。最初に、pACYCDuet-1ベクターのMCS1にIRED遺伝子をMCS2にGDH遺伝子を組み込んだ1Vシステム、次にpET21a(+) ベクターにIRED遺伝子、pACYCDuet-1ベクターのMCS2にGDH遺伝子を組み込んだ2つのベクターが共存する2Vシステムの組換え体を作製した (図2)。さらにpACYCDuet-1ベクターのMCS1にGDH遺伝子をMCS2にS-IRED遺伝子を組み込んだ1V’システムの組換え体も作製し、S-IREDとGDHの活性を評価したところ、IRED活性は1Vシステムと比べて大きく低下しなかったが、GDH活性は400分の1に低下した。したがって、以後の実験では1V’システムは使用しなかった。1Vと2Vシステムの組換え体を培養したのち細胞破砕し、得られた無細胞抽出液をSDS-PAGEによって解析した結果、どちらのシステムでもIREDとGDHの発現が確認された (写真はS-IREDとGDHの組み合わせを掲載しているが、R-IREDと GDHの組み合わせも同じ傾向を示す) (図3)。IRED遺伝子あるいはGDH遺伝子を単独で組み込んだ組換え体、1Vシステムと2Vシステムの組換え体をそれぞれ破砕して調製した無細胞抽出液を用いて、IREDとGDHの活性を評価した (表1)。その結果、IREDあるいはGDH遺伝子単独で発現させた際の酵素活性と比較して、1Vシステムを用いた場合では、IREDとGDHの活性は共に低下した一方、2Vシステムでは、GDH活性は低下したものの、IRED活性は大きく低下しなかった。そこで、1Vと2Vシステムのいずれが光学活性2-メチルピロリジン (2-MP) 生産に優れているかを菌体反応で評価した。その結果、R-IREDとGDH遺伝子の組み合わせの場合、2-MPの生産性 (1V :0.35 gL-1h-1、2V :0.43 gL-1h-1) は2Vシステムの方が若干良いものの、いずれのシステムでも最終的な変換率は100%、2-MPの光学純度は>99% ee (R)であった。S-IREDとGDHの組み合わせでも、同じ傾向を示したが、S-IREDは生成物阻害を受けるため変換率は88%に留まった。近年、2Vシステムのような組換え菌体を「All-in-one」whole-cell biocatalystとして利用することが増加しており、IREDの環状イミンに対する基質特異性の評価や小スケールでのキラル環状アミン生産に有効であることが示された10,11)。
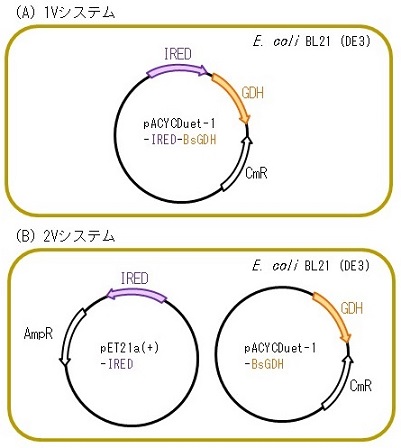
図2 IREDとGDHの大腸菌内共発現システム
.jpg)
sentence.jpg)
表1 発現システムによるIREDとGDHの活性
3.イミン還元活性を示す酵素の種類と配列の特徴
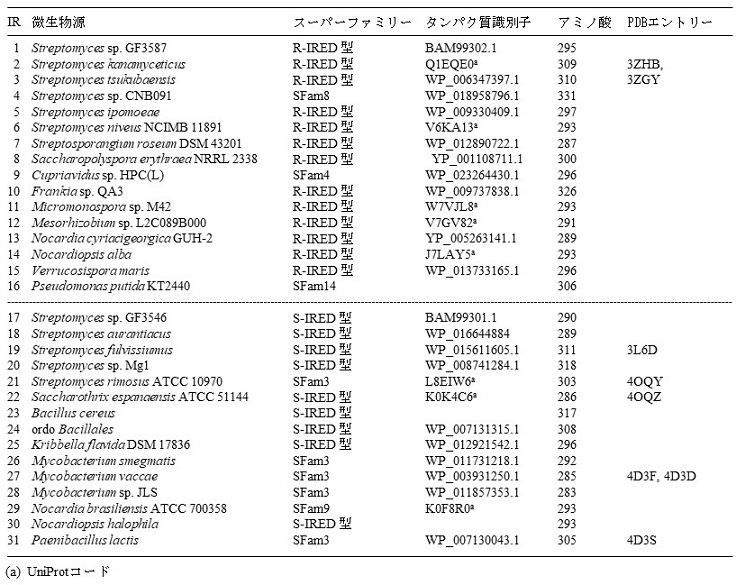
いくつかのIRED構造の特徴が解析されてきたが、反応メカニズム、構造活性相関や立体選択性に関わるアミノ酸残基の情報は限られていた。そこで、IRED配列と触媒機能の相関性についての情報を得る目的でImine Reductase Engineering Database (www.IRED.BioCatNet.de) がシュトゥットガルト大学の研究者らによって設立された12)。2019年8月時点で、このデータベースに登録されている生物は、細菌 (主に放線菌) が1446件と圧倒的に多く、真核生物は12件と非常に少ない。IREDあるいはその候補配列は1409件で、タンパク質として1136件が掲載されている。IRED配列には14種類のスーパーファミリーがあり、R-IRED (相同性ファミリーHFam1.1と1.2を含む)、S-IRED (HFam2.1と2.2)、SFam3〜14に分類されている。これらのうちR-IREDタイプは567件、S-IREDタイプでは168件が登録されている。我々がStreptomyces属由来のR-IRED (GF3587) およびS-IRED (GF3546) を報告して以降、IREDの配列と特徴が調べられ、IREDと低い配列相同性を示すPseudomonas putida KT2440のホモログからもIRED活性を示すもの (SFam14) が見出されている。多数のIREDおよびその候補のうち、すでに活性を示すと報告されているものを選択し、微生物源、スーパーファミリー、アミノ酸配列と立体構造情報についてまとめた (表2)11-13)。IRED 構造のNADPHが結合するRossmann foldモチーフを含むN末端ドメイン (約170アミノ酸) は、高度に保存されている一方、C末端ドメイン (約100アミノ酸) は可変な領域である。この領域は、IREDのスーパーファミリー特異的に保存されているが、R-IREDとS-IREDで異なることが明らかになった。標準的なIRED配列に基づくと、S-IREDは139Proと194Pheが存在しているのに対し、R-IREDでは139番目のアミノ酸がVal、Thr、Ileのような疎水性残基であり、194番目はMetやLeuが多い。イミンの還元反応において、基質のプロトン化に関わるアミノ酸残基が調査されており、X線結晶構造解析が行われたStreptomyces kanamyceticus由来R-IRED (IR2)14)では187Aspが、GF3546 S-IRED (IR17)15)では169Tyrが、イミンのプロトン化に関与する。IREDの相同性検索から、ほとんどのIREDはβ-ヒドロキシ酸脱水素酵素 (β-HAD) と構造類似性を示すにも関わらず、β-ヒドロキシ酸の変換を触媒しない。この原因の一つとして、β-HADでは、酸化還元の際に基質をプロトン化するアミノ酸がLysであることが挙げられる。上述したように、構造特性や酵素活性および反応機構に関与する分子情報の知見も蓄積されてきた。最近、カスケード反応のような多段階酵素反応への利用の観点から、IREDの補酵素をNADPHからNADHにスイッチングする試みも始まっている16)。Frances H. Arnold研究室で開発されたオンラインツールを利用して、Myxococcus stipitatus由来R-IRED (WT) の10種類の変異体が作製された。それらの中でアミノ酸配列の一部がN32E、R33Y、T34E、K37Aに置換された酵素を使用すると立体選択性は変わらないが、補酵素特異性 (NADH/NADPH [min-1/min-1]) が0.022 (WT) から64と約2900倍向上することが示された。この選択性は実用的なレベルに至っていないが、今後、完全なNADH依存IREDの作製とそれを利用したキラルアミン合成が行われることを期待したい。
.jpg)
sentence.jpg)
表2 イミン還元酵素の種類と分類
近年、イミン還元活性を示すIRED以外の酵素が報告されるようなった。例えば、Bacillus subtilis由来グルコース脱水素酵素は、6位に臭素原子やシアノ基をもつ1,2-ジメチルイソキノリウム塩 (10 mM) に作用し、最大58%の変換率で対応するアミンを86%〜>99% ee (R)で生成する17)。また、植物からもイミン還元活性を示す酵素が報告されている。ヒガンバナ科のアルカロイドの生合成に関与しているラッパスイセン (Narcissus pseudonarcissus) 由来のノルオキソマリチジン還元酵素 NR (短鎖型脱水素酵素/還元酵素 (SDR) の一種) が、前駆体のイミンに対して微弱な (骨格中のエノンのC=C還元活性と比べて400分の1) 還元活性を示すことが見出された。そこで、NR配列と87%の相同性を示すヒガンバナ科タマスダレ属 (Zephyranthes treatiae) 由来の推定SDR (Zt-SDR) についても、組換え体から精製した酵素を用いてイミン (10 mM) に対する還元活性が調査された。NRはピロリン環 (表3、1a) に対する活性は低いが (変換率10%)、1,2-ジメチルイソキノリウム塩を用いると変換率>99%で対応するアミンを92% ee (R) で生成し、β-カルボリン (表3、6a) を用いると、変換率99%で対応するアミンを60% ee (S)で生成する。一方、Zt-SDRもNRとほぼ同じ変換率でアミンを生成したが、光学純度は中程度であった18)。このように本来の活性とは異なる「Promiscuous」活性の発見は、キラルアミンの新規合成法の開発につながるかもしれない。
表3 GF3587 R-IRED (IR1) とGF3546 S-IRED (IR17) の基質特異性
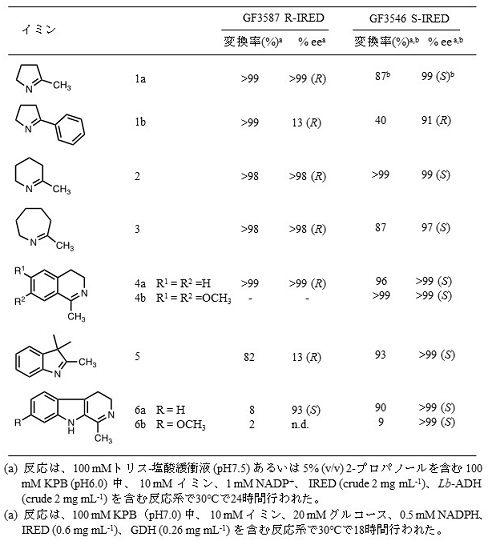
4.キラルアミン合成への応用
GF3587R-IRED (IR1) とGF3546S-IRED (IR17) の基質特異性を示した8,9,11) (表3)。IR1は、2位にメチル基をもつ5〜7員環イミン (1a、2、3) とイソキノリン (4a) に対して高い変換率と立体選択性を示すが、立体障害のあるイミンを用いると反応性あるいは立体選択性 (使用するイミンによってはどちらも) が低くなる傾向を示す。一方、IR17では、2-フェニル-1-ピロリン (1b) に対する変換率は中程度であるが、ピロリン (1a)、ピペリデイン (2)、アゼピン (3)、イソキノリン (4)、3H-インドール (5)、β-カルボリン (6a) には高い変換率と立体選択性を示すことから、基質特異性の広い酵素と言える。また表2に示したIREDのうち、良好な変換率と優れた立体選択性を示したものについて基質特異性を示した11,13) (表4)。(R)-選択性を示すIREDについては、IR1と類似の基質特異性を示すものが多い。IR10は、2-フェニル-1-ピロリン (1b) にも良好な (R)-選択性を示し、IR6、11、12、14はイソキノリン (4a) に高い変換活性と極めて高い (R)-選択性を示す。IR21は3H-インドール (5) を効率よく対応する (R)-アミンに変換する。一方、(S)-選択性を示すIREDは、IR17と同様に広い基質特異性を示すものが多く、それらの中でIR22、25はメトキシ基をもつβ-カルボリン (6b) に対しても高い変換率と選択性を示す。このように種々の環状イミンに作用するIREDは魅力的な生体触媒であり、キラルアミンの新規な合成に関してインスピレーションを与えてくれる。例えば、芳香族ヘテロ環化合物の中で、光学活性なオキサゾリン環やチアゾリン環は医薬品の重要な骨格であり、効率的な合成が望まれている。IREDライブラリーを用いてスクリーニングを行うことで、高い光学純度で対応するアミンを生成するIREDを見いだすことができる19,20) (図4A1-4)。Whole-cell biocatalystを用いた反応条件を最適化することで、スピロ構造の (S)-3-チアゾリン環化合物 (>99% ee) を高変換率で調製することもできる (図4B)20)。また、カルボニル化合物とアミン類から還元的アミノ化によって光学活性アミンを合成する反応も見出されており、IR13とGDHを用いてインダノンと過剰量のプロパルギルアミンから、パーキンソン病治療薬 (R)-ラサギリンが合成された (図4C)21)。しかしながら、IREDによる還元的アミノ化反応では多量の酵素が必要であり、反応平衡をアミン生成側にシフトさせる必要があるため、使用するアミンがカルボニル化合物に比べて数十当量必要となることが問題であった。そこで、高い還元的アミノ化活性を示す菌株の探索がAmycolatopsis orientalisやStreptomyces属放線菌由来IREDの配列情報を基にして行われ、Aspergillus oryzaeから還元的アミナーゼ (AspRedAm、Uniprot Q2TW47) が見出された22)。AspRedAmを用いてカルボニル化合物の還元的アミノ化が調査された結果、シクロヘキサノン、2-ヘキサノン、4-フェニルブタナールと第1級アミンとの組み合わせが、非常に良好 (カルボニル化合物に対するアミンの比率が1〜4当量) な反応性を示し、対応するアミンを高い変換率で生成する一方、インダノンのような骨格からアミンを合成する場合、過剰量 (50当量程度) のアミンが必要であることが示された。最後に、近年報告されたIREDを含む酵素カスケード反応23)によるキラルアミン合成について述べる。細胞内にカルボン酸還元酵素 (CAR)、アミノ基転移酵素 (TA) とIREDを組み込むことで、ケト酸からOne-Potで光学活性環状アミンを合成する方法が報告された24)。しかしながら、この合成法ではCARの反応にATPが必要であることに加えて、IREDの補酵素再生系も必要となるため、現時点で、光学活性環状アミンの効率的な合成に至っていない。今後、上述した還元的アミノ化のように、化合物の特性や反応デザインを考慮した酵素反応の利用が進展すると思われる。
表4 他のIREDの基質特異性
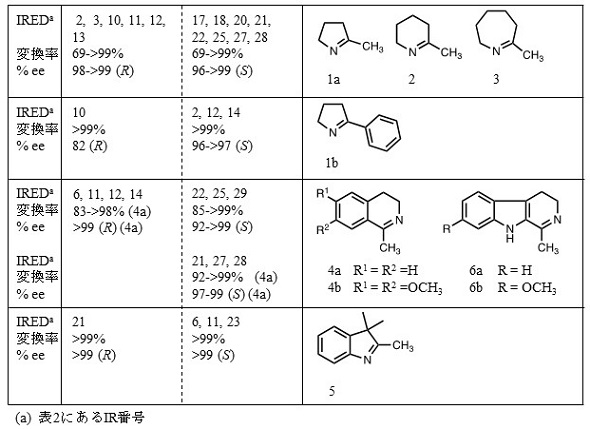
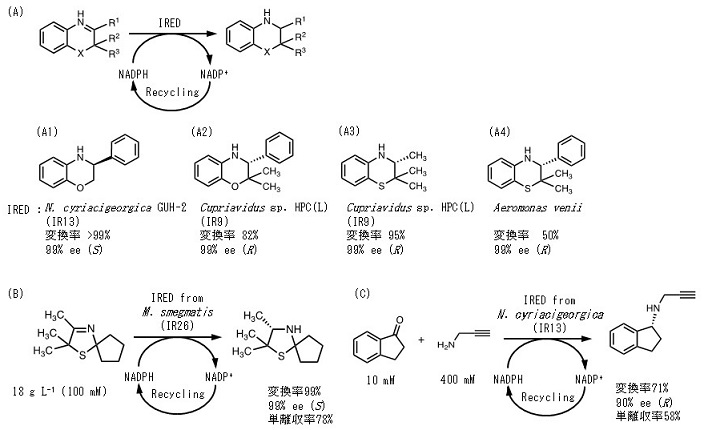
図4 IREDとGDHの共役による光学活性アミン合成への応用
5.おわりに
本稿では、IREDやイミン還元活性を示す酵素の特徴ならびにキラルアミン合成について情報を整理し解説してきた。IREDは、イミンからキラルアミンを合成できるだけでなく、カスケード反応やカルボニル化合物の還元的アミノ化にも利用できるため、いろいろな角度から研究を展開できる酵素の一つと言える。筆者らは、IRED活性スクリーニングに水系で安定なイミンを用い伝統的な手法を駆使して高活性菌を見出したが、現在では、新たなIRED配列が判明するとその配列を用いたIn silicoスクリーニングによって、さらに新たなIREDを見出すことができるようになった。つまり、酵素の配列情報が非常に重要であり、これからも新旧様々な方法による新奇酵素の探索が続くと予想される。近年、SDGsの指針が示され、地球環境(人にも)に優しい化合物の合成ルートの設定が必要不可欠となっている。今後、筆者らも安価な原料から短い工程(化合物の精製を含まない)でかつ安全な合成反応に利用できる有用酵素の探索ならびに有用物質合成法の開発に取り組んでいきたいと考えている。
謝辞
本研究は、長澤 透岐阜大学名誉教授、吉田豊和教授の指導のもとで行われました。本研究の成果は、学生諸君の努力の賜物であり、ご協力頂いた全ての方々に感謝申し上げます。
文献
1) Javis, L. M.: Chem. Eng. News, 94, 12-17 (2016).
2) Guo, F., Berglund, P.: Green Chem., 19, 333-360 (2017).
3) Calvelage, S., Dorr, M., Hohne, M., Bornscheuer, U. T.: Adv. Synth. Catal., 359, 4235-4243 (2017).
4) Knaus, T., Bohmer, W., Mutti, F. G.: Green Chem., 19, 453-463 (2017).
5) Tseliou, V., Masman, M. F., Bohmer, W., Knaus, T., Mutti, F. G.: ChemCatChem, 20, 800-812 (2019).
6) Mitsukura, K., Yoshida, T.: Future directions in biocatalysis 2nd Ed., Chapter 5, pp.97-117 (2017).
7) Mitsukura, K., Suzuki, M., Tada, K., Yoshida, T., Nagasawa, T.: Org. Biomol. Chem., 8, 4533-4535 (2010).
8) Mitsukura, K., Suzuki, M., Shinoda, S., Kuramoto, T., Yoshida, T., Nagasawa, T.: Biosci. Biotechnol. Biochem., 75, 1778-1782 (2011).
9) Mitsukura, K., Kuramoto, T., Yoshida, T., Kimoto, N., Yamamoto, H., Nagasawa, T.: Appl. Microbiol. Biotechnol., 97, 8079-8086 (2013).
10) 満倉浩一:酵素工学研究会第75回講演会講演要旨集, pp.18-21(2016).
11) Velikogne, S., Resch, V., Dertnig, C., Schrittwieser, J. H., Kroutil, W.: ChemCatChem, 10, 3236-3246 (2018).
12) Fademrecht, S., Scheller, P. N., Nestl, B. M., Hauer, B., Pleiss, J.: Proteins Struct. Funct. Bioinf., 84, 600-610 (2016).
13) Wetzl, D., Berrera, M., Sandon, N., Fishlock, D., Ebeling, M., Muller, M., Hanlon, S., Wirz, B., Iding, H.: ChemBioChem, 16, 1749-1756 (2015).
14) Rodriguez-Mata, M., Frank, A., Wells, E., Leipold, F., Turner, N. J., Hart, S., Turkenburg, J. P., Grogan, G.: ChemBioChem, 14, 1372-1379 (2013).
15) Huber, T., Schneider, L., Prag, A., Gerhardt, S., Einsle, O., Muller, M.: ChemCatChem, 6, 2248-2252 (2014).
16) Borlinghaus, N., Nestl, B. M.: ChemCatChem, 10, 183-187 (2018).
17) Roth, S., Prag, A., Wechsler, C., Marolt, M., Ferlaino, S., Ledeke, S., Sandon, N., Wetzl, D., Iding, H., Wirz, B., Muller, M.: ChemBioChem, 18, 1703-1706 (2017).
18) Roth, S., Kilgore, M. B., Kutchan, T. M., Muller, M.: ChemBioChem, 19, 1849-1852 (2018).
19) Zumbragel, N., Machui, P., Nonnhoff, J., Groger, H.: J. Org. Chem., 84, 1440-1447 (2019).
20) Zumbragel, N., Merten, C., Huber, S. M., Groger, H.: Nat. Commun., 9, 1949 (2018).
21) Matzela, P., Ganda, M., Hohne, M.: Green Chem., 19, 385-389 (2017).
22) Aleku, G. A., France, S. P., Man, H., Mangas-Sanchez, J., Montgomery, S. L., Sharma, M., Leipold, F., Hussain, S., Grogan, G., Turner, N. J.: Nat. Chem., 9, 961-969 (2017).
23) Schrittwieser, J. H., Velikogne, S., Hall, M., Kroutil, W.: Chem. Rev., 118, 270-348 (2018).
24) France, S. P., Hussain, S., Hill, A. M., Hepworth, L. J., Howard, R. M., Mulholland, K. R., Flitsch, S. L., Turner, N. J.: ACS Catal., 6, 3753-3759 (2016).