【トピックス】
高基質親和性を有するL-アルギニン酸化酵素の分子進化解析
中野祥吾、丹羽正純、伊藤創平、淺野泰久
静岡県大・食栄、静岡県大・食栄、静岡県大・食栄、富山県大・生・医工研セ
はじめに
諸説あるが、36〜40億年ほど前、生命は地球に誕生した。そして、誕生した生命に試練を与えるかのように、地球レベルでの環境・気候変動が、過去から現在まで起こり続けている。一方で、生命は進化によって多様化すると共に、環境に適応する能力を獲得、与えられた試練を乗り越え、今日まで生存し続けてきた。一般的に進化といえば “人類の進化” といった、巨視的な視点で捉えられることが多い。しかし、進化の実体は、生体内に存在する生命の設計図である遺伝子の改訂の歴史であり、DNA、RNA、タンパク質・酵素などの生体高分子の進化とも言える。このような微視的な視点から見た生体高分子の進化は、文字通り分子進化と呼ばれている。本稿では生体高分子の中でも、特に酵素の分子進化について扱いたい。
酵素は生物の代謝を支える生体触媒であり、触媒する反応の違い等によって現在6000程度のグループに分類されている1)。この6000という多様性は、40億年の分子進化によって取捨選択された結果であり、その過程においてはより多様な酵素が存在していた可能性がある。酵素の祖先、いわゆる祖先型酵素に魅せられた多くの研究グループが、祖先型酵素の復元を試み、どのような反応を触媒し、どのような機能を有しているのか、機能解析を実施してきた。祖先型酵素を復元するには、祖先型設計法 (ASR法) が広く用いられている。ASR法では研究対象となる酵素の類似配列を取得し、マルチプル―シークエンスアラインメント (MSA) と系統解析により系統樹を作成、MSAと系統樹のデータをASR用のソフトウェア (FastML2), PAML3)など) で解析することで、系統樹上の祖先型酵素を復元することが可能となる4)。今日までの研究から、祖先型酵素は現存型酵素と比べて基質選択性が拡張し、多様な基質と反応できるようになる 5)、あるいは耐熱性が向上するといった報告がされてきた6)。これらの結果から生物の起源を考察した報告もなされている7)。また高機能化した祖先型酵素を産業応用につなげていこうとする研究も実施されつつある4)。ただし、高機能化に成功した例は多くはないのが現状である。
ASR法で復元した祖先型酵素を研究対象とした、酵素の分子進化解析は、基礎のみならず応用的視点から鑑みても魅力的な研究テーマである。しかし、ASR法を様々な酵素に適用するためには、解決すべき課題が幾つか存在する。中でも研究対象とする酵素の類似配列をどこまで利用してASR法に適用するのか、といった配列選抜の手法に関しては、設計の成否を左右する重要な部分であるにも関わらず、報告例が少ない。ASR法で設計予定の酵素に関する豊富なデータ (立体構造や変異体の活性データ) が存在すれば、それらデータを参照して配列を選抜することが可能であり、事実、β-ラクタマーゼやキナーゼなどの一部の酵素においてASR法が成功している。しかし、データが少ない新規な酵素では選抜の指針が無い。ASR法を広く酵素の設計に適用するためには、新規配列選抜法の開発及び実証研究が必要である。
そこで本稿では、まず我々が開発している新規配列分類法についてL-アルギニン酸化酵素 (AROD, EC 1.4.3.25) に適用し、ARODの祖先型酵素を設計した例について紹介する。ARODは様々なサンプル中のL-アルギニン濃度定量への応用を目的とし、松井及び淺野博士によってスクリーニングにより新規に取得された酵素である8)。ARODは20種類のアミノ酸のうち、L-Argに対し高い特異性を示し、アミノ基 (主鎖) の酸化反応を触媒する酵素である9)。ARODはその新規性ゆえに機能既知の配列が一つだけ報告されているのみであり、立体構造はもちろんのこと、変異体の活性データも現在までに報告されていない9)。最後に応用を目的として、新しく開発された祖先型AROD (AcAROD) の機能解析を通して、ARODの分子進化に関する仮説を提唱する。
1.離散的モチーフ様配列に基づくAROD類似配列の分類
ARODの祖先型酵素を復元するため、図1で示した配列分類法を用いてARODの類似配列を選抜した。まずデータベースよりPseudomonas sp. TPU7192由来AROD (PtAROD) の配列を取得、これを鋳型配列としてBlastp解析を実施し、PtARODの類似配列を取得した。その後、PtARODの残基長と比較して極端に長い、または短い配列を除去するなど、解析のノイズとなる配列を独自のpythonスクリプトで除去、配列ライブラリとして以下の解析に用いた (図1)。その後PtARODと配列ライブラリから一定数選抜した配列からなるライブラリを作成した (手順3)。各配列ライブラリをアラインメントしたのち、PtARODの配列を基準として、15, 50, 332, 580位の保存性に注目し、ライブラリから選抜した (手順4)。この選抜に使用した残基群は、酵素の機能の違いを定義するアミノ酸残基の保存性のネットワーク (サブファミリーを定義するモチーフ様の配列群) が存在しており、そのような残基は一次配列上で離散的に分布しているという仮説に基づいて定義した。本稿ではこのような残基群を離散的モチーフ様配列と呼ぶこととする。離散的モチーフ様配列群のネットワークを同定・解析するためのアルゴリズムについては開発をほぼ完了しており、成功例も増えつつあるが、一般化するには実証例が少なく、公開に至っていない。いずれにせよARODの設計において、離散的モチーフ様配列のネットワークを分類するためには、経験則的な判断も加味し、15, 50, 332, 580位で選抜する事で十分であると判断した。手順4の選抜を通して、最終的にPtARODの類似配列、10個を取得した (図1)。なお、結果として、選抜した10配列はPtARODと比べて、60%以上の相同性を保持していた。なお開発した手法を既存の方法と比べるため、隠れマルコフモデルを用いたツール、HMMERを用いてAROD類似配列を同様に分類した。結果、HMMERを用いたものでは最終的に42配列が選抜されたが、そのうち30配列はPtARODと比べて配列相同性が35%を下回っていた。以上のことから、離散的モチーフ様配列に基づく分類法では、HMMERでは除去できなかった配列を除去可能であり、より厳密な配列の分類が可能であることが示唆された。
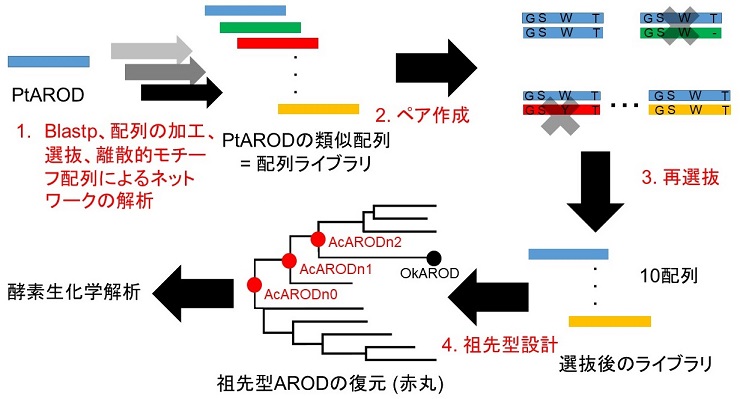
図1 新規配列選抜法および祖先型ARODの設計に関する概念図
次に選抜した10配列のうち、Oceanobacter kriegii由来AROD (OkAROD) を自然界由来ARODとして発現させた。また10配列を用いてFastMLを用いて祖先型設計を行い、系統樹の節に位置する祖先型ARODを3つ設計し、それぞれ発現させた (AcARODn0, AcARODn1、AcARODn2、手順5、図1)。なおAcARODn0が系統樹上、最も共通祖先配列に近く、AcARODn2が最も自然界由来AROD (OkAROD) に近い。以上自然界由来のARODと設計した3つの祖先型ARODについて、酵素生化学解析を実施した。
2.自然界由来および祖先型ARODの酵素生化学解析
4つのAROD (OkAROD, AcARODn2, AcARODn1, AcARODn0) は、いずれも大腸菌発現系を用いることで可溶性画分に容易に得ることができ、酵素生化学解析を実施可能な収量で精製サンプルを回収できた。まずはこれら4つのARODに関して、耐熱性を調べた。結果、OkARODが最もT1/2の値が低く (T1/2=65℃)、AcARODn2 (T1/2=72℃)、AcARODn1 (T1/2=78℃)、AcARODn0 (T1/2=88℃) の順で高くなることが判明した。これは共通祖先の配列に近づくにつれて、ARODの耐熱性が大幅に向上することを示しており、他の研究グループによる祖先型酵素・モデル蛋白質の研究結果と一致していた。次に20種類のL-アミノ酸を基質とし、ARODの基質特異性を調べた。結果、OkARODとAcARODn1、AcARODn2は、報告されているPtARODと同様、L-ArgとL-Lysに反応性を有することが判明した。また、AcARODn2はL-Lysと比較しL-Argに対するkcat/Km値が高く、L-Argの定量性に優れていることが示された。一方で、耐熱性が一番優れていたAcARODn0はこれら2つのL-アミノ酸に加え、5つのL-アミノ酸 (L-His, L-Phe, L-Leu, L-Met, L-Tyr) にも反応性を示すことが判明した。以上の結果は系統樹上の共通祖先配列に近づくにつれて、ARODの基質選択性が拡がることを示していた。以上の結果は図2にまとめている。
4つのARODに関して、基質選択性を示した棒グラフを図2に記載している。ここでAcARODn0の反応性に関して、報告されている他のL-アミノ酸酸化酵素 (LAAO) と比較したところ、Rhizoctonia solani由来のLAAO (RsLAAO, EC 1.4.3.2) と類似していることが判明した10)。AcARODn0が基質とする7つのL-アミノ酸は、RsLAAOによって酸化されることが示されている 。以上のことから、ARODの祖先はRsLAAOのような広い選択性を有するLAAOであった可能性が考えられる。

![]()
次に設計した4つの酵素の応用性を評価した。ARODは血中のL-Arg濃度定量への応用が期待されている酵素である9)。そこでヒト血漿中のL-Arg濃度定量の試験を実施した。結果、AcARODn0を除き、3つのARODに関してはL-Arg濃度定量が可能なことが示された。AcARODn0に関しては、おそらく血漿中の他のL-アミノ酸と反応したため、検量線が正しく描けず、L-Argの定量はできなかった。
3.考察
先にも記述したように、ASR法による祖先型酵素復元の成否は、設計に用いる配列の種類に強く依存することが報告されている11)。本稿では機能既知の配列データのみ利用可能なARODに関して、離散的モチーフ様配列のネットワーク解析により配列ライブラリを分類することで、多様な特性をもつ祖先型ARODを設計可能なことを示すことができた。このような分類はサブサンプリングと呼ばれている。一方で配列のサブサンプリングを実施すると、本来ASR法での設計に加えるべき配列を除去してしまい、祖先型残基を正しく予測できず、祖先型配列の機能を正確に評価できなくなるという懸念も存在する。事実、離散的モチーフ様配列に基づいてサブサンプリングすると、人の目で見て、本来なら除外すべきでない類似配列も除去されることが判明している。ARODのケースでも、そのような配列が少なからず含まれていた。一方でサブサンプリングにより、幾つか配列が除去された状況でASR法を実行しても、祖先型配列のフェノタイプにはほとんど影響が現れないという報告もある12,13)。本研究ではこの論文の情報を基に、本設計と解析が妥当であると解釈した。
次にARODの分子進化について考察する。酵素機能が進化の過程で変化する際、新たな機能を獲得するために、本来備わっていた酵素機能 (例えば耐熱性、可溶性、構造安定性など) が犠牲になることがある。これは酵素機能のトレードオフと呼ばれている14)。一方で進化の過程で導入される変異に対して頑強 (ロバスト) な酵素も存在しており、このような酵素の場合、変異導入によってもトレードオフが起こりにくくなることが報告されている。ASR法によって設計されたAcARODs群は、酵素としての優れた機能と高い耐熱性を併せ持っていた。つまり、変異に対する頑強性を有した設計ができれば、何らかの機能を獲得するために必ずしもトレードオフが生じず、超高機能な酵素を開発でき、産業利用上有用となる可能性を示唆している。今後の研究でAcARODの立体構造が解かれ、活性中心付近のアミノ酸残基に変異を導入した変異体の機能解析を通して、AcARODが変異に強い頑強な酵素であることを証明できると考えられる。本研究の進展によりAcARODの機能を改変し、新たな定量用酵素の開発が可能になると考えられる。
4.おわりに
ARODは設計の指針となる情報 (立体構造や変異体の活性など) が少ない新規酵素であったことから、ASRを適用するには難しい研究対象であった。このような困難さにも関わらず、ARODの分子進化をOkAROD及び復元した3つの祖先型AROD (AcARODn0, AcARODn1, AccARODn2) の酵素生化学解析を通して予測することに成功した。離散的モチーフ様配列のネットワーク解析により、ASR法の成功確率を上げることができ、その他のタンパク質の分子進化解析へも将来、広く適用できるのではないかと期待している。本研究の課題として、サブサンプリングが祖先型設計に及ぼす影響について、今後精査する必要があることも述べておく。一方で我々はタイムマシンを作れないため、分子進化を正確に予測できないことも理解しておく必要がある。またこのような計算機を用いた祖先型解析を実施する上で、新規酵素を微生物・生物からスクリーニングするプロセスが重要な点は忘れてはいけないことを記述しておく。最後に本研究についてARODの分子進化に関する一つの仮説を提唱したものと捉えていただければ幸いである15)。
謝辞
本研究はJSPS 科研費 (16K18688, 17K06931, 17H06169, 18K14391) とJST ERATO (JPMJER1102) の支援を受けて実施された研究です。また富山県立大学の松井 大亮博士にはARODの酵素機能解析に関して助言をいただきました。この場を借りて御礼申し上げます。
文献
1) Donertas, H. M., Martinez Cuesta, S., Rahman, S. A., Thornton, J. M.: PLoS One, 11, e0147952 (2016).
2) Pupko, T., Pe'er, I., Shamir, R., Graur, D.: Mol. Biol. Evol., 17, 890-896 (2000).
3) Yang, Z.: Mol. Biol. Evol., 24, 1586-1591 (2007).
4) Gumulya, Y., Gillam, E. M.: Biochem. J., 474, 1-19 (2017).
5) Devamani, T., Rauwerdink, A. M., Lunzer, M., Jones, B. J., Mooney, J. L., Tan, M. A., Zhang, Z. J., Xu, J. H., Dean, A. M., Kazlauskas, R. J.: J. Am. Chem. Soc., 138, 1046-1056 (2016).
6) Nguyen, V., Wilson, C., Hoemberger, M., Stiller, J. B., Agafonov, R. V., Kutter, S., English, J., Theobald, D. L., Kern, D.: Science, 355, 289-294 (2017).
7) Akanuma, S., Nakajima, Y., Yokobori, S., Kimura, M., Nemoto, N., Mase, T., Miyazono, K., Tanokura, M., Yamagishi, A.: Proc. Natl. Acad. Sci. U.S.A., 110, 11067-11072 (2013).
8) 淺野泰久, 松井大亮: 新規L-アルギニン酸化酵素, L-アルギニンの測定方法, L-アルギニンの測定キット及びL-アルギニン測定用の酵素センサ. 特許6130161 (2017).
9) Matsui, D., Terai, A., Asano, Y.: Enzyme Microb. Technol., 82, 151-157 (2016).
10) Hahn, K., Neumeister, K., Mix, A., Kottke, T., Groger, H., Fischer von Mollard, G.: Appl. Microbiol. Biotechnol., 101, 2853-2864 (2017).
11) Merkl, R., Sterner, R.: Biol. Chem., 397, 1-21 (2016).
12) Randall, R. N., Radford, C. E., Roof, K. A., Natarajan, D. K., Gaucher, E. A.: Nat. Commun., 7, 12847 (2016).
13) Rosenberg, M. S., Kumar, S.: Proc. Natl. Acad. Sci. U.S.A., 98, 10751-10756 (2001).
14) Khersonsky, O., Tawfik, D. S.: Annu. Rev. Biochem., 79, 471-505 (2010).
15) Nakano, S., Niwa, M., Asano, Y., Ito, S.: Appl. Environ. Microbiol., 85, e00459-00419 (2019).