【トピックス】
蛍光タンパク質をベースとした新規酸素センサータンパク質の開発
野亦次郎、久堀 徹
東工大・化生研、JST・CREST
1.はじめに
酸素 (O2) は生物の生命活動を支える重要なガス分子である。酸素は、呼吸や免疫、クロロフィルの生合成やヘムの分解など、生命活動の根幹となる代謝に必須であり、近年では生体内でシグナル分子としてはたらくことも明らかになってきた1,2)。すなわち、生体内の酸素濃度は、酸素に依存したさまざまな代謝に直接影響を与える要因と言える。従って、生細胞内の酸素動態を知ることは、多様な生命現象の理解へとつながる重要な情報を与えるはずである。しかし、生物の細胞内における酸素濃度や酸素動態に関する知見は今日でも極めて限定的であり、細胞内酸素濃度を簡便に測定できる技術の開発が求められている。そこで、本稿では、我々が蛍光タンパク質をベースとして新たに開発した酸素センサープローブを紹介する。
2.新規酸素センサープローブの開発
2-1 既存の酸素測定技術
我々が酸素センサーの開発に着手する以前から、すでに細胞内の酸素濃度を測定する技術そのものは開発されていた。例えば、細胞に微細な電極を突き刺して直接酸素濃度を測定するクラーク型酸素電極3)や、酸素感受性トリチルラジカル分子の電子常時性共鳴 (EPR) を利用した測定法4,5) (EPR oximetry)、燐光の酸素による特異的な消光を原理とする金属−ポルフィリン錯体化合物などの燐光プローブを用いた測定法などである6)。特に、燐光プローブを用いた測定は非破壊的な方法として有効であり、生物組織内の酸素濃度の測定に成功しているが7,8)、大掛かりな測定装置を必要とするなどの問題点もある。近年では、タンパク質を利用したバイオセンサーも開発され、生細胞内の酸素動態が調べられている9,10)。これらのバイオセンサーは、共通して黄色蛍光タンパク質YFPとFMN結合性蛍光タンパク質 (FbFP) を組み合わせてデザインされた融合タンパク質である。この融合タンパク質中のYFPの蛍光団は酸素依存的な化学反応により形成されるため、酸素が存在するとYFPに由来する蛍光の強度が継時的に増加する。一方で、FbFPは酸素に依存せず一定強度の蛍光を示すので、酸素の存在は蛍光強度比 (YFP/FbFP) の変化として検出することができる。しかし、YFPの蛍光団形成は不可逆反応であるため、この方法で生体内の酸素動態 (特に、酸素の減少) を調べることは原理的に困難である。
2-2 新規酸素センサー蛍光タンパク質の開発
そこで、我々が目指したのは、非侵襲的に、難しい光学系を組む必要のない、誰もが簡便に細胞内酸素濃度を測定できる技術の開発であった。そのため、蛍光タンパク質をベースとした遺伝子コード型のバイオセンサーを新規にデザインすることにした。我々が着目したのは、大腸菌が持つ天然の酸素センサータンパク質DosP (Direct oxygen sensor protein) である。DosPはヘムを含むタンパク質で、環境中の酸素濃度にあわせて分子状酸素を可逆的に結合・解離する性質がある11-13)。このDosPのヘム結合ドメイン (DosH) の結晶構造はすでに明らかにされており、酸素結合型と酸素非結合型の構造情報が利用可能であった14,15)。
これまで報告されているFRET (fluorescence resonance energy transfer; 蛍光エネルギー共鳴移動) 型バイオセンサーを見てみると、リガンドの結合 (解離) に伴うタンパク質分子の構造変化を作動原理として利用して、蛍光シグナルの変化を検出するものがほとんどである16,17)。しかし、DosHの構造を詳細に眺めても、酸素の結合・解離による大きな構造変化は見られなかったので、このタンパク質をバイオセンサーに利用するためには、新たな作動原理を探さなければならなかった。そこで着目したのは、蛍光の消光 (fluorescence quenching) 現象である18)。DosHはヘムに由来する複数の吸収帯を示すが、その中で最も長波長側にあるQyピークはDosHが酸素を結合すると減少 (かつ長波長シフト) する19)。従って、DosHと蛍光タンパク質を組み合わせれば、DosHのQyピークの増減に伴って、近接する蛍光タンパク質が受ける蛍光の消光の度合いも増減し、その結果、酸素の有無により蛍光強度が変化することが期待された。
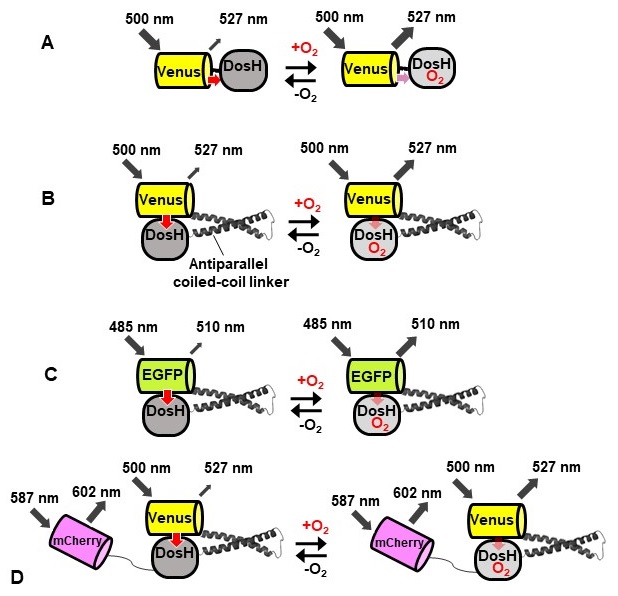

表1 protoANAおよびANAの蛍光寿命

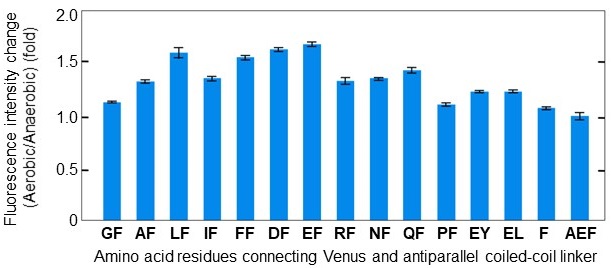
我々は、手始めにDosHと黄色蛍光タンパク質 (Venus) を短いリンカー (Gly-Ser)、および、逆平行リンカー20) (antiparallel coiled-coil linker) で接続した2種類の融合タンパク質をデザインした (protoANA1, protoANA2: prototype anuaerobic/aerobic sensing fluorescence protein; 図1A,B)。それぞれの組み換えタンパク質を精製して、蛍光強度の変化を測定すると、酸素の添加により蛍光強度が約10%増加することがわかった。各タンパク質の蛍光寿命を測定すると、protoANA1は酸素添加の前後で2.55 nsと2.62 ns、protoANA2では1.46 nsと1.60 nsという値が得られ (表1)、いずれの場合も酸素添加による蛍光寿命の増加が観察された。すなわち、DosHへの酸素結合によって、Venusが受ける蛍光の消光の度合いが変化することが示唆された。酸素の結合による蛍光寿命の変化を比較すると、protoANA2の方がわずかに大きな変化を示したため、以降はprotoANA2を対象として改良を行った。ptotoANA2の蛍光変化率をさらに大きくするため、逆平行リンカーとVenusの間の2つのアミノ酸残基を、電荷や疎水性、かさ高さの異なるさまざまなアミノ酸残基に置換して解析をおこなった (図2)。その結果、Glu-Pheという組み合わせが最も高い蛍光変化率を示したので (1.67倍; EF)、このタンパク質をANA-yellow (ANA-Y) と名付けた21,22)。引き続き、接続に用いる残基の数も調べてみたが、1つ、あるいは、3つにしても変化率はほとんど改善されなかった。


2-3 酸素センサータンパク質ANA-Yのin vitro解析
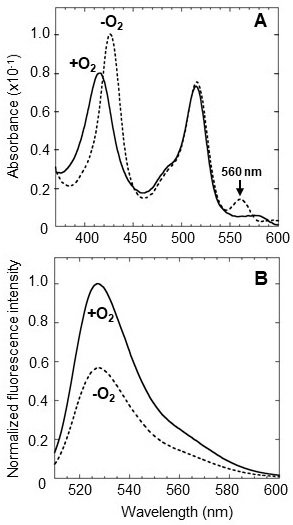
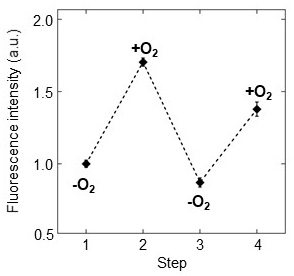
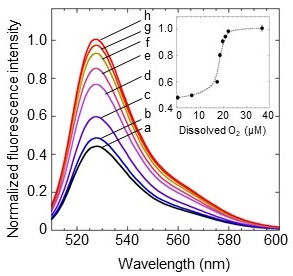
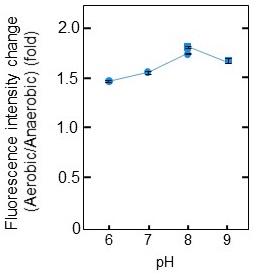
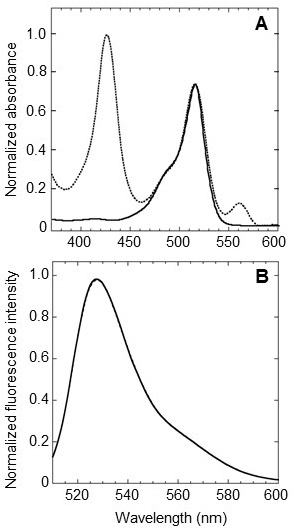
ANA-Yの分光学的性質を調べるため、精製したANA-Yの吸収スペクトル、蛍光スペクトルおよび蛍光寿命の測定を行った。嫌気条件下におけるANA-Yの吸収スペクトルには、酸素を結合していないヘムの特徴を表す425 nm (Soret帯) と560 nm (Qy) のピーク、および、Venusに由来する515 nmのピークが見られた (図3A、破線)。ここに酸素を添加すると、Soret帯およびQyピークがそれぞれ418 nm、580 nmへとシフトし、ヘム-酸素分子複合体の速やかな形成が示唆された (図3A、実線)。蛍光スペクトルを測定すると、酸素添加によりVenusに由来する527 nmの蛍光ピーク強度が約1.7倍に増加し、期待した変化が起こることが確認できた (図3B)。次に、ANA-Yの蛍光寿命を測定すると、酸素添加の前後で1.00 nsと1.55 nsという値が得られ (表1)、蛍光強度および蛍光寿命のいずれにおいても、protoANA2より変化率が大きくなったことを確認することができた。次に、ANA-Yの酸素応答の可逆性を調べたところ、酸素の添加・除去の繰り返しに応じた蛍光強度の可逆的な変化を確認することができた (図4)。前述したとおり、既存の遺伝子コード型酸素センサーは不可逆的であり9,10)、ANA-Yは初めての可逆的なバイオセンサーであることが示された。次に、ANA-Yの酸素検出感度を調べるため、様々な濃度の酸素存在下で蛍光強度を測定した (図5)。その結果、ANA-Yは6μM以上の酸素を検出可能であり、酸素に対する半飽和値は18μMと算出され (図5、inset)、報告されているDosHの酸素半飽和値とほぼ同等であった12,13,19)。次に、ANA-YのpH感受性を調べるため、pH 6.0〜9.0の各条件下でANA-Yの酸素添加による蛍光強度変化を測定した。その結果、pH 6.0において1.5倍、pH 8.0において1.7倍の蛍光変化率を示すなど、わずかなpH感受性を示したが、pH 6.0〜9.0の範囲においては安定に機能することがわかった (図6)。



2-4 色違いおよびレシオメトリックタイプの酸素センサータンパク質の開発
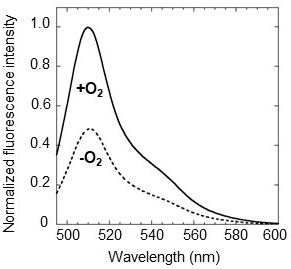
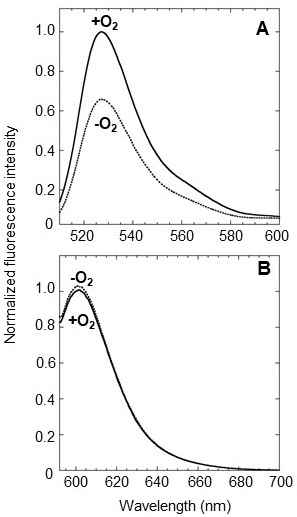
さらに、我々は色違いの酸素センサーを開発するため、ANA-Yの分子デザインをDosHと緑色蛍光タンパク質EGFPの組み合わせにも適用し、ANA-green (ANA-G) と名付けた新たなセンサーを作製した (図1C)。組み換えタンパク質を調製し、蛍光スペクトルを測定すると、酸素添加によりEGFPに由来する510 nmの蛍光強度が約2.1倍に増加した (図7)。さらに、細胞内酸素濃度の定量を可能にするため、レシオメトリックタイプ (2つの蛍光ピークの比として酸素濃度をモニターできる) のセンサーをデザインした。このタンパク質は、ANA-Yに赤色蛍光タンパク質mCherryを連結したもので、これをANA-Q (ANA-quantity) と名付けた (図1D)。ANA-Qの組み換えタンパク質はVenusに由来する527 nmの蛍光ピークに加え (図8A)、mCherryに由来する602 nmの蛍光ピークを示す (図8B)。Venusのピークは酸素濃度に応じて変化するが、mCherryのピークは一定であるため、これらふたつのピークの比 (F527/F602) を求めることにより、細胞内のセンサータンパク質の発現量に依存せずに酸素濃度を定量することができる。このセンサーでは、酸素添加によりF527/F602比率が約1.6倍に上昇した。


2-5 ANAを利用した光合成による酸素発生のモニタリング
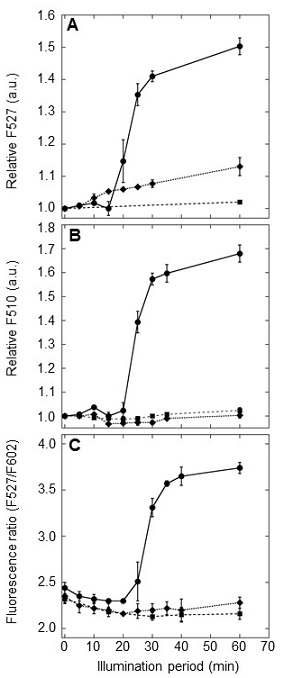
このようにして作製したANAタンパク質を利用して、シアノバクテリアの光合成による微量の酸素発生をin situでモニターすることを試みた。嫌気条件下でキュベットに密閉したANA-Y、ANA-G、ANA-QとシアノバクテリアAnabaena sp. PCC7120 (A.7120) 細胞の混合液に光を照射し続け、ANAの蛍光変化を継時的に測定した。光照射20分後から蛍光強度が増加し、60分後には飽和に達した (図9、●)。一方、光合成の阻害剤であるDCMU (diuron 3- (3,4-dichlorophenyl) -1,1-dimethylurea) の存在下では、光を照射してもANAの蛍光強度はわずかに増加するのみで (図9、◆)、in situでANAを利用して光合成による酸素発生を高い感度で検出し経時的変化をモニターすることに成功した。

2-6 ANAのヘム依存性
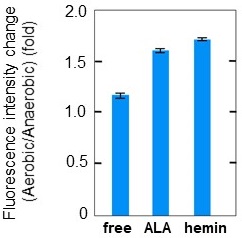
前述の通り、ANAはヘム分子による蛍光の消光を作動原理としているので、ANAが機能するためにはヘムが必須である。そこで、ANA-Yの酸素依存的な蛍光強度変化がヘムに依存することを確認するため、ヘムを結合するHis348残基をAla残基へと置換した変異型ANA-Y (H348A) を作製した。精製したH348Aタンパク質の吸収スペクトルを測定したところ、ヘムに由来するピークは見られず (図10A)、酸素を添加しても蛍光強度は変化しなかった (図10B)。このように、ANAの機能にはヘムが必須であることから、ANAをin vivoで使用する際には、対象とする細胞内のヘム量に注意を払う必要がある。大腸菌を用いてヘムの必要性を検討したところ、LB培地で培養したANA-Y発現株から抽出、精製したANA-Yの蛍光変化率は1.2倍であったが、精製の過程でヘムを添加して補った場合には1.73倍の値が得られた (図11、free, hemin)。この結果は、大腸菌細胞に内在するヘムだけでは、細胞内に発現するすべてのANA-Y分子がヘムを結合した状態では得られないことを示している。そこで、細胞内のヘム含量を増加させるため、ヘム生合成の前駆体である5-アミノレブリン酸 (ALA) を用いてヘム生合成の増強を試みた。終濃度300μMとなるようにALAを添加したLB培地でANA-Y発現株を培養し、この細胞からANA-Yを精製して蛍光変化率を調べたところ、1.63倍 (図11、ALA) という値が得られ、精製過程でヘムを補った場合の変化率 (1.73倍) に近い値を示した。従って、ANAを利用してin vivoでの観察を行う際には、培地などにALAを添加して細胞内のヘム含量を十分高めることが推奨される。


3.おわりに
本稿では、蛍光タンパク質をベースに開発した新規酸素センサータンパク質ANAを紹介した。ANAは、構造変化を作動原理とする従来のバイオセンサーとは異なり、蛍光の消光を作動原理として機能する新しいセンサータンパク質である。天然のセンサータンパク質の中には、我々が活用したDosHのように、基質 (エフェクター) 分子が結合しても大きな構造変化は示さないが、吸収スペクトル変化を起こすタンパク質が数多く存在する。これらのタンパク質の性質を利用すれば、今後、蛍光の消光を作動原理とする新たなセンサータンパク質の開発に可能性を広げることができるものと期待している。
謝辞
蛍光寿命の測定・解析は、彌田智一教授 (現・同志社大学)、神戸純子氏 (東京工業大学)、鈴木元也氏 (東京工業大学すずかけ台分析部門) の協力を得て行われました。また、生物学、工学、化学など専門を異にする多くの方々に助言を頂きました。この場を借りて厚く御礼申し上げます。本研究はJST・CRESTおよびJSPS科研費(18K06150)による支援を受けて行われました。
文献
1) Shimizu, T., Huang, D., Yan, F., Stranava, M., Bartosova, M., Fojtikova, V., Martinkova, M.: Chem. Rev., 115, 6491 (2015).
2) Kosmacz, M., Weits, D. A., Giuntoli, B., Giorgi, F. M., Voesenek, L. A., Perata, P., van Dongen, J. T.: Nature, 479, 419 (2011).
3) Clark, L. C. Jr., Wolf, R., Granger, D., Taylor, Z.: J. Appl. Physiol., 6, 189 (1953).
4) Bobko, A. A., Dhimitruka, I., Eubank, T. D., Marsh, C. B., Zweier, J. L., Khramtsov, V. V.: Free Radic. Biol. Med., 47, 654 (2009).
5) Liu, Y., Villamena, F. A., Sun, J., Wang, T. Y., Zweier, J. L.: Free Radic. Biol. Med., 46, 876 (2009).
6) Akiyama, H., Takahashi, I., Shimoda, Y., Mukai, R., Yoshihara, T., Tobita, S.: Photochem. Photobiol. Sci., 17, 846 (2018).
7) Sakadzic, S., Roussakis, E., Yaseen, M. A., Mandeville, E. T., Srinivasan, V. J., Arai, K., Ruvinskaya, S., Devor, A., Lo, E. H., Vinogradov, S. A., Boas D. A.: Nat. Methods, 7, 755 (2010).
8) Spencer, J. A., Ferraro, F., Roussakis, E., Klein, A., Wu, J., Runnels, J. M., Zaher, W., Mortensen, L. J., Alt, C., Turcotte, R., Yusuf, R., Cote, D., Vinogradov, S. A., Scadden, D. T., Lin, C. P.: Nature, 508, 269 (2014).
9) Potzkei, J., Kunze, M., Drepper, T., Gensch, T., Jaeger, K. E., Buchs, J.: BMC Biol., 10, 28 (2012).
10) Eichhof, I., Ernst, J. F.: Fungal genet. Biol., 92, 14 (2016).
11) Tuckerman, J. R., Gonzalez, G., Sousa, E. H., Wan, X., Saito, J. A., Alam, M., Gilles-Gonzalez, M. A.: Biochemistry, 48, 9764 (2009).
12) Delgado-Nixon, V. M., Gonzalez, G., Gilles-Gonzalez, M. A.: Biochemistry, 39, 2685 (2000).
13) Taguchi, S., Matsui, T., Igarashi, J., Sasakura, Y., Araki, Y., Ito, O., Sugiyama, S., Sagami, I., Shimizu, T.: J. Biol. Chem., 279, 3340 (2004).
14) Park, H., Suquet, C., Satterlee, J. D., Kang, C.: Biochemistry, 43, 2738 (2004).
15) Park, H., Suquet, C., Savenkova, M. I., Satterlee, J. D., Kang, C.: Acta. Crystallogr. sect. D Biol. Crystallogr., 58, 1504 (2002).
16) Miyawaki, A. Llopis, J., Heim, R., McCaffery, J. M., Adams, J. A., Ikura, M., Tsien, R. Y.: Nature, 388, 882 (1997).
17) Imamura, H., Nhat, K. P., Togawa, H., Saito, K., Iino, R., Kato-Yamada, Y., Nagai, T., Noji, H.: Proc. Natl. Acad. Sci. USA, 106, 15651 (2009).
18) Mahajan, P. G., Bhopate, D. P., Kamble, A. A., Dalavi, D. K., Kolekar, G. B., Patil, S. R.: Anal. Meth., 7, 7889 (2015).
19) Ishitsuka, Y., Araki, Y., Tanaka, A., Igarashi, J., Ito, O., Shimizu, T.: Biochemistry, 47, 8874 (2008).
20) Shekhawat, S. S., Porter, J. R., Sriprasad, A., Ghosh, I.: J. Am. Chem. Soc., 131, 15284 (2009).
21) Nomata, J., Hisabori, T.: Sci. Rep., 8,11849 (2018).
22) Nomata, J., Hisabori, T.: Patent Application, Japan, 2017-040778.