【トピックス】
生体膜内でのペプチドの挙動の分子動力学シミュレーション
上田一義、内藤 晶、川村 出、Namsrai Javkhlantugs
横浜国大院・理工、横浜国大院・理工、横浜国大院・理工、モンゴル国立大・化学
1.はじめに
近年、コンピューターの進歩は目覚ましく、それに伴い、計算化学の進展も著しい。もちろん、コンピューターのハードの進歩だけではなく、計算の方法論も飛躍的に進歩している。本稿で述べる分子動力学シミュレーション法について言えば、数万原子からなる系で数百ナノ秒の現象をシミュレーションすることは、今やパソコンレベルで日常的に計算することが可能なレベルに達している。もし、スパコンを使うのであれば、約一億原子の細胞丸ごとのシミュレーション1,2)や、秒のオーダーの長時間シミュレーションを行うことすら可能である。本稿のテーマである、生体膜中のペプチドの挙動に関するシミュレーションに関しても、数多くの研究が行われており多くの成果があげられている3,4)。しかし、モデル系の大きさ、シミュレーション時間の長さの両面で充実した計算が可能になってきたとは言っても、1023オーダーの原子数からなる実験系や、反応・平衡まで数分〜数時間かかる実験系をそのまま、丸ごとシミュレーションするような計算規模は現状では実現できない。そこで、重要なのは系―時間を含めたモデルをデザインすることである。計算を行うモデルの規模や観測時間の長さを工夫すれば、例えば実験的には立証しきれない生体膜中でのペプチドの挙動を明らかにすることが可能で、実験の補完手段として十分な実用性をもたらすことが出来る。本稿では、量子化学計算など様々ある計算機化学の方法のうち、分子動力学シミュレーション法について我々の行った脂質二分子膜中のペプチドの挙動に関するシミュレーション結果を述べる。
2.Bombolitin IIの膜中での構造と配向
Bombolitinはマルハナバチの蜂毒から抽出された17残基のアミノ酸からなる両新媒性ペプチドの一群で、溶血性などを示す。内藤らは5)、細胞膜の破壊機構を明らかにするためにBombolitin II (BLT2) を用いて膜に結合した構造や相互作用を固体NMRの手法を用いて精査した。生体膜を模倣した脂質二分子膜として、中性膜である1,2-dipalmitoyl -sn-glycero-3-phosphocholine (DPPC) を用い、そのベシクルを静磁場中で磁場配向させて、固体NMRスペクトルを観測した。その結果、BLT2はDPPC膜中でα-helix構造を形成し、helix軸が二分子膜面に対し、垂直ではなく膜法線に対し約33°傾いて貫入していることを明らかにした。この構造が、BLT2ペプチドの溶血性と関連していると推定されたが、なぜ膜中で垂直ではなく傾いて存在し、それにどのような意味があるのかについては明らかではなかった。さらにBLT2の場合、helix構造の軸長が二分子膜の膜厚より短く膜を貫通することができないため、傾斜の原因究明とともに膜中での位置の特定にも興味がもたれた。そこで、分子動力学シミュレーションによりBLT2の膜中での存在状態について検討した6)。計算用のプログラムとしてはcharmm7)を用いた。モデル系としては、一分子のBLT2を70個のDPPC分子中に膜面に対し垂直に入れ、DPPC膜外には水分子と対イオンを入れた。全原子数は約15,000個である。現在の計算機水準から見るとやや小さめのサイズのモデルであるが、シミュレーションの目的からはこれで十分妥当な大きさであった。すなわち、図1(a) のように、ペプチドC末端を膜内に貫入させ、N末端を膜表面付近に配置した構造を初期状態として約20nsの熱運動を行ったところ、15ns程度で、図1(b) に示すような傾いた配向での安定な構造をとることを観測することができた。シミュレーションの温度設定はDPPCのゲル-液晶転移温度 (315 K) より高い323Kで行っている。また、実験ではペプチドがN末端、またはC末端のどちらから貫入するのかも不明であったため、シミュレーションではN末端を内側にしたモデルについても試行した。さらに、膜中での位置の検討のため、BLT2を膜の中心部に配置したシミュレーションも行った。中心部に配置したシミュレーションでは、N末端が直ちに膜表面方向に向かって垂直に移動した。これはプロトン化されたN末端のSer1やLys2など、正の電荷を帯びたN末端側の残基が膜分子の負の電荷を持つ脂質頭部のリン酸基と強い静電相互作用をすることによるものであった。しかし、このシミュレーションではC末端部分はN末端の移動に追い付かず、結果的にペプチドのhelix構造がほどけて伸びた構造となってしまった。一方、N末端を膜内に配向させたシミュレーションにおいても、電荷を帯びたN末端が膜表面に移動しようとして、結果的にhelix構造が崩壊してしまった。これらの結果は選んだ初期構造がペプチドの膜中での平衡構造から大きく外れた構造であったことを示している。もちろん、これらの初期構造からでも非常に長時間のシミュレーションを行えば、図1(b) のような平衡構造を観測することが出来るはずである。しかしながら、この結果は良い初期構造 (モデル) を使うと、小規模なシミュレーションでも十分に現象の再現が可能であることを示唆している。
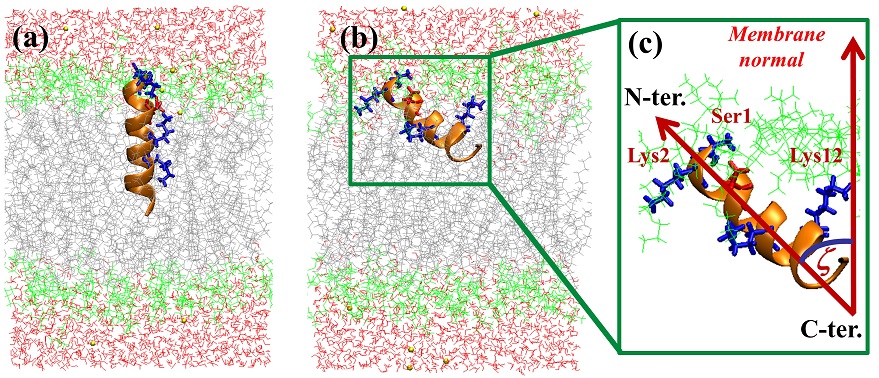

さて、ペプチドが傾くメカニズムであるが、図1(c) を見るとN末端は膜表面に位置する脂質親水基部分に存在し、他方のC末端ではLys12の側鎖が膜表面方向に延びている。詳しく解析した結果、膜内部に位置するLys12側鎖と脂質分子の親水部リン酸基が静電相互作用で引き合っていることに加えて、Lys12の2個のNHと脂質分子中のパルミチン酸アシル鎖部分の膜表面に近い部位にある2個のC=O基が水素結合を形成していることが分かった。これらの相互作用がLys12の側鎖を通してペプチドhelix軸を斜めに引き上げていることが判明した。実験を実施するグループとの緊密な連携でシミュレーションに適したモデルを作成することは、良い結果を得るための重要かつ不可欠な条件である。
3.Bovine lactoferrampinの膜中での構造と配向
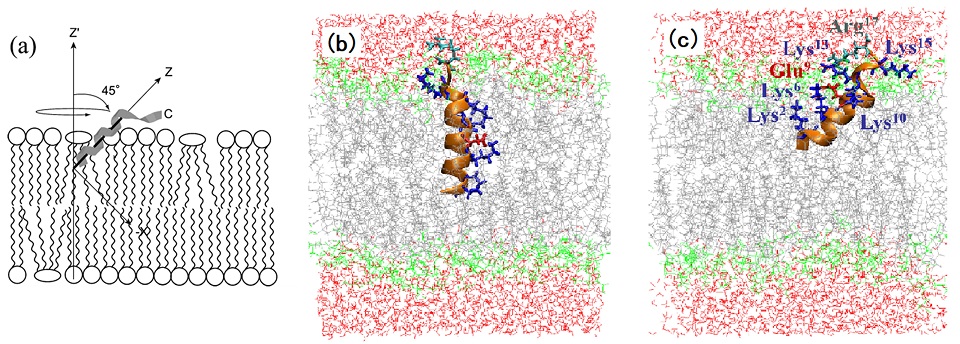
Bovine lactoferrmpin (LFampinB) は牛の母乳に含まれる鉄輸送タンパク質lactoferrinのN1ドメインから見出された抗菌性ペプチドで、前出のBLT2と同じく17残基のアミノ酸からなる。Lactoferrinも抗菌活性を示すが、LFampinBはより強い抗菌活性を示す。このペプチドの抗菌活性機構を解明するため、我々は固体NMR法と分子動力学シミュレーション法を組み合わせてLFampinBの構造と菌体膜との相互作用についての検討を行った8)。NMR実験では、菌体模倣膜として酸性脂質であるDimyristoylphosphatidyl glycerol (DMPG) を主成分とした、DMPG : dimyristoylphosphatidyl choline (DMPC) : cardiolipin (CL) = 65:25:10 (w/w) の成分からなる混合膜を用いた。その結果、LFampinBは模倣膜中で、N末端からLys10付近までα-helix構造を形成していることが確認された。これはPhe11まではα-helix構造を形成しているというHaneyら9)の結果と合致した。さらに、固体NMRの解析からは、N末端が膜内部に膜法線に対し45°の角度で貫入しつつC末端は膜表面と水の界面に接するような状態で相互作用していることが推定された (図2(a))。この構造を確認するとともに原子レベルでどのような相互作用が働いているかを調べるために分子動力学シミュレーションを行った。その初期構造が図2(b) である。固体NMRで求めた図2(a) の構造を参考にN末端を膜内に貫入させ、BLT2の場合と同様にhelix軸を膜面に垂直に貫入させた配向を初期構造とした。シミュレーションの方法はBLT2の場合と同様で、温度は313 Kに設定した。構造はシミュレーションの時間の進行と共にC末端が膜面方向に浮き上がり、Lys13, Lys15, Arg17は膜表面で水と接触しはじめた。一方、膜内にとどまったN末端のhelix部分は傾いた配向となった。図2(c) に平衡に達した際 (10ns後) の構造を示す。一見して、固体NMRにより推定された構造 (図2(a)) を良く再現していることがわかる。詳細に構造を見ると、Lys2, Lys6, Glu9とLys10が側鎖を膜表面に向けて親水基部分が膜分子頭部のphosphatidyl glycerol部分と相互作用している様子がわかる。この相互作用によりhelix部が膜平面方向に引き上げられてらせん軸が斜めに傾くことになる。傾いたhelixの膜中での運動は膜構造の破壊を引き起こすと推定される。

4.Melittinの膜中での構造と配向10)
Melittinはミツバチ、Apis melliferaの毒の主成分であり、生体膜や中性の人工脂質二重膜を破壊するとともに、ホスフォリパーゼ A2によるリン脂質の加水分解を促進する。酸性膜も破壊するが、その能力は中性膜に比べると低いことが知られている。しかし、菌体膜は一種の酸性膜であるため、酸性膜中での挙動を調べることは抗菌性の機構を理解するうえでは重要である。そこで、前二例と同様に酸性膜であるDMPGを用い、その二重膜に結合したmelittinの構造および配向を調べ、酸性リン脂質との相互作用の詳細を明らかにした。また、このペプチドもα-helixを基本構造とすると言われているが、アミノ酸残基数は26であり、前二例と比べると少し長く、配向や相互作用に及ぼす影響の比較は興味深い。
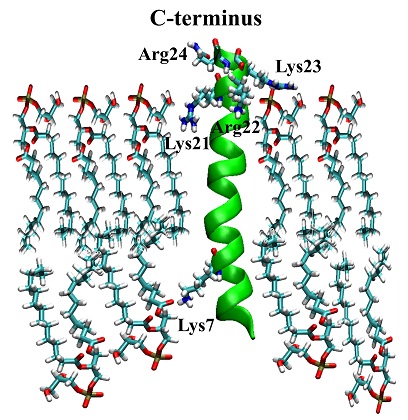
DMPG-melittin試料の13C-NMR測定を行い、melittinのN末端側およびC末端側のα-helixを構成するアミノ酸残基 (13C標識) の化学シフト異方性の挙動を解析した。その結果、DMPG二重膜に結合したmelittinのN末端側のα-helixは-32°、C末端側のα-helixは30°それぞれDMPG二重膜の膜法線に対し傾いた状態で、折れ曲がって存在していることが分かった。C末端側には電荷を持つ酸性アミノ酸が多く含まれ、膜の界面に存在すると思われる。そのため、helixが折れ曲がっているとすると、N末端は膜の親水部までは達せず、疎水性コア部分に存在して、擬-膜貫通型の配向をしていることが推察された。その状態を詳細に検討するために、分子動力学シミュレーションにより、DMPGに結合したmelittinの構造と配向を計算した。初期構造としては折れ曲がりのないα-helix構造を採用し、前二例と同様に膜面に垂直に貫入させた初期配置とした。ゲル-液晶転移温度 (Tc=296 K) より高い、温度313 Kで30nsの分子シミュレーションを行った結果、図3のようなmelittinの構造と配向となった。固体NMRの結果を裏付けるように、C末端側のα-helixは先端部がDMPG二重膜から突出していた。また、N末端側のα-helixは先端部がリン脂質二重膜表面まで届かず、逆に、melittin のN末端近傍にあるリン脂質分子を膜内部に引き込んでいるような構造となっていることが分かった。この引き込みにはmelittin のN末端部のLys7とDMPG二重膜頭部のリン酸基の持つ負電荷との相互作用が関与していることも明らかとなった。一方、C末端側のα-helixではLys21-Arg22-Lys23-Arg24に起因する4つの正電荷がDMPG二重膜の頭部親水性部分と同一平面上に位置し、melittinと膜の間には強い静電相互作用が働いていることも判明した。中性膜であるDMPC中に存在するmelittinは二重膜構造を容易に破壊するものの、DMPGのような酸性膜の中に存在するときはTc以下でも二重膜構造を破壊しにくいことが知られている。酸性膜に対する作用の弱さには、melittin分子のC末端部と酸性膜の間の静電相互作用による膜構造の安定化が関与していると思われる。
![]()
5.おわりに
本稿では、蜂毒ペプチドであるBombolitin, Melittinと、抗菌ペプチドのBovine Lactoferrampinを題材として、固体NMR測定と分子動力学シミュレーションの協働の仕方とそれらの結果を概説した。これらの他にもκ-Opioid recepterとOpioidペプチドのDynorphinについてもDMPC膜中での相互作用を固体NMR測定と分子動力学シミュレーションを組み合わせて検討している11)。今回は、我々のグループの成果についての紹介にとどめているが、同様の取り組みは増加の傾向にある。生体膜や菌体膜における生体高分子の構造及び相互作用の情報はバイオ、創薬、医療の分野においてますます重要となっており、その知見を得るための方法として、固体NMR測定の実験と分子シミュレーションを組み合わせた手法はさらに進展するものと思われる。
6.謝辞
本研究を実施するにあたり、本稿で取り上げた論文の共同研究者の方々には大変お世話になりました。厚く御礼申し上げます。また、本稿執筆の機会を与えて頂きました、横浜国立大学大学院工学研究院の武田穣教授に心より感謝申し上げます。
文献
1) Yu, I., Mori, T., Ando, T., Harada, R., Jung, J., Sugita, Y., Feig, M.: eLife. 2016: 5: e19274.
2) Perilla, J. R., Goh, B. C., Cassidy, C. K., Liu, B., Bernardi, R. C., Rudach, T., Yu, H., Wu, Z., Schulten, K.: Current Opinion in Structural Biology, 31, 64 (2015).
3) Kandasamy, S., Lee, D. K., Nanga, R. P. R., Xu, J., Santos, J., Larson, R. G., Ramamoorthy, A.: Biochim. Biophys. Acta., 1788, 686, (2009).
4) Shi, L., Cembran, A., Gao, J., Veglia, G.: Biophys. J., 96, 3648 (2009).
5) Toraya, S., Javkhlantugs, N., Mishima, D., Nishimura, K., Ueda, K., Naito, A.: Biophys. J., 99, 3282 (2010).
6) Javkhlantugs, N., Naito, N., Ueda, K.: Biophys. J., 101, 1212 (2011).
7) Brooks, B. R., Bruccoleri, R. E., Olafson, B. D., State, D. J., Swaminathan, S., Karplus, M.: J. Comput. Chem., 4, 187 (1983).
8) Tsutsumi, A., Javkhlantugs, N., Kira, A., Umeyama, M., Kawamura, I., Nishimura, K., Ueda, K., Naito, A.: Biophys. J., 103, 1735 (2012).
9) Haney,E. F., Lau, F., Vogel, H. J.: Biochim. Biophys. Acta, 1768, 2355 (2007).
10) Norisada, K., Javkhlantugs, N., Mishima, D., Kawamura, I., Saito, H., Ueda, K., Naito, A.: J. Phys. Chem. B, 121, 1802 (2017).
11) Kira, A., Javkhlantugs, N., Miyamori, T., Sasaki, Y., Eguchi, M., Kawamura, I., Ueda, K., Naito, A.: J. Phys. Chem. B, 118, 9604 (2014).