【トピックス】
メタン生成アーキアにおけるRuBisCOを利用した新規CO2固定経路
蘆田弘樹
神戸大院・人間発達環境
1.はじめに
光合成は、太陽からの光エネルギーを化学的エネルギーに変換し、このエネルギーをCO2の炭素から合成する糖や炭水化物といった有機物へ蓄積する。地球上において光合成生物以外のほとんどの生物の生命活動は、このように光合成によって作り出される有機物に依存しているため、光合成がこれら生命活動・維持のためのエネルギーを作り出す源の反応となっている。植物、藻類、光合成細菌などの光合成において、CO2の有機物への固定化には、CO2固定代謝経路であるカルビンサイクルが広く利用されている。カルビンサイクルは、メルヴィン・カルビンがアンドリュー・ベンソンとジェームズ・バッシャムと共に発見した光合成生物に特徴的なCO2固定経路で、その名には発見者の名前が付けられている。カルビンサイクルは、11種類の酵素によって触媒される13の反応ステップから構成されており、大きく3つのステップに分けることができる (図1)1)。第1はCO2固定ステップで、ribulose-1,5-bisphosphate carboxylase/oxygenase (RuBisCO) によってribulose-1,5-bisphosphate (RuBP) へのCO2固定反応 (カルボキシラーゼ反応) が触媒される。RuBisCOのカルボキシラーゼ反応では、RuBPに1分子のCO2を固定し、2分子の3-phosphoglycerate (PGA) を生成する。RuBisCOによって固定されたPGAの炭素は、第2ステップで光エネルギーから合成された還元力を利用して還元され、glycelaldehyde-3-phosphateへ変換される。第3ステップでは、glycelaldehyde-3-phosphateの一部から、三、四、六、七炭糖間で縮合・転移反応を経て、五炭糖であるRuBisCOのCO2アクセプター基質RuBPが再生される。RuBP再生ステップの最終反応として、ribulose-5-phosphate (Ru5P) からphosphoribulokinase (PRK) の触媒によってRuBPが合成される2)。
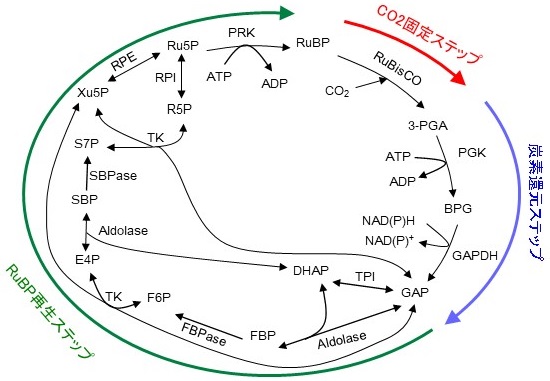

これまで、カルビンサイクルは光合成生物に特徴的な代謝経路であると考えられてきた。カルビンサイクル酵素のうち、RuBisCO、PRK、sedoheptulose-1,7-bisphosphatase (SBPase) はこの回路に特徴的であり3)、残りのステップは解糖系、糖新生、ペントースリン酸経路の酵素触媒ステップと重複している。多数の生物のゲノムシークエンスが進められ、大量の遺伝子配列が明らかにされた結果、興味深いことに光合成生物ではないアーキアやバクテリアがRuBisCOと相同性を示すタンパク質をコードする遺伝子を有することが明らかになってきた。これまで光合成生物に特徴的だと考えられてきたRuBisCO相同遺伝子が非光合成生物に保存されていたことは、光合成やカルビンサイクルの進化を考える上で、興味深い事実である。本稿では、これら光合成を行わない生物の中でも、メタン生成アーキアにおいて明らかになってきたRuBisCOを利用した新規CO2固定経路について紹介したい。
2.アーキアが有するカルビンサイクル酵素ホモログ
アーキアのゲノムデータベース情報から、シークエンスが完了している約半数のアーキアがRuBisCOホモログ遺伝子を保存していることが明らかになっている。現在までのところ、ユーリアーキオータのみがRuBisCOホモログ遺伝子を有しており、クレンアーキオータとコルアーキオータなど他の門においては、発見されていない。これらアーキアのRuBisCOホモログは、超好熱性アーキアであるThermococcus kodakarensis4)、Archaeoglobus profundus5)やメタン生成アーキアであるMethanospirillum hungatei6)、Methanocaldococcus jannaschii5)などでカルボキシラーゼ活性を有する正真正銘のRuBisCOであることが確認されている。RuBisCOは植物、藻類、シアノバクテリア、光合成細菌が有するラージサブユニット8個とスモールサブユニット8個から構成されるform I、光合成細菌が有するラージサブユニットのみから構成されるform II、アーキアが有するラージサブユニットのみから構成されるform IIIに分類される (図2)7,8)。また、自然界には、非光合成バクテリア、アーキア、光合成バクテリアが有するRuBisCOと相同性を示すがカルボキシラーゼ触媒能を示さないRuBisCO-like proteinも存在する。RuBisCO-like protein は、枯草菌で良く解析されており、メチオニン再生経路においてエノラーゼ酵素として機能している。本稿では解説しないので、論文を参照頂きたい9,10)。RuBisCOラージサブユニットの分子系統樹において、アーキアのform III RuBisCOは、form I、IIとは独立したクレードを形成し、メタン生成アーキアRuBisCOから構成されるform III-aとそれ以外のアーキアRuBisCOから構成されるform III-bのサブグループに分かれる。
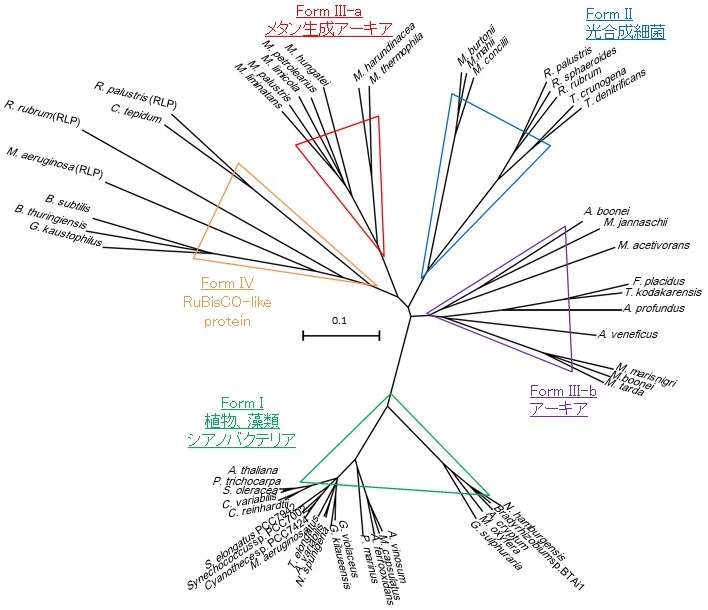

興味深いことに、form III-aに属するRuBisCOを有するメタン生成アーキアは、PRKホモログ遺伝子も同時に有している。これらメタン生成アーキアであるM. hungatei、Methanoculleus marisnigri、Methanosaeta thermophila、Methanosaeta concilliのPRKホモログの大腸菌組換えタンパク質の酵素学的解析から、全てRuBP合成活性を有していたことから、メタン生成アーキアがPRKを有していることが明らかになった6)。筆者らのメタン生成アーキアM. hungatei PRKのX線結晶解析から、このアーキアPRKが全体的な構造だけでなく、活性中心におけるRu5P、ATP結合に関与する触媒アミノ酸残基の配置まで、光合成細菌PRKと類似していることが分かった6)。これらは、アーキア型PRKのはじめての報告となった。
3.メタン生成アーキアにおけるRuBisCOを利用した新規CO2固定経路
一部のアーキアが光合成カルビンサイクルに特有な酵素であるRuBisCOまたはPRKを有しており、特にメタン生成アーキアの一部が、両酵素を同時に有していたことからアーキアにおいてカルビンサイクルが機能している可能性が考えられた。そこで筆者らは、メタン生成アーキアM. hungateiを用いて、RuBisCOとPRKが機能する代謝経路の解析を行った。一般的に、アーキアはペントースリン酸経路を利用しておらず11)、カルビンサイクルにおけるfurctose-6-phosphate (F6P) からRu5Pまでの代謝ステップを触媒するtransketolase、SBPase、ribulose-5-phosphate 3-epimeraseの遺伝子を欠失しており、M. hungateiもこれら酵素遺伝子を欠いていた。このアーキアのゲノム配列情報の解析から、F6PからRu5Pへの代替経路としてRibulose monophosphate pathwayが予想された。Ribulose monophosphate pathway は、一部のバクテリアやアーキアにおいてformaldehydeをRu5Pに固定しF6Pを合成する経路である12)。この経路では、arabino-3-hexulose-6-phosphate synthase (HPS) によりRu5Pにformaldehydeが固定されarabino-3-hexulose-6-phosphateが生成し、6-phospho-3-hexuloisomerase (PHI) によってarabino-3-hexulose-6-phosphate がF6Pに変換される。筆者らは、M. hungateiゲノム情報から予想されたHPSとPHI候補遺伝子からの大腸菌組換えタンパク質を用いた生化学的解析から、この2つの酵素を同定した。
以上のようにM. hungateiにおいて、光合成カルビンサイクルのF6PからRu5Pの代謝ステップがRibulose monophosphate pathwayに置換したRuBisCOを利用した新規のCO2固定経路が予想された。この経路は、中間代謝物であるarabino-3-hexulose-6-phosphateの名前から、reductive hexulose phosphate (RHP) 経路と名付けられた (図3)。RHP経路がM. hungateiの生体内で機能しているか調べるために、無細胞抽出液を用いて、RuBPもしくはF6PからPRKとRuBisCOの触媒反応を経由してPGAが合成されるか解析した。その結果、どちらの出発物質からもPGAの生成が確認されるとともに、PGAの生成はRuBisCOの特異的反応阻害剤である反応中間体アナログ2-carboxy-arabinitol-1,5-bisphosphate (CABP) の添加により完全に抑制された (図4)。さらに、M. hungateiの菌体に13CでラベルしたCO2を与え、様々な代謝産物への13C取り込みの経時変化をCE-MSを用いたメタボローム解析により追跡した。その結果、13CO2を与えた直後からRuBisCOカルボキシラーゼ反応産物であるPGAへの13Cラベルが確認され、さらにラベル速度が他の代謝産物よりも高いことから、M. hungateiにおいてRuBisCOが主要なCO2固定ステップを触媒していることが明らかになった。またメタボローム解析から、RuBisCOによって固定化された炭素の多くが解糖系、糖新生へ、一部の炭素がRHP 経路へ供給されていることが示唆された6)。これらの結果から、M. hungatei生体内でRHP 経路が機能していることが明らかになった。
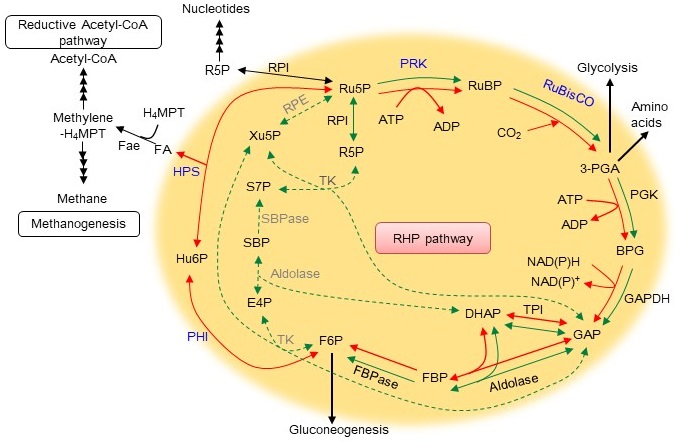

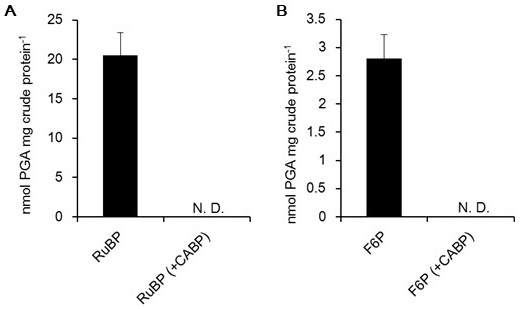

4.RHP経路の生体内における役割
RHP経路の生体内における役割はいくつか予想される。一つ目は、RuBisCOによって固定化した炭素をF6PまたはPGAを介して解糖系と糖新生へ供給することである。二つ目は、核酸合成のためのribose骨格を供給することである。既に上述したが、M. hungateiは他のアーキアと同様にペントースリン酸経路を有していないため、RHP経路を利用してRu5Pを合成し、ribose-5-phosphateへ異性化することで、核酸合成材料を獲得していると考えられる。これは、このアーキアにおける核酸合成がRHP経路に完全に依存していることを意味する。またRHP経路においてRuBPが再生される場合、formaldehydeが放出される。メタン生成アーキアでは、formaldehyde activating enzyme (Fae) の触媒によりformaldehydeをtetrahydromethanopterinに固定し、エネルギー生産のためのメタン生成代謝13)ともう一つのCO2固定経路である還元的acetyl-CoA経路14)の共通中間代謝物であるmethylene tetrahydromethanopterinを合成することが報告されている15)。このことから、3つ目の役割は、還元炭素をメタン生成代謝と還元的acetyl-CoA経路へ供給することであると考えられる。しかしながら、これらの機能予測は、今後生化学的、代謝学的解析により詳細な解析によって確かめる必要がある。また、M. hungateiは遺伝子組換え系が確立されていないので、現在のところ遺伝子破壊を行うことができない。遺伝子組換え系が確立されれば、RuBisCO遺伝子破壊株の生育や代謝を解析することにより、RHP 経路のより詳細な機能解析が可能になると期待される。
5.おわりに
今回、M.hungateiで明らかにしたRHP経路は、多くのメタン生成アーキアがこの経路で働く酵素遺伝子を保存していることから、メタン生成アーキアに広く分布していると予想される。また、超好熱性アーキアであるArchaeglobus属やFerroglobus属にも保存されていることから、メタン生成アーキアだけでなく、他のアーキアも広く利用している可能性が考えられる。RHP経路は一部がRibulose monophosphate pathwayに置換されている以外は光合成カルビンサイクルと共通している。このことと、アーキアが生物進化の初期、光合成が確立する以前に出現した生物であると考えられていることから、RHP経路が光合成カルビンサイクルの進化的原型かもしれない。もしそうだとすれば、RHP経路はRuBisCOが固定した炭素がRibulose monophosphate pathwayステップにおいてformaldehydeとして放出されるためCO2固定経路としては不完全であるが、transketolase、SBPase、ribulose-5-phosphate 3-epimeraseを経路に取り込むことで完全なCO2固定経路である光合成カルビンサイクルが完成したのではないだろうか。今後の研究から、生物進化において、どのように光合成カルビンサイクルが確立したのかが明らかにされると期待される。
謝辞
本研究は、神戸大学・河野卓成研究員、松田真実研究員、蓮沼誠久教授、奈良先端科学技術大学院大学・横田明穂名誉教授、立命館大学・松村浩由教授、ビルラ理工大学・Sandhya Mehrotra博士、大阪大学・溝端栄一講師、井上豪教授との共同研究の成果である。関係者各位に深く感謝する。本研究は、JSTさきがけ、CREST、科学研究費補助金 (26292193) からの研究費により行った。
文献
1) Martin, W., Scheibe, R., Schnarrenberger, C.: Photosynthesis, 9, 9 (2000).
2) Miziorko, H. M.: Adv. Enzymol. Relat. Areas Mol. Biol., 74, 95 (2000).
3) Raines, C. A.: Photosyn. Res., 75, 1 (2003).
4) Ezaki, S., Maeda, N., Kishimoto, T., Atomi, H., Imanaka, T.: J. Biol. Chem., 274, 5078 (1999).
5) Finn, M. W., Tabita, F. R.: J. Bacteriol., 185, 3049 (2003).
6) Kono, T., Mehrotra, S., Endo, C., Kizu, N., Matsuda, M., Kimura, H., Mizohata, E., Inoue, T., Hasunuma, T., Yokota, A., Matsumura, H., Ashida, H.: Nat. Commun., 8:14007, doi: 10.1038/ncomms14007 (2017).
7) Tabita, F. R., Hanson, T. E., Satagopan, S., Witte, B. H., Kreel, N. E.: Phil. Trans. R. Soc. Lond. B. Biol. Sci., 363, 2629 (2008).
8) Badger, M. R., Bek, E. J.: J. Exp. Bot., 59, 1525 (2008).
9) Ashida, H., Saito, Y., Kojima, C., Kobayashi, K., Ogasawara, N., Yokota, A.: Science, 302, 286 (2003).
10) Ashida, H., Saito, Y., Nakano, T., Tandeau de Marsac, N., Sekowska, A., Danchin, A., Yokota, A. J.: Exp. Bot., 59, 1543 (2008).
11) Soderberg, T.: Archaea, 1, 347 (2005).
12) Kato, N., Yurimoto, H., Thauer, R. K.: Biosci. Biotech. Biochem., 70, 10 (2006).
13) Thauer, R. K., Kaster, A. K., Seedorf, H., Buckel, W., Hedderich, R.: Nat. Rev. Microbiol., 6, 579 (2008).
14) Berg, I. A., Kockelkorn, D., Ramos-Vera, W. H., Say, R. F., Zarzycki, J., Hugler, M., Alber, B. E., Fuchs, G.: Nat. Rev. Microbiol., 8, 447 (2010).
15) Goenrich, M., Thauer, R. K., Yurimoto, H., Kato, N.: Arch. Microbiol., 184, 41 (2005).