【トピックス】
酵母による海藻 (褐藻、紅藻) 糖質発酵へ向けて
河井重幸、村田幸作
京大院・農、摂南大・理工
1.はじめに
広大な管轄水域を有する我が国にとって、海洋バイオマスは自国海域内での大量養殖が可能な未利用資源である。海洋バイオマスは海草と海藻 (緑藻、紅藻、および褐藻) に大別できる。緑藻糖質として、グルカン (グルコース重合体:セルロース、デンプン)、硫酸化多糖 (ウルバン) が、褐藻糖質として、マンニトール、アルギン酸、グルカン (セルロース、ラミナリン) が、紅藻糖質として寒天 (アガロース、アガロペクチン)、カラギーナン、グルカン等が知られている。グルカンは全ての海藻に存在するものの、陸性バイオマス (32〜46%) に比して海藻のグルカン含量 (22〜25%) は低い1)。従って、陸性バイオマスの糖化方法を海藻由来糖質に適用しても高濃度のエタノールを生産することができない。グルカン以外の海藻糖質 (ウルバン、マンニトール、アルギン酸、寒天、カラギーナン) からのエタノール等の発酵生産技術を確立する必要がある所以である。
筆者らは、特に藻体が巨大で海の森と言われる藻場を形成する褐藻の糖質 (アルギン酸、マンニトール) を原料とした、微生物 (スフィンゴモナス属細菌A1株、酵母) による有用化合物 (エタノール、ピルビン酸など) の発酵生産系の構築に取り組んできており2-4)、現在は、特に出芽酵母Saccharomyces cerevisiaeを対象とした研究に注力している5)。アルギン酸は、Mブロック (マンヌロン酸残基のみから構成)、Gブロック (グルロン酸残基のみから構成)、およびMGブロック (マンヌロン酸残基とグルロン酸残基が混在) から構成されるポリウロン酸である (図1)。褐藻に35% (w/w 以下同様) 程度含まれる6)。マンニトールはフルクトースの還元体であり、ポリオール (糖アルコール) の一種である。マンニトールデヒドロゲナーゼ (MDH) のはたらきにより、フルクトースへと酸化される。褐藻に5〜26%含まれる。褐藻は1〜18%のラミナリン (グルコースの重合体) も含む。マンニトールとラミナリン含量の季節変動は大きい。
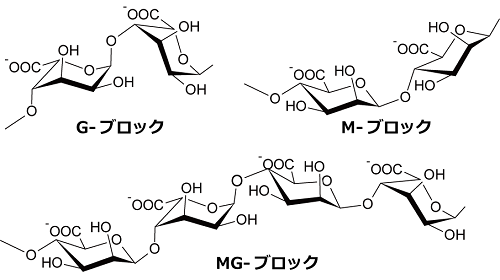
図1 アルギン酸構成単位の分子構造
出芽酵母は、パン、お酒などの食品の生産で伝統的に利用されてきた安全な食品微生物であり、多くの長所を有する優れた発酵微生物である。とりわけ頑丈な細胞壁構造を持つため、過酷な発酵環境 (褐藻糖質利用時に想定) でも充分な発酵能を発揮し得る点が魅力である。出芽酵母を用いたトウモロコシやサトウキビを原料としたエタノール生産がアメリカやブラジルで実用化されている。しかし、出芽酵母はラミナリン構成単糖であるグルコースは容易かつ強力に資化するが、アルギン酸とマンニトールを資化できないという問題があった。本稿では、出芽酵母によるアルギン酸とマンニトール (褐藻糖質) を原料とした発酵生産系構築に関する研究に関して、筆者らの研究も含めた内外の研究状況を紹介すると共に、最近、国外で進展の見られた紅藻の糖質 (アガロース) 利用研究に関しても紹介したい。
2.マンニトールの問題
マンニトールは自然界で最も著量な糖アルコールであり、また出芽酵母はゲノムDNA上にNAD+依存MDH遺伝子を有する。それにも拘らず、幾つかのポリプロイド株や一倍体A184D株などを除き、出芽酵母はマンニトールを資化できない。筆者らも、醸造酵母や一倍体出芽酵母 (BY4742、BY4741、AH109、DBY877、EBY100、SEY6210、T8-1D、YPH500) がマンニトールを唯一の炭素源とする培地 (以下、マンニトール培地) で生育しないことを確認した。その一方、酵母Saccharomyces paradoxus NBRC 0259はマンニトール培地で生育し、マンニトールからエタノールを生産することを見出した4)。さらに、一倍体出芽酵母BY4742株 (約5×106個) を合成マンニトール個体培地に塗布して長期間 (〜1週間) 保温すると数個のコロニーが出現し、これらのコロニーがマンニトール資化能を獲得することを見出した。前述の一倍体出芽酵母7株でも同様の現象が見出された。さらに、Tup1-Cyc8遺伝子への変異によりマンニトール資化能が獲得されることを明らかにし、Cyc8遺伝子への変異導入により出芽酵母にマンニトール資化能を付与できることを示した5)。Tup1-Cyc8は転写コリプレッサー複合体を形成し、多くの遺伝子の転写制御に関わる。BY4742株由来のマンニトール資化能獲得株の一つMK4416株(cyc8>Δ1139-1164アレルを有する)は、11日間で100 g/Lのマンニトールから40g/Lのエタノールを生産した。
一方、Enquist-Newmanらも、3つの出芽酵母株 (Lalvin、Pasteur Red、SEY/Dip) がマンニトールを代謝できないことを確認した上で、これら3株から「マンニトール培地で効率的に生育できるように誘導」した各誘導株を得た7) (どのようにして誘導したのかは残念ながら不明)。さらにマイクロアレー解析等によって、MDH遺伝子 (YNR073C) とMFSトランスポーターホモログ遺伝子 (HXT17またはHXT13) の転写の上昇を見出し、両遺伝子を強制発現させることで出芽酵母にマンニトール利用能を付与できることを実証した7)。なお、YNR073Cのパラログ遺伝子としてDSF1も酵母のゲノム上に存在する。最近、HXT13、HXT15、HXT16、HXT17各産物がマンニトールおよびソルビトールのトランスポーターとして機能すること、およびYNR073CとDSF1各遺伝子産物がMDHとして機能することが示された7,8)。確かに、筆者らもマンニトール培地で生育させたマンニトール資化能獲得株 (TUP1に変異を有する) においては、グルコース培地における転写量と比較して、YNR073C/DSF1の転写量が14.2〜16.4倍、HXT13、HXT15、HXT17の転写量がそれぞれ4.5〜5.2倍、2.2〜2.6倍、3.4〜3.9倍に上昇していることを確認している7)。これらの知見から、出芽酵母のゲノムDNA上のYNR073C/DSF1およびHXT13/HXT15/HXT17各遺伝子の発現がTup-Cyc8のはたらきで抑制されているために、出芽酵母はマンニトールを資化できないと推察された (図2)。いずれにせよ、非組み換え (CYC8のcyc8Δ1139-1164への置換) または組換え (YNR073CとHXT17の強制発現) により、出芽酵母にマンニトール資化能を付与することが可能になった。
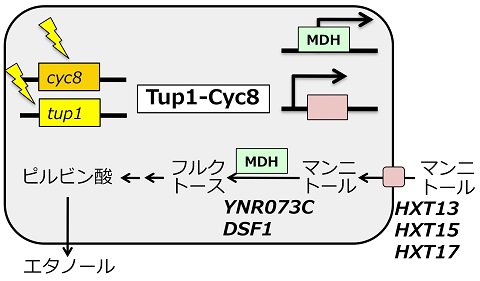
図2 Tup1-Cyc8遺伝子への変異による出芽酵母のマンニトール資化能の獲得
3.アルギン酸の問題
図3にアルギン酸代謝経路を示す。アルギン酸資化性のスフィンゴモナス属細菌A1株においては、アルギン酸が精妙なABCトランスポーターにより細胞内に取り込まれ、細胞内でモノウロン酸 (DEH:4-デオキシ-L-エリスロ-5-ヘキソセウロース ウロン酸) へと変換され、細胞内でDEHがさらに3つの酵素 (同A1株では、DEHレダクターゼA1-R/A1-R’、KDGキナーゼA1-K、KDPGアルドラーゼA1-K) のはたらきで、2-ケト-3-デオキシホスホグルコン酸を経由してピルビン酸とグリセルアルデヒド-3-リン酸に変換される6)。2-ケト-3-デオキシホスホグルコン酸からピルビン酸とグリセルアルデヒド-3-リン酸への開裂は、Entner-Doudoroff経路でも見られる。ABCトランスポーターならびに3つの酵素 (A1-R/A1-R’、A1-K、A1-K) の各遺伝子を出芽酵母はゲノムDNA上に持たない。アルギン酸に特異的なABCトランスポーター構成遺伝子の出芽酵母への導入は明らかに困難と予想されたため、2つの反応すなわち「3-1 DEHの取り込みと資化反応」ならびに「3-2 アルギン酸からDEHへの糖化反応」に分けて、出芽酵母へのアルギン酸資化能の付与を試みた。
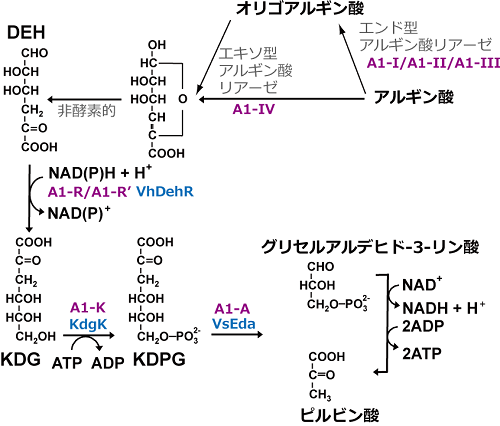

3-1 DEHの取り込みと資化反応
DEHの資化反応に関しては上記の3つの酵素 (A1-R/A1-R’、A1-K、A1-K) に相当する酵素の遺伝子を出芽酵母に導入すればよい。筆者らも2012年より本研究に着手した。しかし、最初にこの試みに成功したのは米国のEnquist-Newmanらのグループであった7)。同グループは、アルギン酸を資化するカビAsteromyces cruciatusからDEH取り込み遺伝子を初めて特定し、同遺伝子ならびに上記の3つの酵素に相当する酵素 (それぞれVhDehR、KdgK、VsEda) の遺伝子のコドンを出芽酵母最適型に変換し、プロモーターの下流およびターミネーター上流に連結し、出芽酵母のゲノムに挿入した (図3)。さらに上記のYNR073CとHXT17の強制発現によりマンニトール資化能も付与し、DEHを炭素源とする培地で適応進化させ、さらにマンニトールおよびDEH混合培地で嫌気条件で生育可能な株を選抜した。得られた株は、98 g/Lの糖 (DEHとマンニトールをモル比1:2の比率で含む) を原料として、36.2 g/Lのエタノールを生産した。ただし、適応進化のメカニズムは不明である。筆者らも、本研究を継続しており、適応進化のメカニズムも明らかにしつつある。別稿でご紹介できれば幸いである。
3-2 アルギン酸からDEHへの糖化反応
アルギン酸はエンド型アルギン酸リアーゼの作用によりオリゴアルギン酸へ、オリゴアルギン酸はエキソ型アルギン酸の作用で不飽和単糖へ分解され、不飽和単糖は非酵素的にDEHへと変換 (糖化) される (図3)6)。エキソ型アルギン酸リアーゼはアルギン酸にも作用する (図3)。そこで、 (1) 強力なエキソ型アルギン酸リアーゼを出芽酵母の細胞表層あるいは細胞外で発現させる、または (2) 前処理の段階でアルギン酸含有液に酵素製剤としてエキソ型アルギン酸リアーゼを添加して反応させるという、2つのアルギン酸糖化方法が想定される。いずれにせよ、どのエキソ型アルギン酸リアーゼを用いるかが問題である。幾つかのエキソ型アルギン酸リアーゼが報告されている。スフィンゴモナス属細菌由来A1-IV、クラウンゴール原因細菌Agrobacterium tumefaciens由来Atu3025、海洋細菌Saccharophagus degradans由来Alg17c、海洋細菌Vibrio splendidus由来OalA、OalB、OalC、海洋細菌Zobellia galactanivorans由来AlyA5、海洋細菌Flavobacterium sp. strain UMI-01由来FlAlyB、AlAlyC、FlAlex6,9)の性質が詳しく調べられている。また、出芽酵母の細胞表層に提示されたS. degradans由来Alg7Kがエキソ型アルギン酸リアーゼ活性を示すことも報告されている10)。FlAlyCとFlAlexはPL-6に、AlyA5とAlg7KはPL-7に、A1-IV、Atu3025、およびOalAは PL-15に、Alg17c、OalB、OalC、およびFlAlyBはPL-17に、それぞれ分類されている。しかし、これらの酵素の活性が全て同一条件、同一方法で測定されていなかったので、どのリアーゼが最適なのかは不明であった。そこで筆者らは、まずはA1-IV、Atu3025、およびAlg17cに着目し、それらの組換え精製酵素を用いて生成DEH量を指標に同一条件で活性を測定し、これらの性能の比較を行った11)。その結果、活性自体はA1-IV>Atu3025>>Alg17cでA1-IVが最大、Alg17cが最低であったが、Alg17cは300 mM程度のNaClにも耐塩性を示した。耐塩性は海洋バイオマスの糖化にとって優れた特性と期待された。他方、Atu3025はA1-IVと異なり、透析と凍結融解に耐性を示したため、酵素製剤として使用するにはAtu3025が最適と考えられた。現在、筆者らはAtu3025を用いてDEHを調製している。
4.紅藻糖質 (アガロース) の問題
紅藻糖質として寒天 (アガロース、アガロペクチン)、カラギーナン、グルカン等が知られている。これらのうち、アガロースの研究が進んでいる。アガロースは、β-D-ガラクトース (Gal) と3,6-アンヒドロ-α-L-ガラクトース (AHG) からなる二糖を繰り返し単位とする多糖である (結合様式はGal-β-1,4-AHG-α-1,3-Gal:図4A)1)。アガロースの分解で生じたオリゴ糖のうち、非還元末端がGalの場合はアガロオリゴ糖 (AOS)、非還元末端がAHGの場合はネオアガロオリゴ糖 (NAOS) と呼ばれる。α-アガラーゼはアガロースのα-1,3結合に作用しAOSへと分解し、β-アガラーゼIはβ-1,4結合に作用してNAOSへと分解、β-アガラーゼIIはβ-1,4結合に作用してアガロトリオース、ネオアガロビオース (AHG-α-1,3-Gal:図4B)、AHGへと分解する。NAOSヒドロラーゼはNAOSのα-1,3結合に作用し、AHGを遊離させるが、アガロトリオースには作用しない12)。
アガロースの糖化方法として最良と考えられているのが、弱酸処理と酵素処理の併用法である (図4B)13)。まず弱酸処理により、非特異的ではあるが比較的α-1,3-結合に特異的にAOS (偶数個の鎖長) へ分解され、図4Bに示す通りにGalとAHGへ糖化される。なお、β-アガラーゼ IIやNAOSヒドロラーゼがアガロトリオースを分解しないという問題があったが、アガロトリオースに作用してGalとネオアガロビオースに分解できるアガロース分解性β-ガラクトシダーゼが発見され、この問題は解決された12)。
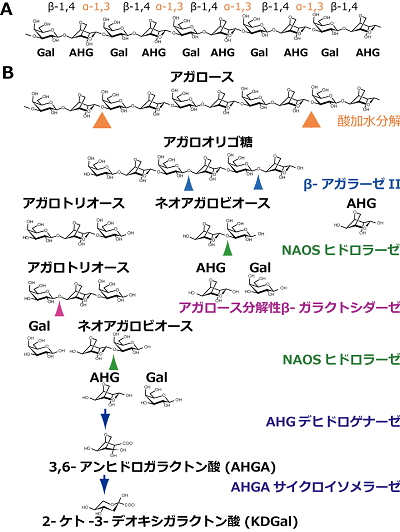

Galは出芽酵母によって容易に発酵されるが、グルコースと比べて約半分の効率でしかないので、出芽酵母のGal資化能を向上させるための努力がなされている6)。少なくともGal代謝酵素の一つPgm2 (グルコース-1-リン酸をグルコース-6-リン酸へ変換) が、Gal資化能向上の鍵酵素である。他方、出芽酵母や大腸菌によって資化されないAHGが、アガロース利用の最大の問題であったが、最近Vibrio sp. EJY株における新規なAHG代謝経路が特定された14)。本経路では、AHGはNADP+-依存AHGデヒドロゲナーゼにより3,6-アンヒドロガラクトン酸(AHGA)へと酸化され、AHGAサイクロイソメラーゼにより2-ケト-3-デオキシガラクトン酸 (KDGal) へと変換される (図4B)。KDGalはさらに2-ケト-3-デオキ-6-ホスホシガラクトン酸 (KDPGal) へと変換されて中央代謝経路へと合流する。KDGalからKDPGal、さらにはピルビン酸とグリセルアルデヒド-3-リン酸への開裂反応は、細菌における酸化的ガラクトース代謝経路 (DeLey-Doudoroff経路) でも見られる。今後はDEHの場合と同様、これらのAHG代謝酵素遺伝子の出芽酵母への導入等による出芽酵母へのアガロース利用能の付与研究が進むと期待される。
5.おわりに
マンニトール問題に関しては、マンニトール付与のための二つの方法、非組み換え (CYC8のcyc8Δ1139-1164への置換) または組換え (YNR073CとHXT17の強制発現) による方法、どちらが優れているかを検証する必要がある。マンニトールおよびDEHの資化問題では、適応進化等による両糖質資化能のさらなる向上、その向上メカニズムの完全解明が待たれる。技術的課題は尽きないが、今後の進展を期待したい。褐藻糖質に関しては、AHG資化能の出芽酵母への付与が当面の課題である。本稿では、緑藻主成分であり褐藻や紅藻にも含まれるグルカン利用に触れなかったが、この問題に関しては筆者らの総説1)を参照いただけたらと思う。また、これも本稿では触れなかったが、アルギン酸利用能を大腸菌に付与した研究例も米国グループ15)および味の素らのグループ16)により報告されている。米国グループは20個以上の遺伝子を導入し、コンブ破砕物 (130 g/L、ただし段階的に追加) から、6日間で35〜41 g/Lのエタノールの生産を確認している。味の素らのグループは、わずか7個の遺伝子の導入により「AlyBで分解したアルギン酸」の利用能の付与に成功し、L-リジン生産大腸菌を用いることで、分解アルギン酸から約40 mg/LのL-リジンの生産を確認している。
海藻原料を輸入に頼らず国内産で賄うことにより、「我が国」の雇用の創出、海洋資源の涵養、水質浄化 (自国海洋保全)、地球温暖化防止 (二酸化炭素吸収) 等の恩恵が期待できる。褐藻アカモクの養殖技術は、京都府農林水産技術センター海洋センターで確立され、京都府沿岸で実地試験が進行中であり、新潟県も同技術を活用されていると聞く。海藻の積極的な養殖と発酵による有効利用は、今後の展開が楽しみで、かつ重要な領域である。そのための技術的課題を一つずつ解決していきたい。
謝辞
本稿で紹介した私どもの研究は、第11回 農芸化学研究企画賞、JST A-STEP FSステージ (AS262Z00582N)、第41回 岩谷科学技術研究助成、アサヒグループ学術振興財団 助成金、松籟科学技術振興財団 助成金による支援のもとで行われました。また、本稿執筆の機会を与えて下さいました、ノボザイムズ ジャパン 松井知子先生に篤く御礼申し上げます。
文献
1) Yanagisawa, M., Kawai, S., Murata, K.: Bioengineered, 4, 224 (2013).
2) Kawai, S., Ohashi, K., Yoshida, S., Fujii, M., Mikami, S., Sato, N., Murata, K.: J. Biosci. Bioeng., 117, 269 (2014).
3) Takeda, H., Yoneyama, F., Kawai, S., Hashimoto, W., Murata, K.: Energy. Environ. Sci., 4, 2575 (2011).
4) Ota, A., Kawai, S., Oda, H., Iohara, K., Murata, K.: J. Biosci. Bioeng., 116, 327 (2013).
5) Chujo, M., Yoshida, S., Ota, A., Murata, K., Kawai, S.: Appl. Environ. Microbiol., 81, 9 (2015).
6) Kawai, S., Murata, K.: Int. J. Mol. Sci., 17, doi: 10.3390/ijms17020145 (2016).
7) Enquist-Newman, M., Faust, A. M., Bravo, D. D., Santos, C. N., Raisner, R. M., Hanel, A., Sarvabhowman, P., Le, C., Regitsky, D. D., Cooper, S. R., Peereboom, L., Clark, A., Martinez, Y., Goldsmith, J., Cho, M. Y., Donohoue, P. D., Luo, L., Lamberson, B., Tamrakar, P., Kim, E. J., Villari, J. L., Gill, A., Tripathi, S. A., Karamchedu, P., Paredes, C. J., Rajgarhia, V., Kotlar, H. K., Bailey, R. B., Miller, D. J., Ohler, N. L., Swimmer, C., Yoshikuni, Y.: Nature, 505, 239 (2014).
8) Watanabe, Y., Takechi, Y., Nagayama, K., Tamai, Y.: Enzyme Microb. Technol., 39, 654 (2006).
9) Inoue, A., Nishiyama, R., Ojima, T.: Algal Res, 19, 355 (2016).
10) Takagi, T., Yokoi, T., Shibata, T., Morisaka, H., Kuroda, K., Ueda, M.: Appl. Microbiol. Biotechnol. (2015).
11) Hirayama, M., Hashimoto, W., Murata, K., Kawai, S.: Int. J. Biol. Macromol., 86, 519 (2016).
12) Lee, C. H., Kim, H. T., Yun, E. J., Lee, A. R., Kim, S. R., Kim, J. H., Choi, I. G., Kim, K. H.: Appl. Environ. Microbiol., 80, 5965 (2014).
13) Kim, H. T., Lee, S., Kim, K. H., Choi, I. G.: Bioresour. Technol., 107, 301 (2012).
14) Yun, E. J., Lee, S., Kim, H. T., Pelton, J. G., Kim, S., Ko, H. J., Choi, I. G., Kim, K. H.: Environ. Microbiol., 17, 1677 (2015).
15) Wargacki, A. J., Leonard, E. M., Win, N., Regitsky, D. D., Santos, C. N. S., Kim, P. B., Cooper, S. R., Raisner, R. M., Herman, A., Sivitz, A. B., Lakshmanaswamy, A., Kashiyama, Y., Baker, D., Yoshikuni, Y.: Science, 335, 308 (2012).
16) Doi, H., Tokura, Y., Mori, Y., Mori, K., Asakura, Y., Usuda, Y., Fukuda, H., Chinen, A.: Appl. Microbiol. Biotechnol., (2016).