【トピックス】
"糖脂質酵素 (Glycolipozyme)" MPIaseの構造と作用機作
西山賢一、島本啓子
岩手大・寒冷バイオ、サントリー生命科学財団・生有研
1.はじめに
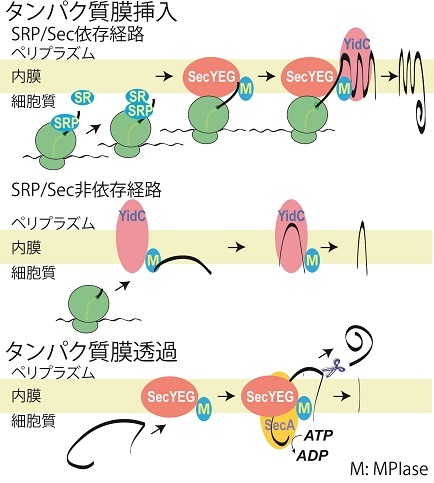
膜タンパク質はすべての生物で発現し、生体膜の内外の物質輸送や情報伝達など生命活動に必須の機能をもっている。膜タンパク質の膜挿入機構は、「シグナル仮説」1)の延長として考えることができる。シグナル仮説では、合成途中の分泌タンパク質のシグナルペプチドがシグナル認識粒子 (SRP) により認識され、新生鎖-SRP-リボソーム複合体が膜上のSRP受容体 (SR) に輸送される。その後、タンパク質膜透過チャネル (真核生物ではSec61複合体、原核生物ではSecYEG) に受け渡され、膜透過が進行する。膜タンパク質の場合は、疎水的な膜貫通領域がSRPに認識された後、同様の経路で膜挿入が進行すると考えられている (図1上段)。タンパク質膜透過チャネルやSRP/SRはすべての生物で保存されているため、タンパク質膜挿入の分子機構は、基本的なレベルではすべての生物で保存されていると考えられる。
SRPがシグナルペプチドや膜貫通領域に作用できるのは、これらを含むタンパク質が合成途中である必要がある2)。したがって、膜貫通領域がC末端にのみ存在する場合や膜タンパク質が非常に小さいときは、SRPを利用して膜挿入することができなくなる。こうした膜タンパク質はSRPにも膜透過チャネル (Sec因子) にも依存せずに膜挿入するため、膜貫通領域と膜内部の疎水的な相互作用により「自発的」に膜挿入するのではないかと考えられてきた。しかし、これらのタンパク質の膜挿入に必須な因子が明らかになってきた (図1中段)。以下、本因子について本稿で解説する。

2.タンパク質膜挿入反応の再構成
タンパク質膜挿入反応の詳細な分子機構を明らかにするためには、反応に関わる因子を分離同定し、タンパク質膜挿入能をもつプロテオリポソームを調製し、試験管の中で膜挿入反応を再構成する必要がある。このとき、細胞内部での反応を忠実に再現した反応系であることが極めて重要である。しかし、無秩序な「自発的膜挿入」のため、膜挿入反応の再構成は困難を極めた。変異株から調製した膜小胞をもちいてSecYEGやSRPに依存して膜挿入することが知られているMtlA (マンニトール・パーミアーゼ)3,4)は、リン脂質のみで形成しタンパク質成分を一切含まないリポソームにまで効率よく膜挿入してしまうことが明らかとなった5)。このMtlAの自発的膜挿入は、リポソームに生理的濃度でジアシルグリセロール (DAG) を加えることにより完全にブロックできることが明らかとなり、膜挿入反応の再構成の前提条件が整うこととなった。タンパク質膜挿入に関与すると報告されていたYidC (後述) やSecYEGを、DAG含有リポソームに再構成し、SRP/SR存在下でMtlAの膜挿入活性を測定したが、膜挿入活性は全く検出されなかった5)。このことは、タンパク質膜挿入に関わる未知の因子が存在することを強く示唆していた。
M13 procoatタンパク質をはじめとするSec/SRPに依存しない膜タンパク質は、膜タンパク質の膜貫通領域と脂質二重層内部との疎水的な相互作用により、自発的に膜挿入すると考えられてきた。プロテアーゼを封入したリポソーム存在下でM13 procoatを試験管内合成すると、自発的膜挿入し、その結果リポソーム内部のプロテアーゼによる消化を受けることからも、自発的膜挿入機構が実験的にも支持されていた6)。しかし、DAGを含むリポソームを用いた場合には、M13 procoatの膜挿入は全く観察されなくなった5,7)。従来、自発的膜挿入するとされてきた膜タンパク質もDAGが存在するとき、すなわち細胞内の環境では自発的には膜挿入しないことが判明した。このことは、SRPにもSec因子にも依存しないで膜挿入するM13 procoatのような膜タンパク質でも膜挿入には何らかの因子が必要であることを強く示唆していた。
3.タンパク質膜挿入反応を触媒するMPIase (Membrane Protein Integrase)
以上の点を踏まえ大腸菌内膜画分を分画し、SRPやSec因子、YidC等を含まない画分を調製し、DAGを含むリポソームに再構成したところ、M13 procoatの膜挿入活性を示す画分を得ることができた5)。この画分にSecYEGを混合し、再構成したところ、MtlAの膜挿入活性も検出された5)。これらの結果は、Sec/SRP依存、非依存にかかわらずすべての膜タンパク質の膜挿入に必要な因子が存在する可能性を強く示唆するものである。この膜挿入因子を、膜挿入活性を指標として精製を進めた結果、SDS-PAGE上で約8 kDaに泳動される因子を単離することができた。この因子をプロテイナーゼK処理すると活性が喪失するため、当初はこの因子はタンパク質であると考えていた5)。
Pf3 coatタンパク質はM13 procoat同様、SRPやSec因子に依存せずに膜挿入するタンパク質である8)。これらの因子には依存しないものの、Pf3 coatの膜挿入は膜電位により促進されるが、膜貫通領域にロイシン残基を3個挿入した変異体3L-Pf3 coatでは、膜挿入に膜電位は必要なくなることが知られている9)。Pf3 coatやM13 procoatはYidC枯渇株では膜挿入に阻害が生じることが知られている8)が、3L-Pf3 coatの場合はYidC枯渇の影響も受けなくなる10)。DAGを含むリポソーム存在下で3L-Pf3 coatを合成しても膜挿入活性は検出されないが、上述の膜挿入因子を組み込んでおけば効率の良い膜挿入が検出された7)。さらに、3L-Pf3 coatの合成量を増加させたところ、膜挿入した3L-Pf3 coatの量はリポソーム上の膜挿入因子量を上回っていた11)。このことは、この因子が膜挿入反応を触媒することを示している。タンパク質膜挿入を触媒することから、この因子をMPIase (Membrane Protein Integrase) と命名した11)。
4.MPIaseの構造決定
MPIaseと名付けた新因子は両親媒性で取扱いには苦労したが、HPLCと液-液分配カラムクロマトグラフィを組み合わせることで純品を得ることができた。精製したMPIaseを加水分解してHPLCで分析したところ、驚いたことにアミノ酸は全く検出されず、その酵素様の活性にもかかわらずタンパク質性の構造を持たないことが明らかになった。グルコサミンとアンモニアが検出されたことから、さらに加水分解物の分析を進め、MPIaseはアミノ糖を構成成分とする糖脂質であると推定した。質量分析 (MALDI-TOF-MS) は特徴的な繰り返しパターンを示し、そのフラグメント解析から、3種類の糖からなるユニットが10回程度連なった構造が示唆された。糖鎖の加水分解物を合成標品と比較することにより、3糖繰返しユニットの成分は「N-アセチルグルコサミン (GlcNAc) 」「N-アセチルマンノサミンウロン酸 (ManNAcA) 」「4-N-アセトアミノフコース (Fuc4NAc) 」であると推定した。また、GlcNAcの6位は部分的にO-アセチル化されていることがわかった。全体の1/3程度が不規則にO-アセチル化されているようである。次に、糖の繋がり方についての情報を得るために二次元NMR解析を行い、13C-NMRケミカルシフト値を帰属した。さらに標品となる部分構造を合成して、天然物とNMRを比較し、結合位置とアノマー位の立体構造を決定した。一方、脂質部は、メタノリシス生成物のGC-MSからジアシルグリセロールであることがわかった。脂肪酸については、大腸菌のロットにより異なる分布が見られた。さらに31P-NMRの結果から、糖鎖とジアシルグリセロール部はピロリン酸ジエステルを介して結合していることが明らかになった。以上の知見から、MPIaseの構造を図2に示す新規糖脂質であると決定した12)。
5.MPIaseの構造―機能相関関係
MPIaseの構造を基に構造―機能相関関係を調べた。MPIaseの糖鎖は脂質部分とピロリン酸で連結されているため、ピロリン酸フォスファターゼで消化すると可溶性の糖鎖が得られる (PP-MPIase)。一方、NaOHで処理すると、O-アセチル基を欠く糖鎖が得られる (NaOH-MPIase)。MPIaseのみに依存する3L-Pf3 coatを基質として膜挿入活性を調べたところ、PP-MPIaseは未処理のMPIaseよりも大幅に活性が上昇していたのに対し、NaOH-MPIaseはほとんど活性が検出されなかった12)。これらのことから、SRP/Sec非依存の膜挿入においてはMPIaseの脂質部分は必須ではないこと、しかしO-アセチル基は必須であることが判明した。一分子のMPIaseには30〜35個のアセチル基が存在する (図2)。プロテイナーゼKは基質特異性が低くMPIaseのN-アセチル基を切断したことから、失活したのではないかと考えられる。


Entrobacterial Common Antigen (ECA) は、グラム陰性細菌の細胞表層に普遍的に見られる糖脂質である。ECAの糖鎖部分はMPIaseと類似していたが、3糖ユニットの繰り返し数がMPIaseでは9〜11とほぼ均質なのに対し、ECAでは18〜55とばらつきが大きい13)。ECAには膜挿入活性は全く検出されなかった12)ことから、糖鎖の繰り返し数が活性に重要であることが判明した。
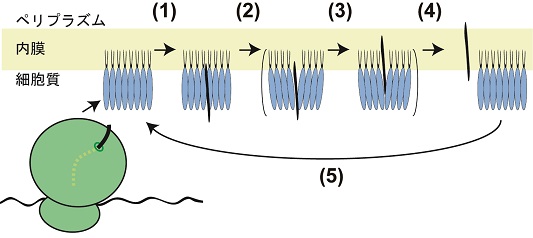
MPIaseの可溶性の糖鎖部分 (PP-MPIase) が膜挿入活性を示すので、MPIaseと基質膜タンパク質との相互作用を解析することが可能となった。膜タンパク質3L-Pf3 coatを試験管内で合成すると、瞬時に凝集体となり、ゲルろ過カラムではボイド画分にのみ回収される。それに対して、PP-MPIase存在下で3L-Pf3 coatを合成すると、両者は複合体を形成し、ゲルろ過カラムでも可溶性画分に回収される。細胞質画分存在下で3L-Pf3 coatを合成しても、こうした現象は観察されなかった12)。すなわち、MPIaseの糖鎖部分は、膜タンパク質に特化した分子シャペロン活性をもっているといえる。この複合体は膜挿入活性を保持しているため、膜タンパク質との相互作用は膜挿入反応の一過程であると考えられる。この際、糖鎖部分に存在する多数のアセチル基が疎水的な膜貫通領域との相互作用に重要であると考えられる。PP-MPIase単独でゲルろ過分析すると約8量体に相当する画分に溶出されるため、MPIaseはオリゴマーを形成している可能性がある。膜挿入反応においては、膜上でオリゴマーを形成したMPIaseが合成直後の膜タンパク質を受け取り、その後何らかの構造変化を経て膜挿入が進行すると考えられる (図3)。基質膜タンパク質が十分に多い場合、膜挿入反応は何サイクルも進行する11)ため、MPIaseは膜挿入反応を触媒しているといえる。こうした、MPIaseの酵素様の働きに基づき、我々はMPIaseがタンパク質膜挿入反応を触媒する「糖脂質酵素 (Glycolipozyme) 」であるという概念を提案している12)。

6.タンパク質膜透過チャネルSecYEGとMPIaseの機能的相互作用
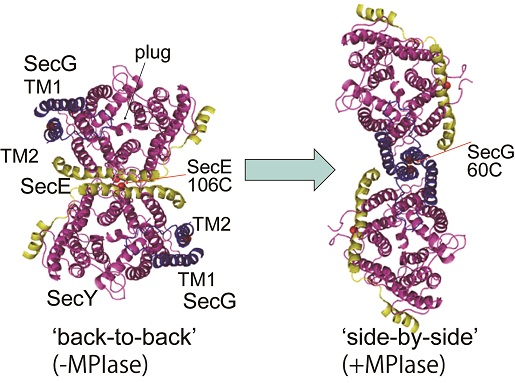
MPIaseはSRP/Sec依存のタンパク質膜挿入にも必須である5)。このことは、MPIaseとSecYEGが直接相互作用して機能する可能性を示している。一方、分泌タンパク質の膜透過反応は、ATPase活性をもつSecAと膜透過チャネルSecYEGにより進行する (図1下段)。MPIaseをSecYEGと共に再構成したところ、タンパク質膜透過活性が10倍以上促進された5,14)。この結果は、MPIaseがタンパク質膜挿入だけでなくタンパク質膜透過にも関与することを示す。MPIaseによるタンパク質膜透過促進の理由を調べたところ、MPIaseがSecYEGの二量体構造に大きな影響を与えることが明らかとなった14)。MPIase非存在下ではSecYEGはSecEを接触面とした二量体 ('back-to-back' 構造、図4左) を形成することがX線結晶構造解析15,16)やSecEのシステイン置換変異体 (SecE 106C) を用いた解析17)から明らかになっている。このとき、SecGは二量体の両極に存在する (図4左)。そのため、SecGのシステイン変異体 (SecG 60C) を含むSecYEGでは、SecGの架橋二量体は形成できない。しかし、このSecYEGにMPIaseを共存させるとSecGの架橋二量体が形成できるようになった。逆に、SecE 106Cを含むSecYEGでは、MPIaseがないとき架橋二量体が形成できるが、MPIase存在下では形成できなくなった。このことは、MPIase存在下でSecYEG二量体は'back-to-back' 構造から'side-by-side' 構造に変化したと考えられる (図4)。SecGは膜内配向性反転サイクルを伴う大きな構造変化によりタンパク質膜透過反応を促進する18)が、SecYEGのみではこのサイクルが作動しないことが報告されている19)。しかし、SecYEGとMPIaseが共存したとき、SecGサイクルが作動することが判明した14)。すなわち、MPIaseによりSecYEG二量体構造が変化し、その結果SecGサイクルが作動可能となって膜透過活性が10倍以上促進される。このとき、MPIaseはSecYEGを活性化する補酵素のような役割を果たすことが考えられる。

おわりに
YidCはSecYEGと相互作用し20)、タンパク質膜挿入に関与する因子である21)。YidCは、膜タンパク質のバイオジェネシスに関与することが知られているミトコンドリアのOxa1pや葉緑体Alb3pと相同的であり22)、YidC枯渇株では膜タンパク質の機能発現に大きな欠陥が生じ致死的となる21)。しかし、YidCを枯渇した株から調製した膜小胞や、精製YidCを用いた再構成系では、タンパク質膜挿入反応におけるYidCの重要性を示す結果はほとんど報告されていない。YidCが "membrane protein insertase" であるという報告もある10)が、少なくとも自発的膜挿入を完全に排除したシステムではYidCだけでは膜挿入反応は全く進行しない5)。これらの報告を総合すると、膜挿入反応においては、まず、MPIaseが機能し、その後YidCに受け渡されて膜挿入が完了する可能性が考えられる。この可能性については今後検証する必要があるが、結晶構造に基づくYidCの機能モデル23)に図3のMPIaseの作用モデルを追加して考えるのは妥当であるように思われる。
タンパク質膜挿入や膜透過の基本的な分子機構はあらゆる生物で保存されている。MPIaseは大腸菌で発見された因子であるが、膜挿入・膜透過の保存性を考えると、高等動植物にもMPIaseと同様の機能をもつ物質が存在する可能性が高いと考えている。MPIaseは生化学的に同定された因子であるため、その生合成機構や遺伝子は全く不明である。生合成遺伝子が同定でき、逆遺伝学的な解析が進めば、大腸菌以外でどの程度普遍的にMPIaseが保存されているのか明らかになると期待できる。
筆者らは、タンパク質膜挿入活性を触媒する因子の精製を進め、結果的に糖脂質MPIaseの同定に至った。糖脂質ではあるものの酵素様の活性を示すことから、「糖脂質酵素 (Glycolipozyme) 」という概念を提唱している。この概念について酵素学的な見地からご批判いただければ幸いである。
謝辞
MPIaseに関する研究は、Freiburg大学Matthias Muller先生、東大分生研 (当時) 徳田元先生、サントリー生有研 (当時) 楠本正一先生、東大新領域上田卓也先生らとの共同研究により進めることができました。諸先生方に深く感謝いたします。また、MPIase研究に参画してくださった研究員、学生諸氏、特にMPIaseの精製や構造決定に深く関わっていただいた前田将秀博士に感謝いたします。
文献
1) Blobel, G., Walter, P., Chang, C. N., Goldman, B. M., Erickson, A. H., Lingappa, V. R.: Symp. Soc. Exp. Biol., 33, 9 (1979).
2) Koch, H. G., Moser, M., Muller, M.: Rev. Physiol. Biochem. Pharmacol., 146, 55 (2003).
3) Koch, H. G., Hengelage, T., Neumann-Haefelin, C., MacFarlane, J., Hoffschulte, H. K., Schimz, K .L., Mechler, B., Muller, M.: Mol. Biol. Cell, 10, 2163 (1999).
4) Koch, H. G., Muller, M.: J. Cell Biol., 150, 689 (2000).
5) Nishiyama, K., Ikegami, A., Moser, M., Schiltz, E., Tokuda, H., Muller, M.: J. Biol. Chem., 281, 35667 (2006).
6) Geller, B. L., Wickner, W.: J. Biol. Chem., 260, 13281 (1985).
7) Kawashima, Y., Miyazaki, E., Muller, M., Tokuda, H., Nishiyama, K.: J. Biol. Chem., 283, 24489 (2008).
8) Kuhn, A.: FEMS Microbiol. Rev., 17, 185 (1995).
9) Kiefer, D., Kuhn, A.: EMBO J., 18, 6299 (1999).
10) Serek, J., Bauer-Manz, G., Struhalla, G., van den Berg, L., Kiefer, D., Dalbey, R., Kuhn, A.: EMBO J., 23, 294 (2004).
11) Nishiyama, K., Maeda, M., Abe, M., Kanamori, T., Shimamoto, K., Kusumoto, S., Ueda, T., Tokuda, H.: Biochem. Biophys. Res. Commun., 394, 733 (2010).
12) Nishiyama, K., Maeda, M., Yanagisawa, K., Nagase, R., Komura, H., Iwashita, T., Yamagaki, T., Kusumoto, S., Tokuda, H., Shimamoto, K.: Nat. Commun., 3, 1260 (2012).
13) Kuhn, H. M., Meier-Dieter, U., Mayer, H.: FEMS Microbiol. Rev., 4, 195 (1988).
14) Moser, M., Nagamori, S., Huber, M., Tokuda, H., Nishiyama, K.: Proc. Natl. Acad. Sci. U. S. A., 110, 9734 (2013).
15) Breyton, C., Haase, W., Rapoport, T. A., Kuhlbrandt, W., Collinson, I.: Nature, 418, 662 (2002).
16) Zimmer, J., Nam, Y., Rapoport, T. A.: Nature, 455, 936 (2008).
17) Kaufmann, A., Manting, E. H., Veenendaal, A. K., Driessen, A. J., van der Does, C.: Biochemistry, 38, 9115 (1999).
18) Nishiyama, K., Suzuki, T., Tokuda, H.: Cell, 85, 71 (1996).
19) van der Sluis, E. O., van der Vries, E., Berrelkamp, G., Nouwen, N., Driessen, A. J.: J. Bacteriol., 188, 1188 (2006).
20) Scotti, P. A., Urbanus, M. L., Brunner, J., de Gier, J. W., von Heijne, G., van der Does, C., Driessen, A. J., Oudega, B., Luirink, J.: EMBO J., 19, 542 (2000).
21) Samuelson, J. C., Chen, M., Jiang, F., Moller, I., Wiedmann, M., Kuhn, A., Phillips, G. J., Dalbey, R. E.: Nature, 406, 637 (2000).
22) Kuhn, A., Stuart, R., Henry, R., Dalbey, R. E.: Trends Cell Biol., 13, 510 (2003).
23) Kumazaki, K., Chiba, S., Takemoto, M., Furukawa, A., Nishiyama, K., Sugano, Y., Mori, T., Dohmae, N., Hirata, K., Nakada-Nakura, Y., Maturana, A. D., Tanaka, Y., Mori, H., Sugita, Y., Arisaka, F., Ito, K., Ishitani, R., Tsukazaki, T., Nureki, O.: Nature, 509, 516 (2014).