【トピックス】
ポリエステル分解酵素の構造と機能
粕谷健一、室井文篤、宋君哲、鈴木美和、橘熊野
1.はじめに
現代社会においてプラスチックは、木材や金属に代わる軽くて安定な材料として我々の生活に不可欠なものとなっている。このプラスチックの中には生物に対して感受性を示すものもある。既に1968年にはポリエステル連結型ポリウレタンの微生物曝露による物性低下が報告されている1)。1990年代以降はプラスチックの生物劣化は、「生分解性」として再注目されるようになった。プラスチックが生分解性を発現するかどうかは、主として酵素により加水分解されるかどうかに起因することが多い。本トピックでは、潜在的な生分解性プラスチックとなりうるポリエステルに着目し、これを加水分解する酵素に焦点を当て解説する。
2.化学合成脂肪族ポリエステル分解酵素
化学合成脂肪族ポリエステルは1920年代にはすでにカローザスにより一通り合成されていたものの、高分子量体が得られず材料として顧みられることはなかった2)。脂肪族ポリエステルが材料として注目され始めたのは、上記ウレタン結合により高分子量体の脂肪族ポリエステルが得られるようになってからである3)。ポリエステル連結型ポリウレタンの生物劣化の主要因は、微生物の生産する酵素によるエステル部分の加水分解である。しかしながら化学合成脂肪族ポリエステルは、もともと天然には存在しないものであり、環境中で何らかの酵素により加水分解を受ける際には、当該酵素の本来の基質のアナログとして認識され分解を受けることとなる。
真菌Rhizopus 属由来のリパーゼ (EC 3.1.1.1) は脂肪族ポリエステルおよび脂環族ポリエステルを分解し、芳香族ポリエステルを分解しなかったことから、本来、脂肪 (トリアシルグリセロール) を分解する酵素であるリパーゼは脂肪族ポリエステルの加水分解酵素でもあることが示された4)。また、16種の真菌および細菌由来のリパーゼを用いて、ポリヒドロキシアルカン酸 (PHA) に対する基質特異性を調べたところ、リパーゼは側鎖の無いPHAに対して幅広い基質特異性を示したが、側鎖のあるPHAであるポリ (3-ヒドロキシブタン酸) (P(3HB)) を全く分解できなかった5)。このように、リパーゼは脂肪族ポリエステルの主鎖方向に対しては緩い基質認識を示した。多くのリパーゼは、本来の基質であるトリアシルグリセロールを認識する際、その疎水ポケット内で疎水性相互作用によりアルキル鎖を弱く固定することが知られているが6)、脂肪族ポリエステルに対しても同様の基質認識を示しているものと推測することができる。一方でP(3HB) 分解酵素は、種々のPHAおよび脂肪族ポリエステルを基質とした場合、リパーゼと比較してより厳密な基質特異性を示し、P(3HB) を除くと、ポリ (3-ヒドロキシプロピオン酸) (P(3HP))、ポリ (4-ヒドロキシブタン酸) (P(4HB))、ポリエチレンスクシナート (PESu) とポリエチレンアジペート (PEA) を加水分解した7)。
PEAを分解する真菌Penicillium sp. 14-38)からPEA分解酵素が精製され、その性質が調べられている9)。PEA分解酵素の分子量は約25kDa、至適温度は45℃、至適pHは4.5であった。また、本酵素の活性はCa2+、およびCd2+により賦活化した。PEA分解酵素はPEA以外の脂肪族ポリエステルでは、側鎖の無いPHAであるポリ (ε-カプロラクトン) (PCL) を分解したが、側鎖のあるP(3HB) を分解しなかった。また、PEA以外のジオールとジカルボン酸からなる脂肪族ポリエステルに対しても分解活性を示した一方で、芳香族ポリエステルを分解しなかった。本酵素はリパーゼ活性を有しており、リパーゼの一種であると考えられている。
ポリブチレンサクシネート-co-アジペート (PBSA) は、Chromatium vinosum由来のリパーゼによって分解を受け、コハク酸、ブタンジオールから分子量834までのオリゴマーが水溶性分解物として溶出した10)。また、PBSAは、これ以外の種々の細菌由来リパーゼによっても酵素分解されることが報告されている11)。一方、自然環境中からPBSA分解細菌Acidovorax delafieldii BS-3が単離され、この株からPBSA分解酵素 (PbsA) が精製された12)。本酵素は、乳化PBSAと固体PBSAに対して分解活性を有していた。固体基質へ1%の界面活性剤MEGA-9を添加したところ、本酵素の分解活性は、著しく低下した。このことから、本酵素は固体基質を分解する際、基質に吸着してから加水分解する2段階反応により基質を分解していることが示唆された。PbsA遺伝子 (pbsA) をクローニングしたところ、本酵素は304アミノ酸残基からなりMoraxella sp. TA144を含む一部の放線菌が生産するリパーゼと比較的高い相同性を示したが、プロテオバクテリアが生産するリパーゼやその他のポリエステル分解酵素とは有為な相同性を示さなかった。また、PbsAには、一般的なリパーゼ活性が認められることから、リパーゼの一種であることが推定される13)。
Asperugillus oryzaeのゲノム上からPBSA分解酵素活性を有する酵素 (CutL1) の遺伝子がクローニングされた14)。CutL1は、クチクラを加水分解する酵素であるクチナーゼ (EC 3.1.1.74) と高い相同性を示した。また、水晶振動子マイクロバランス (QCM) 法による解析からCutL1は、両親媒性蛋白質HabAと複合体を形成し、PBSA基質表面に結合しながら加水分解していることがわかった。この複合体形成は、不均一系におけるPBSA酵素分解促進に関与している可能性がある15)。
ポリエチレンサクシネート (PESu) は、リパーゼによっても分解を受けるが、後述するPenicillium funiculosum由来のP(3HB)分解酵素 (PhaZPfu)16)など、細菌や真菌由来のP(3HB) 分解酵素によっても加水分解される7,18)。PhaZPfuは活性中心付近のサブサイト構造において3つの3HBモノマーユニットを固定することがわかっているが17)、PESuのエチレングリコール-コハク酸ユニットを3HBモノマーユニットの連鎖のアナロジーとして捉えることにより、特異的にPESuも分解するものと推測されている。また、環境中からPESu分解真菌を単離しPESu分解酵素を精製したところ、高いP(3HB) 分解活性を有していることがわかった18)。この結果は、P(3HB) 分解酵素が環境中でPESuを分解していることを示唆するものである。他方、PESuを分解する細菌の多くはP(3HB) を分解できないことから、環境中の細菌においてはPESuを分解する際に別の酵素を利用していると考えられる19)。
3.ポリヒドロキシアルカン酸 (PHA) 分解酵素
脂肪族ポリエステルであるPHAの中には、細菌によって貯蔵物質として生合成されるものがある。細菌の種類や加える炭素源により様々な側鎖の長さおよび化学構造の3-ヒドロキシアルカン酸ユニットが確認されている20)。モノマー単位の側鎖がメチル基で炭素数が4の3-ヒドロキシブタン酸ユニットからなるポリ (3-ヒドロキシブタン酸) (P(3HB)) は、もっとも代表的なPHAであり、自然環境中で最も生分解しやすい生分解性ポリエステルの一つである21-23)。様々な環境中でP(3HB)分解酵素 (EC 3.1.1.74) を生産する微生物が見つかっている24)。ところで、酵素の定義としてP(3HB) 分解酵素とは、短鎖 (C3-C5) のヒドロキシアルカン酸から構成されるPHA、すなわちポリ (3-ヒドロキシプロピオン酸) (P(3HP))、 P(3HB)、 ポリ (4-ヒドロキシブタン酸) (P(4HB))、 ポリ (3-ヒドロキシペンタン酸) (P(5HV)) を基質として、これらを加水分解する一群の酵素を指す25)。このEC番号内には物理的状態の異なる基質、すなわち菌体内PHA (未変性:非晶性) および菌体外PHA (変性:結晶性) に対して分解活性を示す、構造的特徴の異なる酵素が一括りとなっている。前者は細菌体内における代謝に関与する酵素であり、一方後者はいわゆる生分解性ポリエステル分解酵素を指している。従ってここでは、後者である菌体外P(3HB) 分解酵素について記述する。
現在までに、数多くのP(3HB) 分解微生物が単離されており、またこれらの微生物のP(3HB) 分解酵素が特徴付けられている25-47)。P(3HB) 分解酵素は、以下に示すような、いくつかの共通した生化学的性質を有している。(i) 分解酵素の分子量は33,000〜60,000の範囲にあり、単量体ポリペプチドから構成されている。(ii) 分解酵素の等電点は、中性よりアルカリ領域に多く存在する。 (iii) 分解酵素は、中性緩衝液中でDEAEのような陰イオン交換体とは、結合しない。一方ブチルトヨパールのような疎水性担体に対しては、非常に強く結合する。(iv) 分解酵素は、幅広いpH、温度あるいはイオン強度において安定である。一部の酵素は、温度変化に対しても耐性を示す。(v) ジイソプロピルフルオロリン酸 (DFP) あるいは、フェニルメチルスルフォニルフルオライド (PMSF) のような典型的なセリン加水分解酵素阻害剤に対して感受性を示す。
P(3HB) 分解酵素は、活性残基を含む触媒ドメインと疎水性P(3HB) 表面に結合するための基質結合ドメインから構成されている7,37,48-52)。また、2つのドメイン間には、リンカードメインが存在する。このマルチドメイン構造 (触媒ドメイン+基質結合ドメイン) は、セルラーゼやキチナーゼなどの不溶性高分子分解酵素においても一般に見られる53-55)。この構造は、不溶性高分子分解酵素が、固-液界面という不均一反応系において、効率的に基質を加水分解するために有効であると考えられている7)。触媒ドメインは、活性残基としてSer、 His、 およびAspの3残基を有しており、この内Ser残基は、リパーゼボックス様ペンタペプチド配列内に存在する。これらの構造的特徴よりP(3HB) 分解酵素は、リパーゼやエステラーゼと同様にセリン加水分解酵素の一種であることがわかる。Penicillium funiclosum由来P(3HB) 分解酵素 (PhaZPfu) は、現在までに報告されているP(3HB) 分解酵素の中で最も小さい分子量 (33254Da) を有している16,38)。PhaZPfuは、他のすべてのP(3HB) 分解酵素に見られるリンカードメイン、および基質結合ドメインを欠損していた16) (図1)。このためPhaZPfuは、他のマルチドメイン型P(3HB) 分解酵素と比較して著しく基質結合能が低く16,38)、さらにマルチドメイン型酵素に特有な自己阻害効果も本酵素では観察されなかった。一方で、いくつもの研究グループによって、菌体外P(3HB) 分解酵素の機能と高次構造との関係を明らかにするために酵素の結晶化が試みられてきたがうまくいかなかった。このことはP(3HB) 分解酵素のマルチドメイン構造と関係していると考えられている。そこで我々はシンングルドメイン構造のPhaZPfuに着目し、P(3HB) 分解酵素としてはX線結晶構造解析に初めて成功した17)。本酵素は触媒ドメイン中の活性残基がクレバスの中心にあり、クレバスの周辺に芳香族系のアミノ酸残基が集中して存在していた17) (図2)。このことから酵素-基質間の弱い結合は、これらの芳香族系アミノ酸残基とP(3HB) 表面における疎水的相互作用によりもたらされている可能性が示唆された。
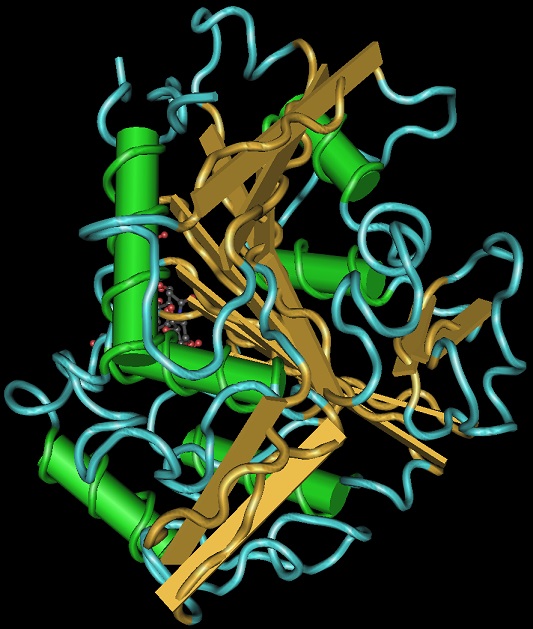
図1 Penicillium funiclosum由来のP(3HB)分解酵素(PhaZPfu)の立体構造(PDB, 2D30)


3HBオリゴマーを基質とした分解実験から、Ralstonia pickettii T1由来P(3HB) 分解酵素 (PhaZRpiT1) は4つのサブサイトを有していると推定されている56)。P(3HB) 分解酵素のポリエステルに対する基質特異性はリパーゼのそれと比較するとはるかに厳密で、基質ポリエステルの側鎖はメチル基以下であり、かつ酸素原子間の主鎖の炭素数は3あるいは4である条件を満たす場合のみ分解できる7)。このような厳しい基質特異性は、P(3HB) 分解酵素のサブサイトが、基質と強い相互作用で結ばれていることを示唆している。一方、リパーゼの幅広い基質特異性は、疎水ポケット内における基質認識において、疎水的相互作用等の弱い相互作用が原因であると考えられる6)。前述のPhaZPfuの高次構造解析からPhaZPfuは静電的相互作用によって3HBモノマーユニットを固定している3つのサブサイト構造を有していることがわかった17)。P(3HB) 分解酵素の高い基質特異性は、このような共通して見られる構造的特徴に起因していることを示唆している。
Delftia acidovorans YM1609P由来 (3HB) 分解酵素およびPhaZRpiT1の基質結合ドメインとグルタチオン S-トランスフェラーゼ (GST) との融合蛋白質が作成され、その性質が調べられた。その結果、基質結合ドメインは、P(3HB) に対してだけでなく、酵素が分解活性を示さないPLAあるいは、PCL等のポリエステルにも結合したが、セルロース、キチン、ポリプロピレン等には結合しないことがわかった7)。このことより、基質結合ドメインがポリエステルと何らかの特異的な相互作用により結合していることが示唆された。また、同時に、基質結合ドメインが触媒ドメインと独立して基質認識をしていることも示された。また、PhaZRpiT1の基質結合ドメインとP(3HB) 分子との間の引力を原子間力顕微鏡で測定したところ、約100pNであることがわかっている。PhaZRpiT1の基質結合ドメインの441位のロイシン残基および443位のチロシン残基をそれぞれヒスチジンに置換させたところ、P(3HB) 分解活性が野生型と比較して著しく低下し、これらの残基がP(3HB) 表面との相互作用に直接関わっていることが示された57)。
4.おわりに
プラスチックが環境中で生分解するためには、プラスチックを分解できる酵素の存在が最低条件となる。エステル結合はポリエステル分解酵素の攻撃対象となるため、ポリエステルは潜在的な生分解性を有していると考えられる。しかしながら、一般的に芳香族系ポリエステルは、生分解性を示さないか著しく低いことが知られている。ポリエステルが実際の酵素の基質になり得るかどうかは、一次構造的特徴のみならず融点やガラス転移点など材料の物理的性質にも大きく影響されることがわかっている58)。つまりポリエステル分解酵素から得られる情報を生分解性材料の設計に活かすためには、材料自身の一次構造のみならず高次構造や物性も考慮する必要があると言える。
文献
1) Darby, R., Kaplan, A.: Appl. Microbiol., 16, 900 (1968).
2) Carothers, W. H.: Collected Papers of Wallace Hume Carothers, Interscience publishers, inc. (1940).
3) Fujimaki, T.: Polym. Degrad. Stab., 59, 209 (1998).
4) Tokiwa, Y., Suzuki, T.: Agric. Biol. Chem., 42, 1071 (1978).
5) Mukai, K., Doi, Y., Sema, Y., Kousuke, T.: Biotechnol. Lett., 15, 601 (1993).
6) Derewenda, Z. S.: Adv. Pro. Chem., 45, 1 (1994).
7) Kasuya, K., Ohura, T., Masuda, K., Doi, Y.: Int. J. Biological. Macromol., 24, 329 (1999).
8) Tokiwa, Y., Suzuki, T.: J. Ferment. Technol., 52, 393 (1974).
9) Tokiwa, Y., Suzuki, T.: Agric. Biol. Chem., 41, 265 (1977).
10) Kitakuni, E., Yoshikawa, K., and et al.: Environ. Toxicol. Chem., 20, 941 (2001).
11) Ando, Y., Yoshikawa, K., and et al.: Polym. Degrad. Stab., 61, 129 (1998).
12) Uchida, H., Nakajima-Kambe, Y., and et al.: FEMS Microbiol. Lett., 189, 25 (2000).
13) Uchida, H., Shigeno-Akutsu, Y., and et al.: J. Biosc. Bioeng. Lett., 93, 245 (2002).
14) Maeda, H., Yamagata, Y., Abe, K., and et al.: 2005. Appl. Microbiol. Biotechnol., 67, 778 (2005).
15) Ohtaki, S., Maeda, H., and et al.: Appl. Envuron. Microbiol., 72, 2407 (2006).
16) Kasuya, K., Tezuka, Y., Ishii, N., and et al.: Macromol. Sympo. Microbiol., 249-250, 540 (2007).
17) Hisano, T., Kasuya, K., Tezuka, Y., Ishii, N., and et al.: J. Molecul. Biol., 356, 993 (2006).
18) Ishii, N., Inoue, Y., Shimada, K., and et al.: Polym. Degrad. Stab., 92, 44 (2007).
19) Tezuka, Y., Ishii, N., Kasuya, K., Mitomo, H.: Polym. Degrad. Stab., 84, 115 (2004).
20) Gao, X., Chen, J-C., Wu, Q., Chen, G-Q.: Cur. Opin. Biotechnol., 22, 768 (2011).
21) Luzier, W. D.: Proc. Natl. Acad. Sci. USA, 89, 839 (1992).
22) Nishida, H., and Tokiwa, Y.: J. Environment., Polym., Degrad., 1, 227 (1993).
23) Kasuya, K., Takagi, K., and et al.: Polym. Degrad. Stab., 59, 327 (1998).
24) Jendroseek, D., Shirmer, A., Schlegel, H. G.: Appl. Microbiol. Biotechnol., 46, 451 (1996).
25) http://www.chem.qmul.ac.uk/iubmb/enzyme/
26) Lusty, C. J., Doudoroff, M.: Biochemistry, 56, 960 (1966).
27) Tanio, T., Fukui, T., and et al.: Eur. J. Biocem., 124, 71 (1982).
28) Nakayama, K., Saito, T., Fukui, T., and et al.: Biochim. Biophy. Acta., 827, 63 (1985).
29) Jendrossek, D., Knoke, I., and et al.: J. Environ. Polym. Degrad., 1, 53 (1993).
30) Mukai, K., Yamada, K., Doi, Y.: Polym. Degrad. Stab., 41, 85 (1993).
31) Brucato, C. L., Wong, S. S.: Arch. Biochem. Biophy., 290, 497 (1991).
32) Kasuya, K., Doi, Y., Yao, T.: Polym. Degrad. Stab., 45, 379 (1994).
33) Mukai, K., Yamada, K., Doi, Y.: Polym. Degrad. Stab., 43, 319 (1994).
34) Kita, K., Ishimaru, M., and et al.: Appl. Environ. Microbiol., 61, 1727 (1995).
35) Oda, Y., Osaka, H., Urakami, T., Tonomura, K.: Curr. Microbiol., 34, 230 (1997).
36) Uefuji, M., Kasuya, K., Doi, Y.: Polym. Degrad. Stab., 58, 275 (1997).
37) Kasuya, K., Mitomo, H., and et al.: Biomacromol., 2, 194 (2000).
38) Miyazaki, S., Takahashi, K., Shiraki, M., and et al.: J. Polym. Environ. Polym., 8, 175 (2000).
39) Takade, M., Kitashima, and et al.: J. Biosc. Bioeng., 90, 416 (2000).
40) Kim, D. Y., and Rhee, Y. H.: Appl. Microbiol. Biotechnol., 61, 300 (2003).
41) Romen, F., Reinhardt, S., Jendrossek, D.: Arch. Microbiol., 182, 157 (2004).
42) Calabia, B., and Tokiwa, Y.: Biotechnol. Lett., 28, 383 (2006).
43) Papaneophytou, C., Pantazaki, A., Kyriakidis, D.: Appl. Microbiol. Biotechnol., 83, 659 (2009).
44) Bhatt, R., Patel, K. C., Trivedi, U.: J. Polym. Environ. Polym., 18, 141 (2010).
45) Garcia-Hidalgo, J., Hormigo, D., Prieto, M., Arroyo, M., Mata, I.: Appl. Microbiol. Biotechnol., 93, 1975 (2012).
46) Wang, Z., Gao, J., Li, L., Jian, H.: World J. Microbiol. Biotechnol., 28, 2395 (2012).
47) Gracia-Hidalgo, J., Hoemigo, D., Arroyo, M., de la Mata I.: PLoS One, 12 (2013).
48) Jendrossek, D., Bakhous, M., Andermann, M.: Can J. Microbiol., 41, 160 (1995).
49) Shinomiya, M., Iwata, T., Kasuya, K., Doi, Y.: FEMS Microbiol. Lett., 154, 89 (1997).
50) Kasuya, K., Inoue, Y., and et al.: Appl. Environ. Microbiol., 63, 4844 (1997).
51) Ohura, T., Kasuya, K. Doi, Y.: Appl. Environ. Microbiol., 65, 189 (1999).
52) Kasuya, K., Takano, T., and et al.: Int. J. Biol. Macromol., 33, 221 (2003).
53) Gilkes, N. R., Henrissat, B., Kilburn, D. G., Miller, R. C., Warren, R. A.: Microbiol. Rev., 55, 303 (1991).
54) Hansen, Christian K.: FEBS Lett., 305, 91 (1992).
55) Bork, P., Doolittle, R. F.: Proc. Nat. Sci., USA, 89, 8990 (1992).
56) Shirakura, Y., Fukui, T., Saito, T., and et al.: Biochimi. Biophy. Acta, 880, 46 (1986).
57) Matsumoto, N., Fujita, M., and et al.: Biomacromol., 9, 3201 (2008).
58) Marten, E., Muller, R-J., Deckwer, W-D.: Polym. Degrad. Stab., 88, 371 (371).