【トピックス】
カイメンから見出された多様な二次代謝産物を生産できる新規細菌分類群
モリ テツシ、竹山春子、Micheal C. Wilson、Joern Piel
早大・理工、早大・理工、スイス工科大・チューリッヒ校、スイス工科大・チューリッヒ校
1.はじめに
細菌はあらゆる環境に生息し、それぞれの環境に適応するために独自の進化を繰り返しなから様々な機能を獲得してきた。種によっては、生息している環境に重要な役割を担っていることもある。また、生物生体内にも多くの細菌が生息しており、宿主と共生している例が多く報告されている。海洋生物からも多くの細菌が共在・共生していることが発見されており、これらの細菌は様々な生理活性物質や有用化合物の遺伝子資源として注目されている。海洋生物中、特に無脊椎動物であるカイメン (ポリフェラ門、約15,000種) からは、産業や医療分野において非常に重要な生理活性を持つ二次代謝産物であるポリケチド、抗生物質1)、抗腫瘍物質2)、免疫抑制剤3)が多く発見されている。カイメンは生態系のなかで生き残るための化学防衛機構を備えている事が知られている4)。カイメンが多数の細菌と共生関係にあることから、近年ではカイメンの化学防衛機構に関わっている生理活性物質や二次代謝産物が共生細菌由来であると考えられている。そして、このような多くの有用物質やこれらを合成する新種のポリケチドシンターゼ (PKS) または非リボソームペプチドシンターゼ (NRPS) がカイメン共生細菌から発見されている5-7)。しかしながら、カイメン共生細菌には難培養細菌が多く、未だに謎の部分が多い。
2.カイメン難培養共生細菌群からの新規“万能”細菌の発見
今までは、細菌から単離された二次代謝産物等の生理活性物質の大半は、単離培養可能な細菌群である、Actinomycetes (放線菌)、Myxobacteria (粘液細菌)、CyanobacteriaそしてPseudomonasおよびBacillus属の細菌に限定されており、新たな生産菌が求められている。そこで、新規の生理活性物質を得るために、近年では新たな探索資源として、難培養細菌も注目されている。その主な理由としては、難培養細菌叢は非常に多様であり、幅広い分類群に分布していることから、新規かつ高活性な生理活性物質生産能を保有する細菌が存在する可能性が期待できる点である。最近の遺伝子配列決定技術の飛躍的な進歩により、難培養細菌を含めた環境中のゲノムDNA (メタゲノム) が、迅速、かつ大規模に、そして比較的安価、に解析できるようになった8)。そこでこれらの解析アプローチにより、新規な二次代謝産物の生合成遺伝子クラスターの発見および同定報告が増えてきている9,10)。しかしながら、メタゲノムを用いた研究においては、生合成遺伝子クラスターの取得は可能だが、その生合成遺伝子クラスターの生産菌の情報は得られないといった欠点が挙げられる。この問題を解決するためには、単一細胞レベルでの解析が必要不可欠である11)。単一細胞の分取、解析においては、フローサイトメトリーやマイクロ流体デバイスなどが活用されており、動物細胞から真核微生物そして細菌まで幅広い対象で研究が推し進められている。しかし、環境中の難培養細菌からの二次代謝産物の生産菌について単一細胞解析技術を用いた報告はまだなされていない。
筆者らは、カイメン内に生息する共生細菌に対し、単一細胞またはメタゲノム解析でのアプローチを利用した、新たな細菌種の探索を目的とした研究をスタートした12)。本研究では、すでに多くの二次代謝産物が発見されているカイメン、Theonella swinhoei Y (黄色ケモタイプ) を対象とし、このカイメンに共生している細菌叢から二次代謝産物を生産する細菌の検出と解析を試みた (図1)。
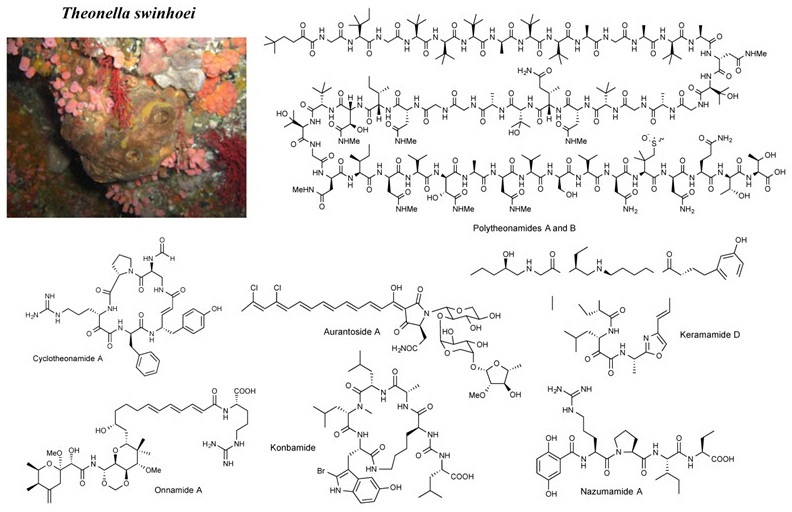
![]()
2-1 単一細胞解析を用いたT. swinhoei Yからの二次代謝産物生産菌の同定
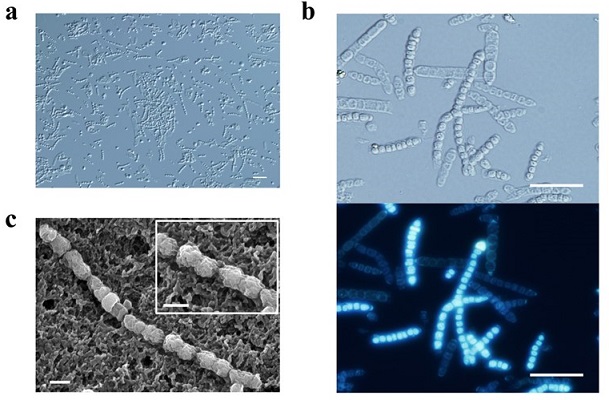
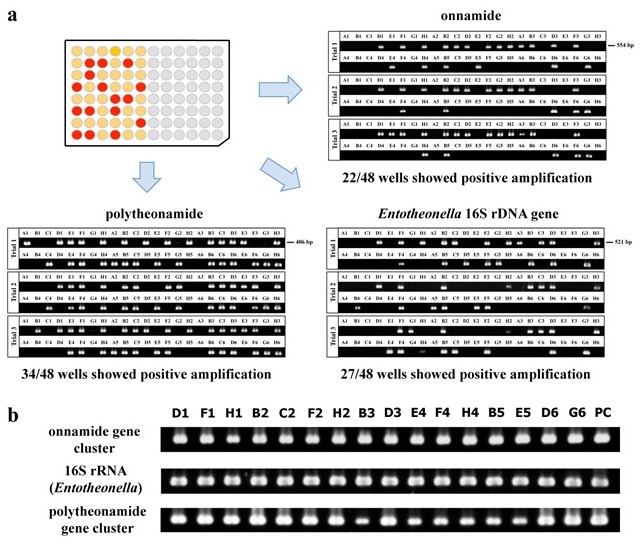
onnamide (onn)13)やpolytheonamide (poy)14)など多様な二次代謝産物が、T. swinhoei Yから分離されている。これらを生産する菌が、単一種に由来するのかを決定するには、生合成遺伝子クラスターの有無を単一細胞レベルで確認することが必要である。T. swinhoei Yには、単細胞と糸状形態の細菌が見られる。そこで、八丈島で採集したT. swinhoei Yの内部組織を破壊し、カイメン破砕物を自然沈降させ、上清を遠心分離したところ、ペレット画分には主に糸状性細菌、上清画分には単細胞性細菌が分画された。ペレット画分で観察された糸状性細菌の形態はパラオで採集されたT. swinhoeiの共生細菌であるCandidatus Entotheonella palauensis (Candidatus: 難培養を示す)15)と非常に似ていた (図2)。また、Candidatus Entotheonella palauensisはT. swinhoeiから検出される抗菌物質の真の生産者である可能性が示唆されている16)。引き続き、分画された糸状性細菌をフローサイトメーターで96穴ウェルプレートに分取した。分取した単一細胞 (糸状体なので厳密には単一細胞とは言えないが、一糸状体を構成している細胞はクローンと考え、ここでは単一細胞としている) を破砕し、Multiple displacement amplification (MDA) 法17)により全ゲノム増幅を行った。ここで、onnおよびpoy生産共生細菌の同定を行うために、増幅ゲノムに対し、Nested-PCRによるonnおよびpoyの合成PKS遺伝子断片の増幅と16S rDNAの増幅、解析を行った (図3)。48ウェルの内onnの増幅が確認されたウェルは22ウェル、またpoyの増幅が確認されたのは34ウェルだった。更に、同じプレートについて16S rDNA解析を行ったところ、27ウェルがEntotheonella sp.由来だと判明した。結果的に同じウェルからonn、poy、およびEntotheonellaの16SrDNA遺伝子が発見されたウェルの数は16であった。この結果より、onnamideおよびpolytheonamideは同一の細菌から生産されること、そして、その生産菌はEntotheonella sp.であることが示された (図3)。


2-2 Entotheonella sp.の“万能性”を探る
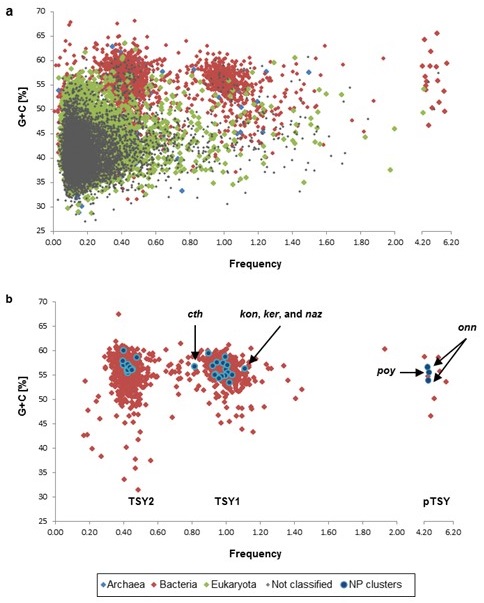
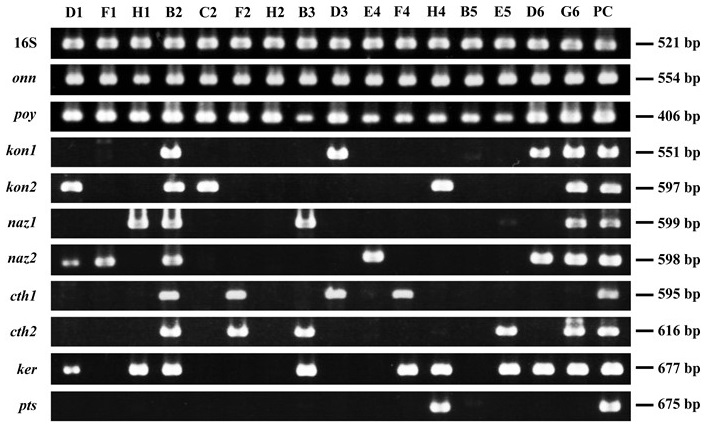
本研究での単一細胞解析によって発見されたEntotheonella sp.菌体が2種類の二次代謝産物を生産できることから、この細菌の“万能性”をメタゲノム解析で確認した。メタゲノム解析は次世代シーケンサー、Illumina、PacBio、454およびSanger法で行い、サンプルは糸状性細菌が多く観察された画分を使用した。シーケンスの結果により、得られたリード数の78.3%が18,093個の長鎖のDNAコンティグにアセンブルされた。更に、これらのDNAコンティグ領域の出現頻度やG/C含量を基準に解析を行ったところ、非常に似た2種類の細菌ゲノムの存在が見出された (図4)。それぞれのゲノムから取得した16S rDNA遺伝子情報を検討したところE. palauensisと高い相同性 (約97%) を示した。また、この2細菌種間の16S rDNA遺伝子の相同性は97.6%であった。結果的にメタゲノム解析により、同等なゲノムサイズ (約9 Mbp) を保有している2種類のEntotheonella株が確認され、それぞれをTSY1 (優占種) およびTSY2株と名付けた。次に、得られたメタゲノム情報から、二次代謝産物関連の生合成遺伝子クラスターを探索した結果、28種類の生合成遺伝子クラスターが同定された。この28種類の中から、すでにT. swinhoeiから活性が確認されたcyclotheonamide (cth)、keramamide (ker) およびnazumamide A (naz) の生合成遺伝子クラスターと完全に一致した。ここで、それぞれの生合成遺伝子クラスターの存在を確認するために、onnおよびpoy生合成遺伝子クラスターが同定されたプレートからcth、ker、naz、konbamide (kon)、そして以前発見されたNRPS生合成遺伝子クラスターproteusin (pro) をNested-PCRで確認した。その結果、onn、poy、Entotheonella 16S rDNA遺伝子の増幅が確認されたウェルから、これらの遺伝子クラスターの存在が認められた (図5)。更に、メタゲノムの解析結果、cthの生合成遺伝子クラスターはTSY1株のゲノムに存在することが確認された。このことは、T. swinhoeiから活性が確認された二次代謝産物のほとんどがTSY1株由来であることを示すとともに、その“万能性”を支持するものである。
![]()

3.おわりに
本研究では、単一細胞やメタゲノム解析技術を組み合わせて応用することにより、筆者らは主に難培養細菌から形成されるカイメンの共生細菌叢から初めて新規な“万能”細胞の発見・同定に成功した。これらの細菌の培養を可能にすれば、新たな薬品の生産も期待できる。本研究は、単一細胞およびメタゲノム解析技術の応用、そして難培養細菌に着目したことで、二次代謝産物の分野において、長年課題となっていた新薬発見・創薬の問題解決に資するものである。
文献
1) De Silva, E. D., Scheuer, P. J.: Tetrahedron Lett., 22, 3147 (1981).
2) Hirata, Y., Ohya, S.: Pure Appl. Chem., 58, 701 (1986).
3) Gunasekera, S. P., Gunasekera, M., Longley, R. E., Schulte, G. K.: J. Org. Chem., 55, 4912 (1990).
4) Thomas, T. R., Kavlekar, D. P., LokaBharathi, P. A.: Mar. Drugs, 8, 1417 (2010).
5) Piel, J., Hui, D., Wen, G., Butzke, D., Platzer, M., Fusetani, N., Matsunaga, S.: Proc. Natl. Acad. Sci. U S A, 101, 16222 (2004).
6) Kim, T. K., Fuerst, J. A.: Environ. Microbiol., 8, 1460 (2006).
7) Fisch, K. M., Gurgui, C., Heycke, N., van der Sar, S. A., Anderson, S. A., Webb, V. L., Taudien, S., Platzer, M., Rubio, B. K., Robinson, S. J., Crews, P., Piel, J.: Nat. Chem. Biol., 5, 494 (2009).
8) Handelsman, J.: Microbiol. Mol. Biol. Rev., 68, 669 (2004).
9) Gurgui, C., Piel, J.: Methods Mol. Biol., 668, 247 (2010).
10) Freeman, M. F., Gurgui, C., Helf, M. J., Morinaka, B. I., Uria, A. R., Oldham, N. J., Sahl, H. G., Matsunaga, S., Piel, J.: Science, 338, 387 (2012).
11) Templer, R. H., Ces, O.: J. R. Soc. Interface, 5 Suppl 2, S111 (2008).
12) Wilson, M. C., Mori, T., Ruckert, C., Uria, A. R., Helf, M. J., Takada, K., Gernert, C., Steffens, U. A., Heycke, N., Schmitt, S., Rinke, C., Helfrich, E. J., Brachmann, A. O., Gurgui, C., Wakimoto, T., Kracht, M., Crusemann, M., Hentschel, U., Abe, I., Matsunaga, S., Kalinowski, J., Takeyama, H., Piel, J.: Nature, 506, 58 (2014).
13) Atsunaga, S., Fusetani, N., Nakao, Y.: Tetrahedron, 48, 8369 (1992).
14) Hamada, T., Matsunaga, S., Yano, G., Fusetani, N.: J. Am. Chem. Soc., 127, 110 (2005).
15) Schmidt, E. W.: Nat. Chem. Biol., 4, 466 (2008).
16) Schmidt, E. W., Obraztsova, A. Y., Davidson, S. K., Faulkner, D. J., Haygood, M. G.: Marine Biology, 136, 969 (2000).
17) Binga, E. K., Lasken, R. S., Neufeld, J. D.: Isme Journal, 2, 233 (2008).