【トピックス】
酵母の新規アシル基転移酵素の同定と機能解析
玉置尚徳、中山玲子、熊谷英彦
鹿児島大・農、京女大・家政、石川県大
1.はじめに
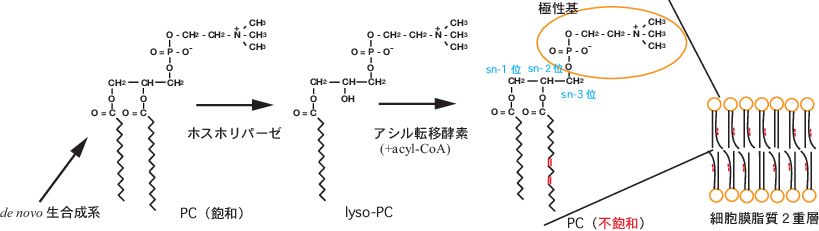
細胞膜は半透性の脂質2重層からなり、これによって細胞質と外環境を隔てている。細胞膜の主な構成成分であるリン脂質には著しい構成脂肪酸の偏りが知られている。グリセロリン脂質では、一般にグリセロール骨格1位 (sn-1位) には飽和脂肪酸が多く、sn-2位には不飽和脂肪酸が多い。また、グリセロリン脂質のsn-3位にある極性基 (コリン、エタノールアミン、イノシトール、セリンなど) の種類によってsn-2位の脂肪酸組成は著しく異なる。このような脂肪酸種の不均衡な分布は、リン脂質のde novo合成経路1) では説明できないことから、脂質のリモデリング2) によって形成されると考えられている。脂質のリモデリングとは、de novo合成系によって生成されたグリセロリン脂質が、sn-2位の脂肪酸の切断を受けリゾリン脂質となった後、新たな脂肪酸の導入を受けるというシステムである (図1)。前者を触媒するのがホスホリパーゼなどのエステル分解酵素であり、後者を触媒するのがアシル基転移酵素と考えられている。しかしながら、これらの酵素とリモデリングの関連はほとんど解明されておらず、特にリゾリン脂質を受容体基質とするアシル基転移酵素に関しては全く同定されていなかった。本稿では、筆者らが出芽酵母において同定した新規なアシル基転移酵素の性質と機能に関して紹介する3)。

2.血小板活性化因子合成酵素遺伝子の同定
筆者らは、もともと血小板活性化因子 (Platelet activating factor; PAF) 合成酵素の同定をめざして研究を進めていた。PAFは、1位にアルキル型脂肪酸、2位にアセチル基、3位にホスホコリンが結合したグリセロリン脂質であり、哺乳類において微量で血小板の凝集や血管の収縮などを引き起こす生理活性脂質として見出された4)。PAFの生理活性には2位のアセチル基が必須であり、PAF分解酵素によってこのアセチル基が加水分解されると生理活性は失われる。一方PAFの合成は、2位にアセチル基を持たないLyso-PAFにacetyl-CoAからアセチル基を転移することで行われると考えられていた。PAFの強力な生理活性やアレルギーとの関連から、PAF合成酵素同定の試みは主に哺乳類において行われていたが、膜酵素であることと可溶化により不安定化することから精製は成功していなかった。筆者らは酵母においてもPAFが産生されることを見出し5)、PAF合成酵素活性が酵母でも認められることを見出した。当初、酵母の大量培養菌体より膜画分を調製し、可溶化後に部分精製を行い、アミノ酸配列の決定を試みたが、ついに成功しなかった。
出芽酵母のゲノム解析は1996年に完了し6)、いち早くポストゲノム時代に突入していた。そして数年のうちに酵母を用いた様々な研究ツールが発明されてきた。その1つが、酵母遺伝子破壊株セットであった。これは、出芽酵母のもつ約6000の遺伝子のうち破壊しても致死とならない4700あまりの遺伝子をそれぞれ破壊した株のセットである。
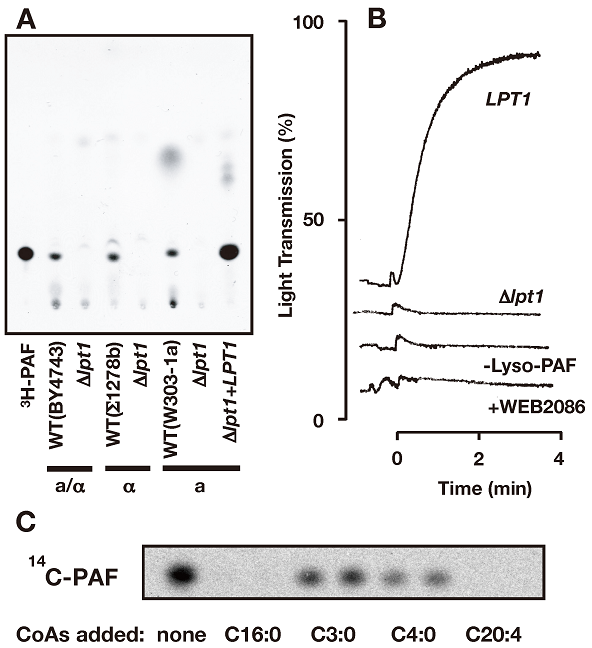
筆者らは、この4700あまりの遺伝子破壊株についてPAF合成酵素活性を測定し、酵素活性を示さない株をスクリーニングすることで本酵素遺伝子の同定を行った。PAF合成酵素活性の測定は、14Cでラベルしたacetyl-CoAとlyso-PAFを基質として用い、反応生成物である14C-PAFを薄層クロマトグラフィーにて展開後、イメージングプレートを用いたオートラジオグラフィーにより検出することで行った。3次スクリーニングまで行った結果、PAF合成酵素活性が全く検出されない株を1株見出した。当該株で破壊されている遺伝子は、ORF番号YOR175cでmembrane bound O-acyltransferase (MBOAT) ファミリー7) に属する機能未知の遺伝子であった。本遺伝子をLPT1 (lysophospholipid acyltransferase 1) と命名し、PAF合成能の確認を行った。
まず、遺伝的背景の異なる3種類の酵母株においてLPT1遺伝子を破壊するとPAF合成活性は消失した。また、その破壊株にLPT1遺伝子を多コピープラスミドをもちいて導入すると野生株よりも高いPAF合成活性を示した (図2A)。次に、合成されたPAFが生理機能を示すかどうかを血小板の凝集活性により評価した。遺伝子破壊株にLPT1高発現ベクターを導入した株を用いて合成したPAFは、ウサギ由来血小板の凝集活性を示したが、LPT1遺伝子を持たないコントロールベクターを導入した場合には、血小板の凝集は認められなかった (図2B)。以上の結果より、LPT1はPAF合成酵素遺伝子であることが示された。次に筆者らは、様々な鎖長のアシル基を持つCoAを共存させた場合のPAF合成活性を調べた。その結果、アセチル基よりも少し鎖長の長いpropionyl-CoA (C4:0) やbutyryl-CoA (C3:0) を共存させるとPAF合成活性に低下が認められ、さらに鎖長の長いpalmitoyl-CoA (C16:0) やarachidonoyl-CoA (C20:4) が共存するとPAF合成活性は認められなくなった (図2C)。これらの結果は、酵素Lpt1は、PAF合成酵素というよりも長鎖のアシル基を好んで転移するリゾリン脂質アシル基転移酵素である可能性が考えられた。

3.新規リゾリン脂質アシル基転移酵素の性質
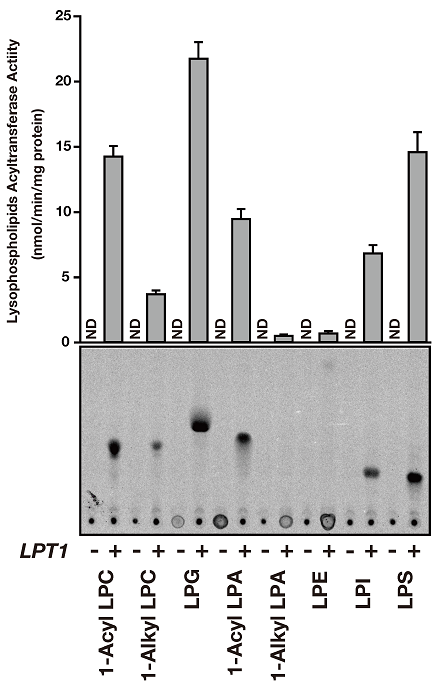
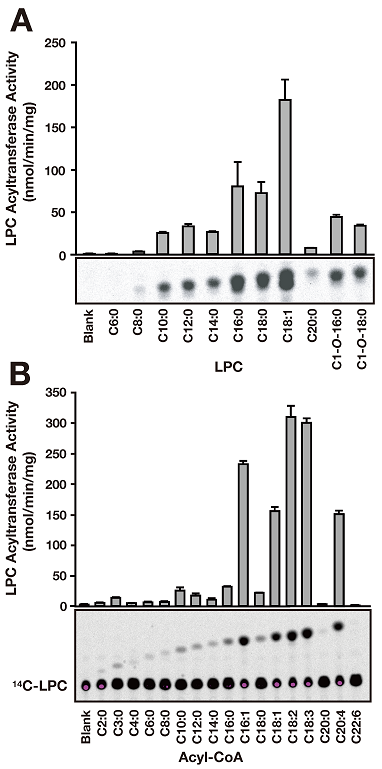
本酵素の基質特異性を調べることにした。本酵素は、膜酵素と考えられたことから、さまざまな可溶化剤を用いて可溶化および精製を試みたが、いずれの場合も可溶化により酵素活性が著しく低下したことから、酵素標品としては細胞膜を含むミクロソーム画分を用いることとした。すなわち、LPT1遺伝子破壊株にLPT1高発現ベクターを導入した株とコントロールベクターのみを導入した株よりそれぞれミクロソーム画分を調製し、アシル基供与体として [1-14C]linoleoyle-CoA、様々なリゾリン脂質をアシル基受容体として反応を行った (図3)。その結果、本酵素を高発現させた株ではlysophosphatidylcholine (LPC)、lysophosphatidylglycerol (LPG)、lysophosphatidic acid (LPA)、lysophosphatidylethanolamine (LPE)、lysophosphatidylinositol (LPI)、lysophosphatidylserine (LPS) を受容体基質とした酵素反応生成物が認められ、本酵素が極性基の異なるリゾリン脂質に対して幅広い基質特異性を有することが示された。特にsn-1位がアルキル基であるよりもアシル基を有するリゾリン質に高い特異性を示した。次に、[1-14C]linoleoyle-CoA をアシル基供与体、sn-1位のアシル基の種類の異なる様々なLPCを受容体基質として酵素反応を行い酵素活性を比較したところ、本酵素は1-hexanoyl-LPC (C6:0) 以外の様々なアシル基をもつリゾリン脂質を受容体基質として認識した (図4A)。特にsn-1位に中鎖のアシル基 (C16:0, C18:0, C18:1) を有するリゾリン脂質に高い特異性を示した。筆者らはさらに [1-14C]palmitoyl-LPCを受容体として本酵素のアシルCoAの選択性を調べた (図4B)。その結果、酵素Lpt1は中鎖アシルCoAを好み、中でも特に不飽和結合をもつものに高い特異性を示すことが明らかとなった。この結果は、本酵素が脂質のリモデリングに関与して細胞膜を構成するグリセロリン脂質への不飽和結合の導入に関わっている可能性を示唆している。


4.細胞内におけるアシル基転移酵素の機能
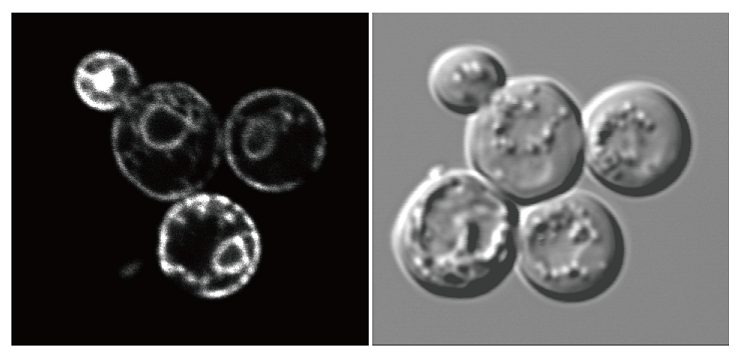
本酵素の活性は、ミクロソーム画分に見出されたことから本酵素は膜酵素であることが予想された。そこで、Lpt1の酵母細胞内における局在性を明らかにする目的でGFP遺伝子をLPT1遺伝子の3’末端に融合させたLPT1-GFPを酵母に導入し、蛍光顕微鏡を用いて観察を行った。その結果、Lpt1-GFPによる蛍光は核の周りの小胞体や細胞膜近傍の小胞体に認められた (図5)。

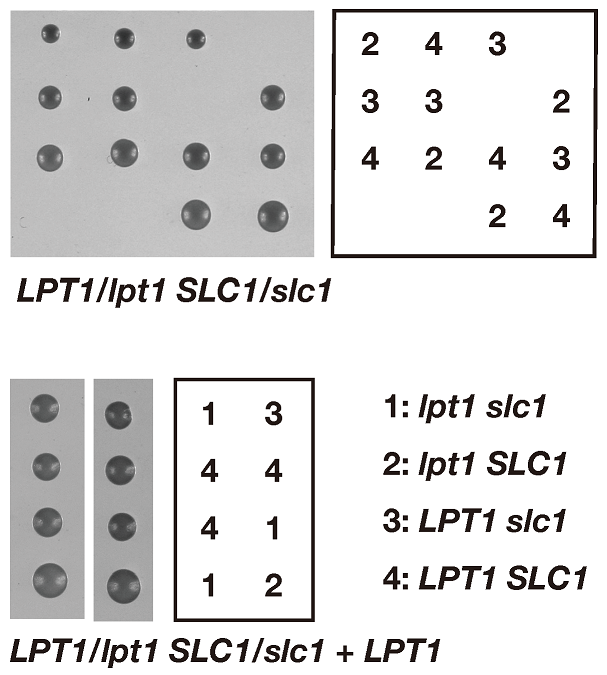
細胞膜を構成する様々なグリセロリン脂質は、すべてphosphatidic acid (PA) から合成される。酵母では、これまでにLPAアシル基転移酵素遺伝子としてSLC1が報告されていた8)。基質特異性の解析結果から、Lpt1もLPAにアシル基を転移する高い活性を示したことから、酵母細胞内におけるLpt1とSlc1の役割について検討を行った。LPT1遺伝子、SLC1遺伝子をそれぞれ単独で破壊した場合には、顕著な表現型の変化は認められなかった。そこで、異なる接合型の1倍体酵母において一方はLPT1遺伝子、もう一方はSLC1遺伝子を破壊した株を作成し、接合により遺伝子型が、(LPT1/lpt1SLC1/slc1) となった2倍体株を得た。本2倍体株を用いて胞子を形成させ1つの2倍体細胞に由来する4つの胞子をマイクロマニピュレーターを用いて分離し栄養寒天培地にてインキュベートした (四分子解析) (図6)。その結果、1つの細胞に由来する4つの胞子のうち発芽してコロニーを形成できない株が認められた。コロニーを形成した株の遺伝子型を調べると、(lpt1 SLC1)、(LPT1 slc1)、(LPT1 SLC1) のいずれかの遺伝子型の組み合わせを示したが、(lpt1 slc1) 遺伝子型を示すコロニーは認められなかった。この結果よりSLC1とLPT1の両方の遺伝子が欠失すると酵母は合成致死となることが考えられた。さらに、確認する目的で2倍体細胞 (LPT1/lpt1 SLC1/slc1) にLPT1遺伝子をプラスミドに載せて導入後、四分子解析を行ったところ (lpt1 slc1) の遺伝子型を示す株の胞子もコロニーを形成できた。このことよりLPT1とSLC1はPA合性に関して重複した役割を担っており、両遺伝子が機能しなくなると酵母は致死となることが明らかとなった。

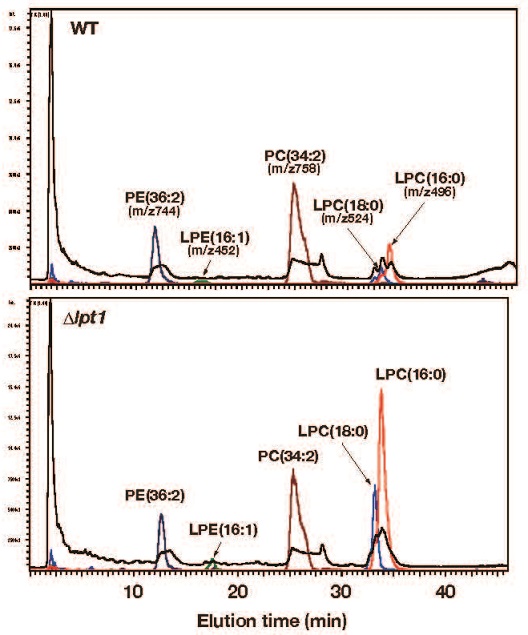
LPT1遺伝子の単独欠損株では、表現型の変化は認められなかったが、細胞内のグリセロリン脂質の組成に変化が生じていないか調べた。野生株とLPT1遺伝子破壊株の菌体より総脂質を抽出し液体クロマトグラフィー質量分析計 (LC-MS) による解析を行った。その結果、LPT1遺伝子破壊株では、PCやPEの量は、野生株と比べて変化は見られなかったが、LPCやLPEが、顕著に増加していることが示された (図7)。これらの結果から、Lpt1はLPCやLPEにアシル基を転移する主要な酵素であり、その遺伝子欠損のため著量のリゾリン脂質が細胞内に蓄積したものと結論づけた。

5.おわりに
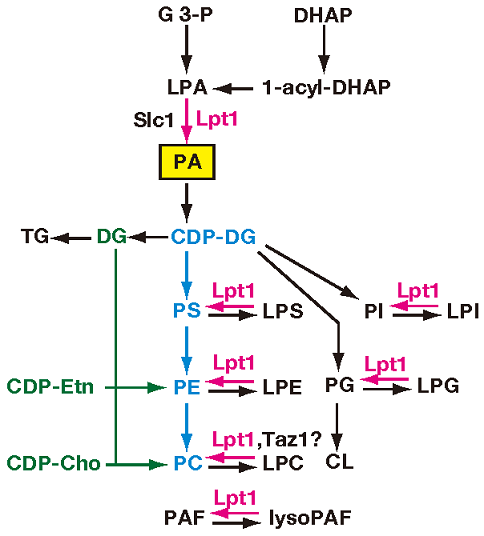
これまでの結果から、我々が見出した酵素Lpt1は、LPA、LPC、LPI、LPS、LPG、LPEなど様々なリゾリン脂質にアシルCoAからアシル基を転移する酵素であることが明らかとなった。酵母においてLpt1は、もう一つのアシル基転移酵素Slc1とともにPA合成に中心的役割を果たしていることが示された (図8)。脂質のリモデリングに関しては、1950年代に提唱された2) が、そのなかで不飽和結合を導入する酵素は、膜酵素でありその精製の難しさから遺伝子が同定されるまで50年以上の年月を要したことになる。筆者らが今回行った遺伝子破壊株のセットを用いたスクリーニングは、ポストゲノムのツールの有効な活用法であり、これまでに発見できなかった酵素や遺伝子を見出す新たな手段として有効であると考える。筆者らの同定した出芽酵母のLPT1に関しては,ほぼ同時に欧米の4つのグループからも報告され9-12)、遺伝子の名前もALE1/SLC4/LCA1と命名されており、激しい競争が展開されていたことがうかがえる。また、酵母での発見をきっかけに線虫、マウス、ヒトの遺伝子に関しても報告が行われ、本酵素が酵母からヒトに至るまで保存されている生物に普遍的な酵素であることが示された13)。

文献
1) Kennedy, E. P., Weiss, S. B.: J. Biol. Chem., 222, 193 (1956).
2) Lands, W. E.: J. Biol. Chem., 231, 883 (1958).
3) Tamaki, H., Shimada, A., Ito, Y., Ohya, M., Takase, J., Miyashita, M., Miyagawa, H., Nozaki, H., Nakayama, R., Kumagai, H.: J. Biol. Chem., 282, 34288 (2007).
4) Benveniste, J., Henson, P. M., Cochrane, C. G.: J. Exp. Med., 136, 1356 (1972).
5) Nakayama, R., Kumagai, H., Saito, K.: Biochim. Biophys. Acta, 1199, 137 (1994).
6) Goffeau, A., Barrell, B. G., Bussey, H., Davis, R. W., Dujon, B., Feldmann, H., Galibert, F., Galibert, J. D., Jacq, C., Johnston, M., Louis, E. J., Mewes, H. W., Murakami, Y., Philippsen, P., Tettelin, H., Oliver, S. G.: Science, 274, 546 (1996).
7) Hofmann, K.: Trends Biochem. Sci., 25, 111 (2000).
8) Athenstaedt, K., Daum, G.: J. Bacteriol., 179, 7611 (1997).
9) Riekhof, W. R., Wu, J., Jones, J. L., Voelker, D. R.: J. Biol. Chem., 282, 28344 (2007).
10) Benghezal, M., Roubaty, C., Veepuri, V., Knudsen, J., Conzelmann, A.: J. Biol. Chem., 282, 30845 (2007).
11) Jain, S., Stanford, N., Bhagwat, N., Seiler, B., Costanzo, M., Boone, C., Oelkers, P.: J. Biol. Chem., 282, 30562 (2007).
12) Chen, Q., Kazachkov, M., Zheng, Z., Zou, J.: FEBS Lett., 581, 5511 (2007).
13) Shindou, H., Shimizu, T.: J. Biol Chem., 284, 1 (2009).