【トピックス】
超好熱アーキア由来糖代謝酵素への変異導入による活性の促進
河原林 裕
九大院・農、産総研
1.はじめに
地球上には、極限環境と呼ばれるヒトが生存できないような過酷な環境が多数存在している。その中には、温泉や海底熱水の様な高温環境も含まれるが、近年これらの極限環境からも多くの微生物が単離されてきている。高温環境から単離された微生物は、最適生育温度が60℃から98℃と高温である。この温度で生育することができるということは、この温度で細胞内の様々な代謝を行える、言い換えると酵素がこの高温でも機能しているのである。この特性は、これまでの酵素・蛋白質に関する常識を打ち破るものであった。耐熱性酵素の中でも特に最も多く利用されているのが、アメリカ合衆国イエローストーン国立公園の高温プールより単離されたThermusaquaticus 1)から見出された耐熱性DNAポリメラーゼTaq DNAポリメラーゼである2)。高温でも安定性が高いこの酵素の出現により、PCR増幅中の二本鎖DNAの一本鎖への乖離の際の高温加熱後のDNA合成酵素の再添加が不要となり、PCR増幅技術は現在のような手軽な技術へと発展したのである。好熱性微生物では、このTaq DNAポリメラーゼを筆頭に全ての酵素の耐熱性・安定性が高いことから、様々な産業応用の期待も高い。
そこで、我が国でも1990年代半ばに当時の通商産業省 (現経済産業省) が主導する形で超好熱アーキアのゲノム解析が取り組まれ、Pyrococcus horikoshii 3)、Aeropyrum pernix 4)、Sulfolobus tokodaii 5)という3種のゲノムの全長塩基配列およびゲノム情報が公開された。このゲノム情報は多数の耐熱性酵素の宝庫であり、大腸菌等の異種微生物を宿主として用いて生産された蛋白質でも高い耐熱性・安定性が維持されるので、ゲノム情報を利用することで耐熱性酵素を容易に獲得することが可能となった。
筆者の研究室でも、75℃から80℃およびpH 2.0から3.0が最適生育条件である好気性・好酸性超好熱アーキアS. tokodaiiのゲノム情報から糖代謝関連酵素を見出し、その解析に取り組んできた。その際の詳細な酵素活性特性の解析から、本超好熱アーキア由来耐熱性糖代謝酵素では、酵素活性そのものが常温菌由来酵素に比べて、比較的低いことが明らかとなってきた。そこで、耐熱性糖代謝酵素の活性促進に取り組んだ結果についてまとめてみる。
2.超好熱アーキアS. tokodaii由来ST0452蛋白質の特徴
好気性・好酸性超好熱アーキアS. tokodaiiは、エネルギーを獲得する際にグルコースをリン酸化せずに代謝する変形Entner-Doudoroff経路を利用していることが知られている6)。この様なユニークな糖代謝経路を有することは、ゲノム情報中にもユニークな糖代謝に関連する酵素が存在していると推定されるので、本アーキアのゲノム情報を用いて糖代謝関連酵素だと推定される遺伝子を含む多数の遺伝子から、大腸菌のpET発現系を用いて蛋白質の発現を試みた。約半数の発現クローンから耐熱性・可溶性蛋白質が大腸菌内で生産されていることが確認された。その中からグルコース-1-リン酸とチミジン-3-リン酸 (TTP) を結合して、TDP-グルコースを合成する活性を有すると推定されたST0452蛋白質について、その活性の詳細な解析を行った。その結果、本ST0452蛋白質は高い熱安定性を有すると共に、グルコース-1-リン酸とTTPとを結合してTDPグルコースを合成できることが確認された7)。さらに興味深いことに、基質特異性の解析から、最も高い活性を示した基質はN-アセチルグルコサミン-1-リン酸とウリジン-3-リン酸 (UTP) であることが示された7)。
さらにバクテリアのN-アセチルグルコサミン-1-リン酸とUTPを結合する活性を有する酵素はグルコサミン-1-リン酸にアセチルCoAのアセチル基を転移させてN-アセチルグルコサミン-1-リン酸を合成する活性もあわせ有していることが知られている。そこで、本ST0452蛋白質についてもこの活性の確認を行ったところ、確かにこの活性も確認された。さらに驚くべきことに、これまでどの生物由来の酵素からも見いだされていなかったガラクトサミン-1-リン酸にアセチル基を転移させてN-アセチルガラクトサミン-1-リン酸を合成する活性も検出された8)。
グルコサミン-1-リン酸にアセチルCoAのアセチル基を転移させてN-アセチルグルコサミン-1-リン酸を合成する活性およびN-アセチルグルコサミン-1-リン酸とUTPを結合する活性を有するバクテリアのGlmU酵素と本ST0452蛋白質とのアミノ酸配列の一致度は20%と低く、これらの酵素活性を有することはアミノ酸の一次配列からは全く予想することは不可能であった。アミノ酸配列が異なっていても類似の酵素活性を示すというこの結果は、アミノ酸配列の類似性からその酵素機能を推定するだけでは充分ではないという事を示している。次世代シーケンサも登場して、以前に比して格段にゲノム配列の獲得は容易になったが、ゲノム情報は単なる推定で、実験的な機能の確認がさらに意味を増しているとも言える。
3.ST0452蛋白質と常温性バクテリア由来酵素の酵素活性比較
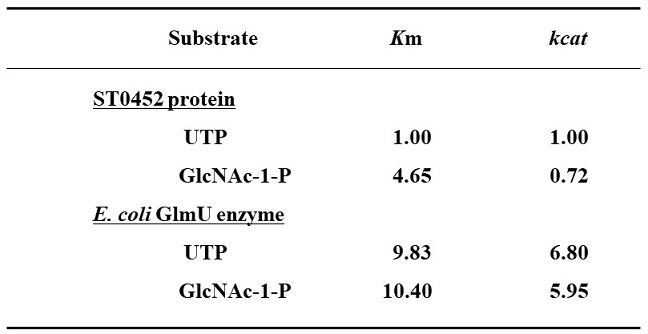
さて、本ST0452蛋白質が有するN-アセチルグルコサミン-1-リン酸とUTPを結合する酵素活性のKm値およびkcat値を求めた結果を表1に示す。常温性バクテリアの代表である大腸菌由来酵素の値も同じ表に示した。両者の比較から、アーキア由来ST0452蛋白質のKm値は、大腸菌由来酵素のKm値に比べて約10分の1であった。一方、本ST0452蛋白質のkcat値は、大腸菌由来酵素のkcat値に比べて数分の1であった9)。このことは、本ST0452蛋白質は、大腸菌由来酵素に比してかなり薄い基質でも利用できるが、その反面反応速度は数倍遅いということを示している。この反応速度が遅いという特徴は、本ST0452蛋白質の応用面を考慮するとデメリットである。一方、高い耐熱性と安定性は本ST0452蛋白質の大きなメリットである。そこで、本ST0452蛋白質が有する耐熱性を維持したままで反応速度を促進した酵素が作製できると、応用的にもたいへん有効である。そこで、本ST0452蛋白質が有する耐熱性と安定性を低下させることなく、N-アセチルグルコサミン-1-リン酸とUTPを結合させる活性の反応速度を促進した変異体の構築に取り組んだ。
![]()
![]()
4.ST0452蛋白質活性中心へのアミノ酸置換の導入
まず耐熱性・安定性を低下させない変異の導入手法として、活性中心に位置するアミノ酸残基への変異の導入を計画した。活性中心は酵素が基質を取り込む場所であり比較的酵素表面から内側に入りこんだポケットを形成している領域である。よって、この活性中心領域に存在するアミノ酸に変異を導入した場合でも蛋白質全体の構造には大きな影響を与えないはずである。言い換えると、それらのアミノ酸残基を他のアミノ酸に置換しても耐熱性・安定性への影響は少ないであろうと予想した。さらに活性中心のアミノ酸を変化させると本酵素が有する活性特性に何らかの影響があると予想されるので、活性中心として予測されるアミノ酸残基に変異を導入した。
図1に示すように、真正細菌由来類縁酵素の解析から推定された活性中心の重要なアミノ酸9個を他のアミノ酸に置換する変異を導入した。これらの活性に重要だと推定されているアミノ酸の多くは、塩基性或いは酸性のアミノ酸である。そこで、最も性質の異なるアミノ酸であるアラニンおよびロイシンまたはフェニルアラニンに置換した変異体をコードした発現クローン11個を作製した9)。


5.変異体ST0452蛋白質の耐熱性の確認
作製した変異体ST0452蛋白質発現用クローンを、外来蛋白質発現用大腸菌 (E. coli BL21 CodonPlus RIL) に導入したところ、全ての発現クローンから可溶性蛋白質の生産が確認された。この蛋白質を80℃で20分間加熱処理したところ、図2に示すように元のST0452蛋白質と同様に全ての変異体蛋白質が可溶性の状態を維持していた。この結果は、全ての変異体蛋白質が、耐熱性に関しては元のST0452蛋白質の耐熱性とほとんど変化していないことを示している。今回の変異体作製に際して、重要な課題とした「耐熱性と安定性を下げない」変異体を作製することができた9)。そこで、これら全ての変異体蛋白質について、N-アセチルグルコサミン-1-リン酸とUTPを結合する酵素活性の解析を行った。

6.変異体蛋白質の酵素活性の解析
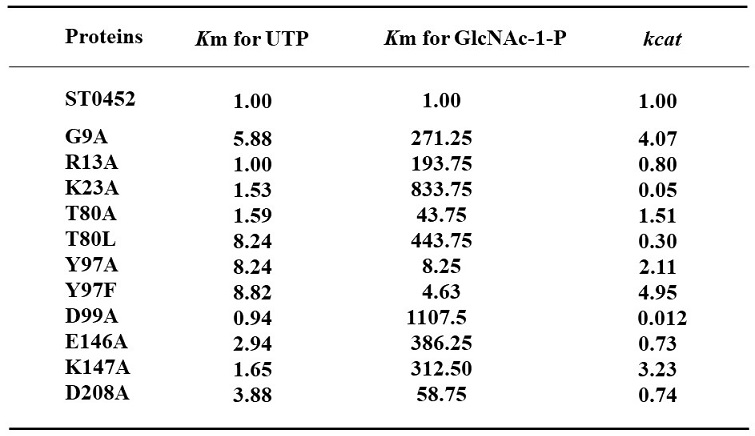
各変異体ST0452蛋白質の活性特性を調べたところ、表2に示したように全ての変異体のKm値が元のST0452蛋白質の値に比べて数倍から数十倍上昇していた。このことは、ST0452蛋白質の活性中心に位置するアミノ酸残基に変異を導入すると、元のST0452蛋白質に比して高い濃度の基質が必要だということを示している。さらにいくつかの変異体では、元のST0452酵素に比してkcat値が小さくなったことから、これらの変異ではアミノ酸置換の導入は酵素活性を下げてしまった9)。
![]()
![]()
しかしながら、表2に示すようにG9A、T80A、Y97A、Y97F、K147A変異体では、kcat値の上昇が見られた。このことは、これらの変異体では反応速度を元のST0452蛋白質よりも促進させることができたということを示している。しかしながら、これらのどの変異体もKm値については元のST0452蛋白質の値に比して大きくなっているので、見かけの反応は促進されているとは言い難い。しかし、実用的には実質的な反応速度の促進効果が出せれば充分だと思われる。そこで、上昇したKm値を補うために、濃い濃度の基質が供給されれば、元のST0452蛋白質よりも効率的に反応が進むことが予想されるので基質濃度を高くした条件での反応について検討した。
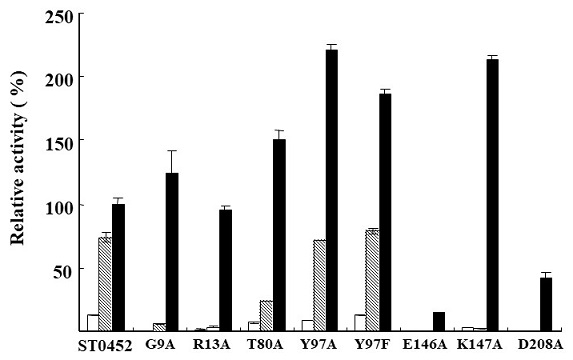
そこで、今回kcat値が促進されたものを含め全ての変異体蛋白質について、本ST0452蛋白質での活性測定に用いていた基質濃度よりも高い基質濃度での反応を解析した。最初に、UTP基質濃度が高い条件下での反応の進行を解析した。図3の網掛け棒で示すように、この条件では元のST0452蛋白質と同等程度の活性を示すものが2個見出されてきた。次に、N-アセチルグルコサミン-1-リン酸基質濃度も上げて、両方の基質濃度が高いという反応条件下での活性を解析した。その結果、図3の黒棒に示すように元のST0452蛋白質の活性を上回る活性を示すものがG9A、T80A、Y97A、Y97F、K147Aの5個見出されてきた9)。これらは全てkcat値が元のST0452蛋白質より上昇していたものである。このことは、これらの変異体ST0452蛋白質は、高濃度の基質を供給することで元のST0452蛋白質よりも格段に効率良く反応を進行させることができるということを示している。また同じS. tokodaii由来の他の酵素でも同様に、活性中心に位置する反応の進行に重要だと推定されるアミノ酸残基をアラニンに置換した際にもkcat値の上昇がみられた。これらの結果は、超好熱アーキアS. tokodaiiの有する酵素では、活性中心の重要なアミノ酸をアラニンに置換するとKm値は上がってしまうが、kcat値を上昇させることができるという一般的な性質を有しているとも推定される。このように、耐熱性を維持したままで酵素活性のkcat値を促進させることが出来た酵素の例は非常に珍しいので、今後の本手法の他酵素への応用等にも期待が大きい。

ではなぜ、この活性中心に位置する活性に重要なアミノ酸残基をアラニンに置換した時にkcat値を促進出来るのであろうか。この問いに対する正確な答えを得るには、本変異体ST0452蛋白質と基質・反応生成物との共結晶構造の解明が必要だと思われる。ここでは、一つの仮説を記しておきたい。今回ST0452蛋白質の活性中心に位置しておりアミノ酸置換を導入したアミノ酸の多くは、酸性か塩基性のアミノ酸残基である。これらの荷電しているアミノ酸残基を今回の変異体作成では、電荷も極性も無いアミノ酸残基であるアラニンに置換した。酵素反応の活性中心に荷電しているアミノ酸残基が存在するのは、基質や補酵素、金属イオンと酵素自身がしっかりした複合体を形成するためである。しかし、超好熱アーキアの生活環境である高温環境では、基質や反応生成物の運動エネルギーが高く、酵素がより強固に基質を捉えないと反応を進めることが出来ないのではないか。その結果、反応生成物も比較的強固に酵素に捕捉されて、反応生成物の酵素からの遊離が遅いのではないかと推測される。その結果、大腸菌由来酵素と比べてkcat値が数倍小さいものとなっているのではないだろうか。その活性中心に位置するアミノ酸残基を、基質や反応生成物との相互作用が弱いアラニン等に置換したことで、反応生成物の遊離が容易になり、その結果反応全体が早く進行するようになったのではないかと推測される。この仮説を確認するためには、これら変異体ST0452蛋白質の反応生成物との共結晶構造の解析等を進めることが必要であろう。一方、大腸菌等の常温菌由来酵素も活性中心には本ST0452蛋白質と同様のアミノ酸残基を有している (図1)。しかしながら、本ST0452蛋白質と異なり、これらのアミノ酸残基を他のアミノ酸残基特にアラニンに置換すると、活性が大きく低下してしまう。このことは、超好熱アーキア由来の酵素と大腸菌等の常温菌由来酵素では、活性中心以外の領域に存在するアミノ酸残基の違いが、大きく影響していることを示唆する。活性中心周辺のアミノ酸残基の役割も解明することが困難な課題だと思われるが、一般的な酵素の活性促進や耐熱性酵素の創製等にも結びつく重要な課題だと考えられるので、今後この課題の解明にも取り組んでいきたい。
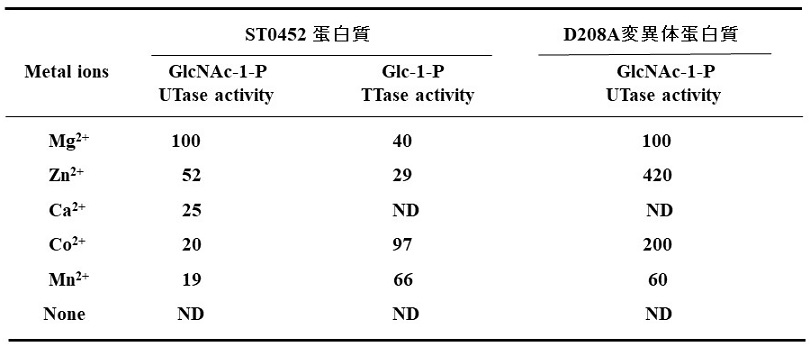
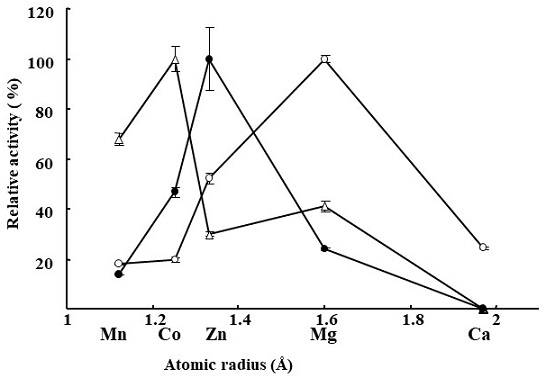
さて、基質濃度を濃くした場合でも、活性の促進が見られなかったD208A変異体であるが、この208番目のアミノ酸残基は金属イオンとの相互作用に重要なアミノ酸残基だと推定されている。そこで、このD208A変異体蛋白質の酵素活性を種々の金属イオンを添加した条件下で解析し、その金属イオン種に対する酵素活性の違いを元のST0452蛋白質のものと比較した。その結果を表3に示した。元のST0452蛋白質のグルコース-1-リン酸とTTPを結合する活性の場合にはCo2+イオンを反応液に添加した時に最も高い活性を示すが、N-アセチルグルコサミン-1-リン酸とUTPを結合する活性の場合にはMg2+を添加した時に最も高い活性を示す。一方、Zn2+を添加した時にD208A変異体蛋白質は、元のST0452蛋白質がMg2+添加の際に示す活性の4.2倍の活性を示すことが確認された9)。このことは、本ST0452蛋白質の208番目のアミノ酸は金属認識に重要な働きをしており、さらに変異を導入することで、元のST0452蛋白質と異なる金属イオン特異性を有するように変化することを示している。ST0452蛋白質が有する2つの酵素反応グルコース-1-リン酸とTTPを結合する反応、N-アセチルグルコサミン-1-リン酸とUTPとを結合させる反応、D208A変異体蛋白質のN-アセチルグルコサミン-1-リン酸とUTPとを結合する反応について、金属イオンの種類と相対活性の相関を図4に示した。ここからわかるように、元のST0452蛋白質およびD208A変異体蛋白質の各酵素反応に最も適した金属イオンが存在し、各蛋白質の反応・活性の促進度合は、各金属イオンと最適金属イオンとのイオン半径の違いに従って活性促進度合いが低下している。これらの結果は、本ST0452蛋白質では基質と金属イオンの両方を最適な条件にすることで目的とする反応を効率的に進行させることができることを示している。
![]()
![]()

7.おわりに
超好熱アーキアが有している多数の酵素は、耐熱性だけでなく様々な産業応用に有利な利点を有している。一方、反応の進行速度は常温菌由来の酵素に比して比較的低いという欠点を有している。しかし、本稿で示したような変異を導入することで、超好熱アーキア由来の糖代謝酵素が持つ反応速度を促進出来ることが示された。基質に対するKm値も上昇するがこのことは逆に、基質化合物に低濃度で含まれる不純物等は基質として利用されにくくなるので、産業応用的には有利に働く点だとも言える。今後、立体構造解析による変異導入による活性促進のメカニズムの解明や、超好熱アーキア由来の他の酵素における同様の変異導入による活性促進の可能性等について検証を行なっていきたい。
謝辞
本稿中のデータについては、筆者の研究室で共に研究に励んでくれた多くの大学院生・学術研究員の皆さんの努力の賜物である。この場をお借りして感謝を表したい。
文献
1) Brock, T. D., Freeze, H.: J. Bacteriology, 98, 289 (1969).
2) Chien, A., Edgar, D. B., Trela, J. M.: J. Bacteriology, 127, 1550 (1976).
3) Kawarabayasi, Y., Sawada, M., Horikawa, H., Haikawa, Y., Hino, Y., Yamamoto, S., Sekine, M., Baba, S., Kosugi, H., Hosoyama, A., Nagai, Y., Sakai, M., Ogura, K., Otsuka, R., Nakazawa, H., Takamiya, M., Ohfuku, Y., Funahashi, T., Tanaka, T., Kudoh, Y., Yamazaki, J., Kushida, N., Oguchi, A., Aoki, K., Kikuchi, H.: DNA Research, 5, 55 and 147 (1998).
4) Kawarabayasi, Y., Hino, Y., Horikawa, H., Yamazaki, S., Haikawa, Y., Jin-no, K., Takahashi, M., Sekine, M., Baba, S., Ankai, A., Kosugi, H., Hosoyama, A., Fukui, S., Nagai, Y., Nishijima, K., Nakazawa, H., Takamiya, M., Masuda, S., Funahashi, T., Tanaka, T., Kudoh, Y., Yamazaki, J., Kushida, N., Oguchi, A., Kikuchi, H.: DNA Research, 6, 83 and 145 (1999).
5) Kawarabayasi, Y., Hino, Y., Horikawa, H., Jinno, K., Takahashi, M., Sekine, M., Baba, S., Ankai, A., Kosugi, H., Hosoyama, A., Fukui, S., Nagai, Y., Nishijima, K., Otsuka, R., Nakazawa, H., Takamiya, M., Kato, Y., Yoshizawa, T., Tanaka, T., Kudoh, Y., Yamazaki, J., Kushida, N., Oguchi, A., Aoki, K., Masuda, S., Yanagii, M., Nishimura, M., Yamagishi, A., Oshima, T., Kikuchi, H.: DNA Research, 8, 123 (2001).
6) De Rosa, M., Gambacorta, A., Nicolaus, B., Giardina, P., Poerio, E., Buonocore, V.: Biochemistry J., 224, 407 (1984).
7) Zhang, Z., Tsujimura, M., Akutsu, J., Sasaki, M., Tajima, H., Kawarabayasi, Y.: J. Boil. Chem., 280, 9698 (2005).
8) Zhang, Z., Akutsu, J., Kawarabayasi, Y.: J. Bacteriology, 192, 3287 (2010).
9) Zhang, Z., Akutsu, J., Tsujimura, M., Kawarabayasi, Y.: J. Biochemistry, 141, 553 (2007).
10) Gehring, A. M., Lees, W. J., Mindiola, D. J., Walsh, C. T., Brown, E. D.: Biochemistry, 35, 579 (1996).