【トピックス】
回虫成虫フマル酸還元酵素の構造・機能相関と特異的阻害剤の分子設計
稲岡ダニエル健、大森惇子、北 潔、原田繁春
東大院・医、東大院・医、東大院・医、京工繊大院・工
1.はじめに
寄生虫が引き起こす感染症は、人類や家畜の健康を著しく損ない多くの命を奪ってきた。しかし、先進諸国では医学や公衆衛生の発達に伴って感染症によるヒトへの被害は激減し、むしろガンや生活習慣病などに大きな関心が寄せられている。その一方で、開発途上国の人々は今なお寄生虫感染症による深刻な被害を受けている。例えばトリパノソーマが引き起こす中南米のシャーガス病やアフリカ睡眠病は、「顧みられない熱帯病 (neglected tropical disease, NTD)」と呼ばれ、先進諸国は積極的に治療薬の開発を行ってはこなかった。また、睡眠病はヒトだけでなく、家畜への感染によって地域の経済発展を妨げており、「安全で副作用のない抗寄生虫薬の開発」は開発途上国の発展に不可欠であり、まさにわが国が科学先進国として取り組むべき課題である。
真核生物である寄生虫と哺乳類には、代謝経路やそこで働いているタンパク質に多くの共通点がある。そのためマラリアをはじめワクチン開発は困難をきわめており、また薬剤も耐性株の出現や強い副作用など問題が多く、いわゆる特効薬は非常に少ない。一方、寄生虫はライフサイクルにおける環境変化に適応するため、固有の代謝経路も発達させており、宿主哺乳類とは大きく異なったこれらの代謝系は格好の薬剤標的と考えられる。その中でも、我々は寄生虫の嫌気的環境下におけるエネルギー代謝経路について「分子寄生虫学」の観点から研究を進め1-5)、その成果を抗寄生虫薬の開発に向けてきた。なかでも、回虫成虫複合体II (回虫成虫キノール−フマル酸還元酵素:QFR) に対するフルトラニル6,7) やアフリカトリパノソーマのシアン耐性酸化酵素の特異的阻害剤アスコフラノン8-10) など、特異的かつ高い阻害活性を示す化合物を見出した。また、阻害剤とタンパク質の複合体構造も明らかにし、より優れた阻害剤の分子設計 (Structure based drug design、SBDD) を行い、抗寄生虫薬として有望な化合物を明らかにしつつある。本稿では、代表的寄生蠕虫であり、また虫体サイズが大きく研究対象に適していることから内外で精力的に研究が進められている回虫を取り上げ、回虫成虫QFRの立体構造と機能相関、阻害剤の分子設計について述べる。
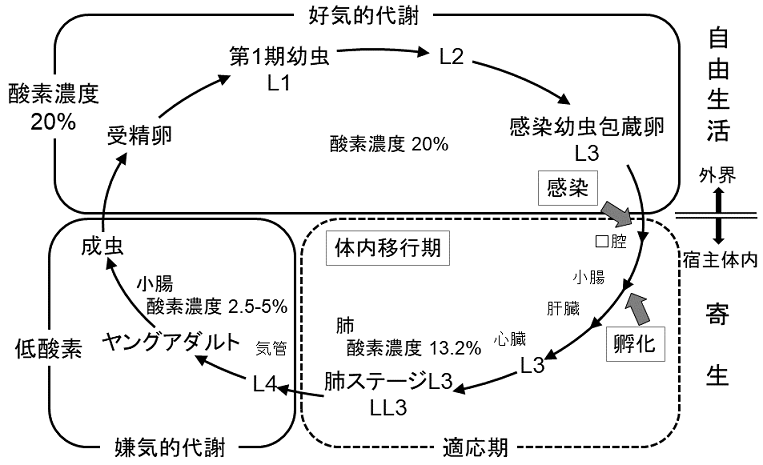
2.回虫のライフサイクルと複合体II
ライフサイクルの中で好気的環境と嫌気的環境で生息するステージをもつ寄生虫は、酸素濃度の変化に応じてエネルギー代謝の方法を変えている2-5,11,12)。回虫の場合、糞便とともに受精卵が酸素の豊富な外界 (酸素濃度20 %) に排出されると卵内で発達して幼虫 (L3ステージ) になる。この感染幼虫包蔵卵が口から宿主哺乳類の体内に入ると小腸で孵化し、この幼虫は肝臓から心臓を経て肺に (酸素濃度 13 %、LL3ステージ) 到達し、咽頭から食道と進み最後は小腸 (酸素濃度 2.5−5.0 %) に戻って成虫になる (図1)。L3ステージの幼虫は好気的エネルギー代謝でATPを合成するが13)、酸素濃度の低い環境で生息する成虫は、酸素を使わない糖分解経路 (ホスホエノールピルビン酸−コハク酸経路) でATPを合成している4,14)。この経路の最終段階で働いているNADH−フマル酸還元系は「フマル酸呼吸」とも呼ばれ、糖の分解過程で生じたNADHを複合体I (NADH脱水素酵素) が再酸化することでNAD+の再生を行っている。この再酸化と共役して低電位電子伝達体のロドキノン (RQ) はロドキノール (RQH2) に還元され、次に、複合体IIがフマル酸還元酵素 (QFR) としてRQH2を酸化し、最終電子受容体であるフマル酸を還元する。一方、好気的環境下で生息している幼虫 (L3ステージ) の場合、複合体IIはTCA回路のコハク酸-ユビキノン還元酵素 (SQR) としてコハク酸の酸化に共役したユビキノン (UQ) 還元反応を触媒する。

これまでに、大腸菌15)、ブタ16)、ニワトリ17)のSQR、大腸菌18)、Wolinella succinogenes19)と回虫7)のQFRの立体構造が決定されている。これらは回虫成虫QFRや回虫幼虫SQRも含め、2つの親水性サブユニット (Fp、Ip) と膜を貫通している2つの疎水性サブユニット (CybL, CybS) から構成されている。お互いアミノ酸配 列の類似した類縁の膜タンパク質であるが、W. succinogenes QFRには膜貫通サブユニットが1つしかない。
回虫成虫QFRと幼虫SQRのサブユニット構成は、成虫ではFpA Ip CybL CybSA、幼虫の場合はFpL Ip CybL CybSLと表すことができるように、IpとCybLサブユニットは共通であるが、FpとCybSサブユニットは異なっている。さらに好気的環境から嫌気的環境に移る途中のLL3ステージ (図1) ではQFRとSQRの混合型 (FpL Ip CybSA CybL) 複合体IIが発現している。このように、ライフサイクルにおける酸素濃度の変化に対応するため、回虫複合体IIはサブユニットを入れ替えている。
これまでに我々は、ブタ回虫から回虫成虫QFRを抽出・精製・結晶化し、SPring-8やPFでX線回折強度データを測定し立体構造を決定した7)。また、回虫成虫QFRを特異的に阻害するフルトラニルとの複合体構造も決定した20)。そこで本稿では、主にQFRの構造と機能の相関、およびフルトラニルをベースにした抗線虫薬のSBDDについて最近の成果を紹介するとともに、新奇なサブユニット構造を持つトリパノソーマのSQRについてふれたい。
3.回虫成虫QFRの構造・機能相関
回虫成虫QFRの全体構造を図2Aに示す。CybLとCybSサブユニットがミトコンドリア内膜を貫通するアンカーサブユニットである。補欠分子族としてFpサブユニットにはFAD、Ipサブユニットには3つの鉄-硫黄クラスター ([2Fe-2S]、[4Fe-4S]、[3Fe-4S]) 、CybLとCybSサブユニットの接触面にはヘムbが結合している。ヘムbを除いて、補欠分子族に近接しているアミノ酸残基は、他生物種でも良く保存されている。また、Ip、CybL、CybSサブユニットの境界にはRQが、FADのイソアロキサジン環の近くにはフマル酸や類似化合物であるマロン酸の結合部位が存在する (図2B)。この構造から、RQ結合部位でRQH2の酸化反応 (RQH2→RQ+2e−+2H+) が起こり、それに共役したフマル酸 (HOOC−CH=CH−COOH) のコハク酸 (HOOC−CH2−CH2−COOH) への還元がFADH2によって行われていることが分かる。結晶構造中でRQとフマル酸の間を結んでいる補欠分子族 (RQ−[3Fe-4S]−[4Fe-4S]−[2Fe-2S]−FAD−フマル酸) は、隣接する補欠分子族同士が14 Å以下の距離 (図2B) で配置しているので、量子力学的トンネル効果21)によって低電位キノンであるRQH2 (酸化還元電位:Em'=−63 mV) から補欠分子族を経由してフマル酸 (Em'=+30 mV) へ電子が伝達される。この電子伝達経路は他生物種のQFRやSQRの立体構造でも共通に見られるので、電子伝達の方向 (QFRの場合はRQH2からフマル酸へ、SQRはコハク酸からUQへ) は基質の酸化還元電位によって決まると考えられる。実際、in vitroで回虫成虫QFRはSQRとしても機能できる。
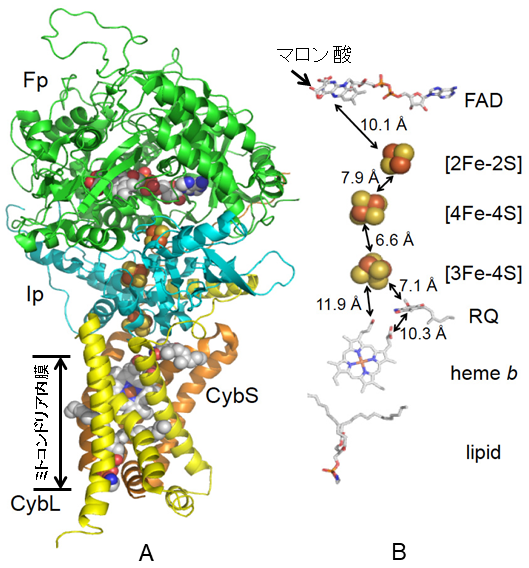

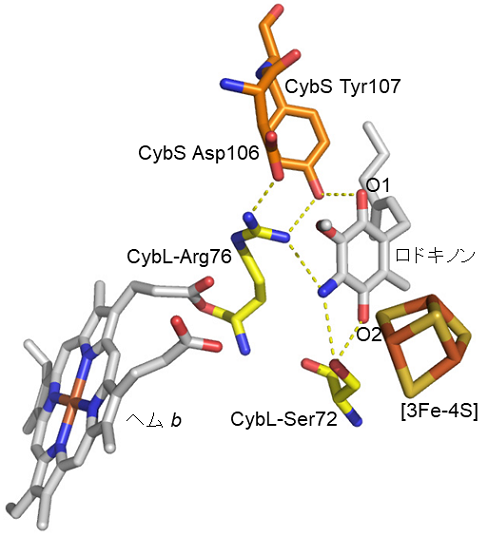
それでは、回虫成虫QFRでRQH2の酸化はどの様な機構で進むのだろうか。RQ結合部位の構造を見ると、RQに最も近い補欠分子族である [3Fe-4S] とRQのO1やO2は、それぞれ9.1 Åと7.1 Åの距離にある (図2)。一方、ヘムbもRQや [3Fe-4S] から、それぞれ10.3 Åと11.9 Åの距離にある。従って、ヘムbの酸化還元電位 (Em'=−34mV) がRQH2よりも高いことを考えると、RQH2の酸化によって生じた電子には直接 [3Fe-4S] に渡されてFADに伝達される経路とヘムb を経由する経路 (RQH2→ヘムb→ [3Fe-4S]→[3Fe-4S]→[2Fe-2S]→FAD→フマル酸) が考えられる。しかし、大腸菌QFRのようにヘムbを持たない複合体IIもあること、ヘムb近傍に存在するアミノ酸残基はほとんどが保存されていないことから、ヘムbの電子伝達経路への関わりは重要ではないと考えられる。さらに、RQH2の酸化によって生じる2個のH+が辿る経路についても明らかになった。CybLサブユニットのSer72とArg76、およびCybSサブユニットのAsp106とTyr107はブタ、ニワトリ、大腸菌SQRにも保存されているアミノ酸残基である。RQはこれらと2つの水素結合ネットワーク (RQ O1…Tyr107…Arg76…Asp106、RQ O2…Ser72…RQ-N…ArgC76…Asp106) を経由して (図3) 外部の水分子につながっており、このネットワークよってH+が排出されると考えられる。

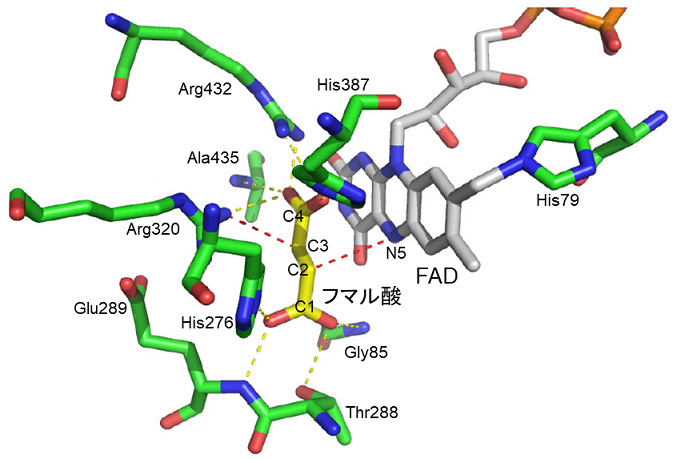
それでは、フマル酸はどの様な機構でコハク酸に還元されるのだろうか。この機構はフマル酸との複合体構造 (図4) から明らかにできた。フマル酸は分子内に共役した3つの二重結合があるので平面構造が最も安定である。しかし、回虫成虫QFRに結合しているフマル酸分子では、C2、C3とC4カルボキシル基は同一平面上にあるのに対し、C1カルボキシル基はその平面から外れていた (二面角C3−C2−C1−O1Aは83.7°)。一方、この不安定な構造はフマル酸とFpサブユニット間の水素結合で安定化されていた (図4)。C1カルボキシル基が平面から外れると、π電子はフマル酸分子全体に均一に分布できなくなり、C2とC3炭素上に正と負の部分電荷 (C2δ+、C3δ−) が生じる。これこそがフマル酸還元の鍵である。すなわち、FAD N5とC2δ+は3.5 Åしか離れていないので、RQH2の酸化と共役して還元されたFAD (FADH2) のN5−Hからヒドリド (H−) がC2δ+へ移動し、C3δ−はArg320からH+を受け取ってフマル酸がコハク酸に還元されると考えられる。この機構はフラボシトクロムc22)やトリパノソーマクルージーのジヒドロオロト酸脱水素酵素23)にも見られる。

4.回虫成虫QFRに特異的な阻害剤フルトラニル
フルトラニルは複合体IIを標的とする電子伝達系阻害作用によって担子菌類に特異的な抗菌活性を持ち、実際に農薬として使われている。また、哺乳類SQRに比べて回虫成虫QFRを790倍も強く阻害するので (回虫成虫QFRに対するIC50=58 nM、ブタSQRに対する阻害活性IC50=46 µM)、抗線虫薬を開発するためのリード化合物として大いに期待できる6)。そこで、特異性の要因を明らかにし、より優れた阻害剤を見出すために、フルトラニルと回虫成虫QFRおよびブタSQRとの複合体構造を解析した。両酵素を構成する4つのサブユニットは、お互いアミノ酸配列に高いホモロジーがあり、立体構造も極めて良く似ている。フルトラニルはキノン結合部位に結合し、その周辺5 Å以内にあるアミノ酸残基のうち73 %が共通であった (図5)。それにもかかわらず、QFRに対してナノモルオーダーの高い阻害活性を示すのは何故だろうか。
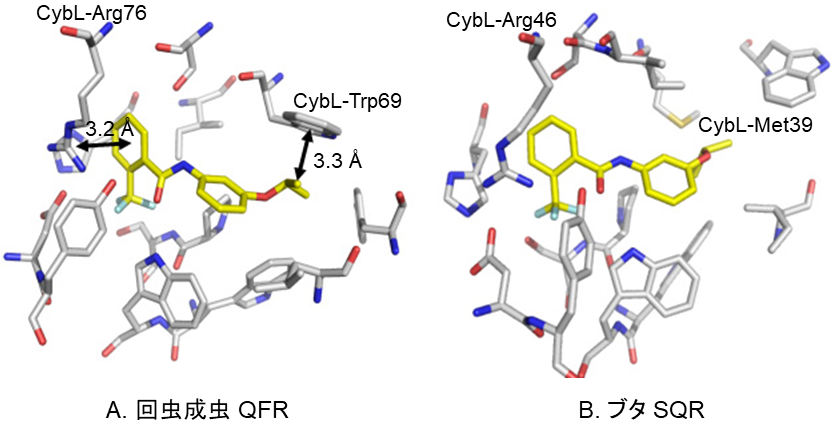

結晶構造から3つの違いを見出すことができた。第一に、フルトラニルのイソプロポキシ基 (−O−CH(CH3)2 ) は回虫成虫QFRのTrp69 (CybLサブユニット) のインドール環と3.3 Åの距離にあり、両者の間にC−H…π相互作用が働いていた (図5A)。一方、ブタSQRではこのトリプトファン残基がMet39であるため、π電子が関わる静電的相互作用がない (図5B)。第二点はフルトラニルのトリフルオロメチルベンゼン環とアルギニン残基との相互作用である。電子吸引性置換基であるトリフルオロ基はベンゼン環からπ電子を吸引し、このベンゼン環を電子欠乏状態にしている。回虫成虫QFR CybLサブユニットのArg76は、ロドキノールの酸化で生じるプロトンをQFR外部の水分子にリレーする水素結合ネットワークの一員 (図3) であるとともに膜貫通サブユニットの分子内部にあるので、側鎖のグアニジノ基は電気的に中性であると考えられる。回虫成虫QFRではπ電子欠乏状態にあるベンゼン環と電子豊富な窒素原子をもつグアニジノ基が3.2 Åの距離で重なって静電的相互作用をしているが (図5A)、ブタSQRはそのような配置になっていない (図5B)。実際、トリフルオロメチルを電子供与性置換基 (メチル基等) に変えると、回虫成虫QFRに対する阻害活性は大きく低下 (IC50=515 nM) するが、ブタSQR (IC50=90 µM) では変化は小さかった。第三は、フルトラニル結合部位のポケットの大きさが回虫成虫QFRの方が大きいことである。これは、ポケットを構成しているアミノ酸残基のうち、回虫成虫QFRのグリシンとプロリン残基がブタSQRでは嵩高いイソロイシンとトリプトファン残基であるためである。これらの知見に基づいてさらに優れた阻害剤を見出すため、様々なフルトラニル誘導体設計・合成し、阻害活性の測定と複合体構造の解析を行った。その結果、回虫成虫QFR (IC50=5.47 nM) をブタSQR (IC50=111 µM) よりも20,000倍強く阻害する化合物を見出すことができた (大森他。投稿準備中)。現在、この成果をもとに家畜動物の線虫による感染症治療薬への実用化研究を行っている。
5.ユニークなサブユニット構成を持つTrypanosomacruziの複合体II
代表的な「顧みられない熱帯病NTDs」のひとつであり、中南米の風土病として知られるシャーガス病の原因となっているアメリカ型トリパノソーマT. cruziの複合体IIは、6つの親水性サブユニット (SDHA、SDHBN、SDHBC、SDHE〜SDHG) と6つの膜貫通サブユニット (SDHC、SDHD、SDHH〜SDHK)、合計12個のサブユニットでできている (図6)24)。この内5つのサブユニット (SDHA, SDHBN, SDHBC, SDHC, SDHD) に複合体IIの機能に重要なアミノ酸残基が存在している。SDHAはFp、SDHCとSDHDはそれぞれCybLとCybSに対応しているが、IpサブユニットはSDHBNとSHBCの2本のポリペプチド鎖に分かれている。このようにT. cruzi複合体IIは他生物種の複合体IIとサブユニット構成が大きく異なっている。さらに、様々な阻害剤に対する感受性にも違いがある。例えば、アトペニンA5は哺乳類 (ウシ) SQR (IC50=4 nM) や回虫成虫QFR (IC50=12 nM) を極めて強く阻害するのに対し、T. cruzi複合体IIに対するIC50は6.4 µMと非常に高い。また、マロン酸 (IC50=40 µM) による阻害も他生物種の複合体IIに比べると小さい。このような違い、特に哺乳類複合体IIとの違いは、T. cruzi複合体IIに特異性の高い阻害剤を創成できる可能性が高く、シャーガス病治療薬のターゲットタンパク質として大いに期待できる。
6.おわりに
これまでに述べてきたように、寄生虫はライフサイクル中の好気的環境と嫌気的環境で、異なるエネルギー代謝経路が働くことで環境に適応している。しかし、ステージ特異的にそれらが発現する分子機構についてはほとんど理解が進んでいない。というのも、寄生虫を分子レベルで研究する、謂わば「分子寄生虫学」はやっと始まったばかりなのである。この様な状況の中で複合体IIが外界の酸素濃度センサーの役割を担っているという報告25)は非常に興味深い。実際、酸素濃度が異なる環境で生息する回虫の幼虫と成虫はステージ特異的に複合体II (幼虫SQR、成虫QFR) を発現し26)、その制御は哺乳類の細胞に存在しているhypoxia-inducible factor-1 (HIF-1) と類縁のタンパク質が行っている可能性が高い27)。一方、ヒトの複合体IIにも2種類のFpサブユニット (type Iとtype II) が見出されており、これらはわずか2個のアミノ酸残基が違うだけである28,29)。興味深いことに、type II Fpを持つ複合体IIは虚血状態にある細胞で発現している30)。すなわち、嫌気的環境下で生息している回虫成虫と同様、ヒト細胞でも低酸素状態に直面すると、ミトコンドリア中でフマル酸呼吸によるエネルギー代謝を行う鍵酵素として、type IIは高いQFR活性を持っている可能性がある。このような、寄生虫だけでなくヒトにも見られるミトコンドリア複合体IIの多様性は生体エネルギーや分子進化を研究する上で格好の研究材料であると同時に創薬ターゲットとしても有望である。
謝辞
本研究は文部科学省ターゲットタンパク研究プログラムと農林水産省生研センターイノベーション創出事業、また一部をJSTとJICAによるSATREPSの支援を受けて行った。またフルトラニル誘導体の合成、供与をいただいた東京大学大学院薬学系研究科井上将行教授および日本農薬の皆様に謝意を表します。
文献
1) Kita, K., Hirawake, H., Takamiya, S.: Int. J. Parasitol., 27, 617 (1997).
2) Komuniecki, R., Komuniecki, P. R.: J.C. Boothroyd, R. Komuniecki (Eds.): Molecular Approaches to Parasitology, Wiley-Liss, Inc., New York, 109 (1995).
3) Tielens, A. G. M., Rotte, C., van Hellemond, J. J., Martin, W.: Trends. Biochem. Sci., 27, 56 (2002).
4) Kita, K., Takamiya, S.: Adv. Parasitol., 51, 95 (2002).
5) Kita, K.: Today, 8, 155 (1992).
6) Osanai, A., Harada, S., Sakamoto, K., Shimizu, H., Inaoka, D. K., Kita, K.: Acta Crystallogr. Sect. F Struct. Biol. Cryst. Commun., 65, 941 (2009).
7) Shimizu, H., Osanai, A., Sakamoto, K., Inaoka, D. K., Shiba, T., Harada, S., Kita, K.: J. Biochem., 151, 589 (2012).
8) Shiba, T., Kido, Y., Sakamoto, K., Inaoka, D. K., Tsuge, C., Tatsumi, R., Takahashi, G., Balogun, E. O., Nara, T., Aoki, T., Honma, T., Tanaka, A., Inoue, M., Matsuoka, S., Saimoto, H., Moore, A. L., Harada, S., Kita, K.: Proc. Natl. Acad. Sci. USA, 110, 4580 (2013).
9) Kido, Y., Sakamoto, K., Nakamura, K., Harada, M., Suzuki, T., Yabu, Y., Saimoto, H., Yamakura, F., Ohmori, D., Moore, A., Harada, S., Kita, K.: Biochim. Biophys. Acta, 1797, 443 (2010).
10) Kido, Y., Shiba, T., Inaoka, D. K., Sakamoto, K., Nara, T., Aoki, T., Honma, T., Tanaka, A., Inoue, M., Matsuoka, S., Moore, A., Harada, S., Kita, K.: Acta Crystallogr. Sect. F. Struct. Biol. Cryst. Commun., 66, 275 (2010).
11) Amino, H., Osanai, A., Miyadera, H., Shinjyo, N., Tomitsuka, E., Taka, H., Mineki, R., Murayama, K., Takamiya, S., Aoki, T., Miyoshi, H., akamoto, K., Kojima, S., Kita, K.: Mol. Biochem. Parasitol., 128, 175 (2003).
12) Amino, H., Wang, H., Hirawake, H., Saruta, F., Mizuchi, D., Mineki, R., Shindo, N., Murayama, K., Takamiya, S., Aoki, T., Kojima, S., Kita, K.: Mol. Biochem. Parasitol., 106, 63 (2000).
13) Kita, K., Hirawake, H., Miyadera, H., Amino, H., Takeo, S.: Biochim. Biophys. Acta, 1553, 123 (2002).
14) Omura, S., Miyadera, H., Ui, H., Shiomi, K., Yamaguchi, Y., Masuma, R., Nagamitsu, T., Takano, D., Sunazuka, T., Harder, A., Kolbl, H., Namikoshi, M., Miyoshi, H., Sakamoto, K., Kita, K.: Proc. Natl. Acad. Sci. USA, 98, 60 (2001).
15) Iverson, T. M., Luna-Chavez, C., Cecchini, G., Rees, D. C.: Science, 284, 1961 (1999).
16) Sun, F., Huo, X., Zhai, Y., Wang, A., Xu, J., Su, D., Bartlam, M., Rao, Z.: Cell, 121, 1043 (2005).
17) Huang, L., Sun, G., Cobessi, D., Wang, A. C., Shen, J. T., Tung, E. Y., Anderson, V. E., Berry, E. A.: J. Biol. Chem., 281, 5965 (2006).
18) Yankovskaya, V., Horsefield, R., Tornroth, S., Luna-Chavez, C., Miyoshi, H., Leger, C., Byrne, B., Cecchini, G., Iwata, S.: Science, 299, 700 (2003).
19) Lancaster, C. R. D., Kroger, A., Auer, M., Michel, H.: Nature, 402, 377 (1999).
20) Harada, S., Inaoka, D. K., Ohmori, J., Kita, K.: Biochim. Biophys. Acta, in press.
21) Page, C. C., Moser, C. C., Chen, X., Dutton, P. L.: Nature, 402, 47 (1999).
22) Taylor, P., Pealing, S. L., Reid, G. A., Chapman, S. K., Walkinshaw M. D.: Nat. Struct. Biol., 6, 1108 (1999).
23) Inaoka, D. K., Sakamoto, K., Shimizu, H., Shiba, T., Kurisu, G., Nara, T., Aoki, T., Kita, K., Harada, S.: Biochemistry, 47, 10881 (2008).
24) Morales, J., Mogi, T., Mineki, S., Takashima, E., Mineki, R., Hirawake, H., Sakamoto, K., Ōmura, S., Kita, K.: J. Biol. Chem., 284, 7255 (2009).
25) Baysal, B. E., Ferrell, R. E., Willett-Brozick, J. E., Lawrence, E. C., Myssiorek, D., Bosch, A., van der Mey, A., Taschner, P. E., Rubinstein, W. S., Myers, E. N., Richard, C. W., Cornelisse, C. J., Devilee, P., Devlin, B.: Science, 287, 848 (2000).
26) Saruta, F., Kuramochi, T., Nakamura, K., Takamiya, S., Yu, Y., Aoki, T., Sekimizu, K., Kojima, S., Kita, K.: J. Biol. Chem., 270, 928 (1995).
27) Goto, M., Amino, H., Nakajima, M., Tsuji, N., Sakamoto, K., Kita, K.: Gene, 516, 39 (2013).
28) Tomitsuka, E., Hirawake, H., Goto, Y., Taniwaki, M., Harada, S., Kita, K.: J. Biochem., 134, 191 (2003).
29) Tomitsuka, E., Goto, Y., Taniwaki, M., Kita, K.: Biochem. Biophys. Res. Commun., 311, 774 (2003).
30) Sakai, C., Tomitsuka, E., Esumi, H., Harada, S., Kita, K.: Biochim. Biophys. Acta, 1820, 643 (2012).