【トピックス】
バイオマス糖化における糖質結合モジュール (CBM) の特性と応用
苅田修一
三重大院・地域イノベーション学
1.はじめに
植物細胞壁に含まれる構造多糖類は、この地球上で最も多く生産される有機物質であり、これを再生可能なエネルギー源、あるいは、資源として利用することは、人類の長年の夢である。自然界では、これらの構造多糖類が微生物により分解されており、生物圏の炭素フローにおいて微生物は重要な役割を果たしている。これらの微生物が、どのように植物細胞壁を酵素分解しているかというメカニズムを解明することは、これらの利用を実現するために重要であり、実際に、これまでに膨大な研究が行われてきた。本トピックでは、植物細胞壁多糖類を分解する酵素がもつ特徴のひとつである、糖質結合モジュール (CBM) について紹介したい。ただし、現在CBMは、糖を認識して結合するタンパク質ドメインとして広く定義されており、動物における糖鎖認識に関するものも含まれている。本トピックでは、特に植物細胞壁多糖類に結合するものについて紹介する。
2.糖質結合モジュール (CBM)
セルロースを加水分解するセルラーゼが他の酵素と異なるのは、基質であるセルロースが水に不溶性の基質であることである。したがって、セルラーゼは、これを加水分解するために、その基質表面に結合しなければならない。Trichoderma reesei由来のセルラーゼ (セロビオヒドロラーゼCel7A) をパパインで加水分解することで、酵素を触媒作用のあるドメインと、基質結合ドメインに分けることができた。触媒ドメインは、基質結合ドメインを取り除くと、可溶性の基質に対する活性には変化がないものの、結晶性セルロースに対する酵素活性が失われることが示された1)。このように、セルラーゼの一部分であるセルロース結合ドメインは、そのアミノ酸配列から、いくつかのグループに分けられてきた2)。現在ではセルロース結合ドメインは、広く糖質を標的として結合するドメインとして、「糖質結合モジュール (Carbohydrate-binding module) (CBM) 」と命名され、CAZyデータベース (http://www.cazy.or/) 上に記載されている。目下のところCBMファミリーは、64まで定義されており、更新が続いている3)。
このデータベースでは、アミノ酸配列のみが考慮されファミリーが定義されているので、同じファミリーに属するCBMにおいても、結合基質が異なっている場合もある。便宜上、CBMのファミリーの場合、CBMを略号とし、そこにファミリーの番号を記述する。CBM1は、ファミリー1に属する糖質結合モジュールを示し、CBM30は、ファミリー30に属するものを示している。セルラーゼ (エンドグルカナーゼ、エキソグルカナーゼに属し、EC3.2.1.4、3.2.1.91、3.2.1.176の活性を持つ酵素) がもっているCBMファミリーは、1、2、3、4、5、6、8、10、11 、17、18、28、30、32、35、37、44、46、49、63と広範囲に属している。CBMのコードされている位置は、N末端側、C末端側、あるいは複数の触媒ドメインの間にある場合もあり、酵素によって異なる。また、CBMがタンデムに複数並んでいる場合もある。また、特定の糖質加水分解酵素ファミリー (GH) との組み合せで存在している場合もある。たとえば、GH10に属するキシラナーゼとCBM9との組み合せが知られている。
表1に示すように、CBM1をもつ酵素は多様である。しかし、いずれも植物細胞壁多糖類に関連する酵素であり、セルロースに結合するという特性が、これらの酵素を植物細胞壁に集めている。酵素本来の分解基質に結合しなくてもセルロースに結合することで、細胞壁多糖類の分解においては十分に効果を発揮すると考えられる4)。CBM1は、真核生物に特有で、一方、CBM4、10、11 、12、17などは、原核生物のみに限定されている。またCBM37は、属特異的で、これまでのところ、Rumiococcus属にしか見つかっていない。CBM43やCBM49は、植物特異的である。
表1 糖質結合モジュール (CBM1) を持っている酵素

3.CBMの結合様式とタンパク質構造
糖質への結合様式により、CBMをタイプにわけることが、Borastonらにより提案され、これが広く受け入れられている5)。タイプAのCBMは、フラットな結合面をもち、結合面には、トリプトファンやチロシンといった芳香環をもつアミノ酸が並んでいる。これらのアミノ酸の疎水的な相互作用により、セルロースの結晶表面に結合する。ファミリーとしては、1、2、3、5、10などが知られている。タイプBは、タンパク質表面にクレフトと呼ばれる基質を結合する溝がある。したがって、溝に入るような遊離した糖鎖が結合に不可欠である。クレフト内には、芳香族アミノ酸残基や極性をもつアミノ酸が配置されており、酵素のサブサイトのような構造をもっている。ほとんどのCBMは、このタイプBに属している。タイプCは、短いクレフトをもっているが、クレフトの端が埋まっているため、糖鎖の末端部分にしか結合ができないものである。CBM9がその代表例である。
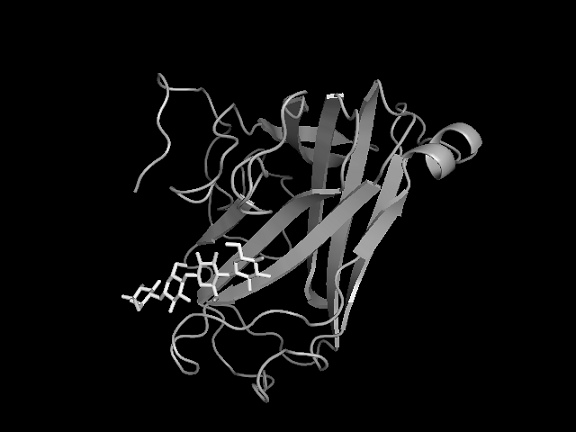
CBMのタンパク質としての構造のほとんどが、β-バレル構造で、その他に、レクチンのようなトレフォイル構造が知られている6,7)。図1にCBM28の構造を示した。β-バレル構造を有したタイプBのCBMである8)。
![]()
4.CBMの機能
酵素がCBMをもつことの意義としては、基質表面に結合することにより、酵素の有効基質濃度を上昇させる効果が考えられる。一般に、酵素は、基質濃度の上昇に伴い反応速度が増加する。セルラーゼの場合、基質であるセルロースは水に不溶性であるために、基質表面に結合することが、酵素の基質濃度を上げることになる。また、CBMをもつ酵素の多様性を考えると、必ずしも、触媒の基質特異性とCBMの標的が同じである必要はなく、CBMの標的に結合することで、本来の基質により近づけるという利点をもっていると考えられる。
5.CBMの結合特性の解析
5-1 CBMは、どこに結合するのか?
セルロース微結晶繊維のレベルで、タイプAに属するCBM1やCBM3は、セルロース結晶の110面と呼ばれる疎水面に結合する9)。これは、高速原子間力顕微鏡で観察されたT. reesei Cel7Aの動きとも一致しており、CBM1をもつ酵素は、結晶性セルロースの110面に結合し、基質分解を行っている10)。最近のコンピュータによる分子シミュレーションでも、同じ結果が得られている11)。
一方、Baeらは、C. stercorariumのCBM6とR. albus Cel5Bの融合タンパク質を用いて、アルファルファの細胞壁への結合を金コロイドで標識した抗体を用いて調べた。その結果、維管束細胞の細胞壁にはあまり結合せず、主に柔細胞の細胞壁に特異的に結合することを見いだした12)。また、CBMにHisタグをつけて発現させ、蛍光標識した抗ヒスタグ抗体を用いた検出や13)、CBMと蛍光タンパク質との融合タンパク質を発現することで、蛍光顕微鏡下において、CBMの結合する部位を特定することができる14)。
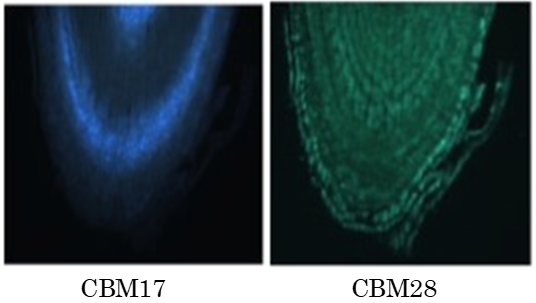
これらの結果から、CBMは、植物細胞壁に特異的に結合するが、細胞壁の場所や、細胞の種類により結合場所が異なっていることが明らかになり、なぜCBMが多様なのかを説明している15)。たとえば、Clostridium josuiに由来するCBM17とCBM28は、セロオリゴ糖に対する結合特性では、似た性質を示すものの、蛍光タンパク質との融合タンパク質を用いて、植物細胞壁の結合部位を観察すると明らかに異なる部位に結合していた。また、結合しやすい細胞壁が細胞の種類によって異なっていた (図2)16)。
![]()
実バイオマスは、そのままの状態では、酵素糖化することができないので、前処理が必要になる。その前処理を評価するために、糖化試験をしなければならないが、CBMの結合量が多い試料は、糖化率も高いという相関があり、糖化試験の前に分解しやすさを評価できる17)。また、セルロース結晶化度の変化をFT-IRやX線回折よりも感度よく、CBMの結合は、モニターすることができる18)。
5-2 CBMの結合のパラメータ
CBMの糖質への結合は、いくつかのパラメータによって評価できる。たとえば、結合定数や最大結合量である。基本的には、結合解析にはLangmuirの等温吸着曲線が使用されており、その逆対数プロットから、結合定数Kaと最大結合量を算出することができる19)。これは、異なる濃度の糖質結合モジュール溶液に、不溶性糖質を加え、結合しなかった上清の未結合部分のタンパク質濃度を定量することより、結合したタンパク質量を算出し、結合量を推定するものである。結合定数は、おおよそ、105〜106µmol-1 のオーダーにある。最近になり、Sugimotoらは、この解析にHillの式を導入した。従来のLangmuirの等温吸着式での回帰では、異なる結合定数をもつ結合部位が複数存在しないと回帰できなかったが、この問題を解決した20)。
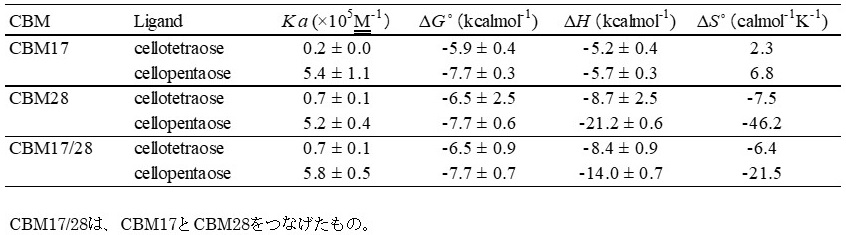
一方、タイプBのような可溶性の基質にも結合できるようなCBMの場合、等温滴定熱量計 (ITC) が評価に使用される。これは、CBM溶液に、結合基質であるオリゴ糖を滴下することで、結合の際に発生するわずかな発熱量を捉え測定する。これにより、タンパク質と基質との結合定数、結合比、発熱量が得られ、エントロピー変化を算出することができる。可溶性基質の場合の結合定数は、104〜106のオーダーにあり、基質の長さによって変化し、通常長いものほど、高い結合定数を示す。表2にCBM17とCBM28のITCから得られた結合パラメータを示した。CBM17と28を比較すると同じような結合定数をもっているが、結合における発熱量が異なっている。これは、CBM17では、エントロピーの寄与が大きいことを示している。またCBM17と28がつながったものでは、テトラオースは、2分子結合できるが、ペンタオースは、1分子しか結合ができない21)。
表2 等温滴定熱量計によるCBM17と28の結合特性
6.CBMの耐熱性の寄与
いくつかのドメインを外すと触媒の耐熱性が低下するという報告があり、このようなドメインを熱安定化ドメインと呼んでいた22)。このようなドメインのひとつがCBM22であり、後にCBMであることが判明した。一般に、CBMはβ-バレル構造をしており、触媒ドメインよりもコンパクトで、熱安定性が高い。C. stercorarium Xyn10Bの場合、示差走査熱量計で変性温度を測定すると、GH10の触媒ドメイン (CM10) の変性温度は74℃であった。一方CBM22の変性温度は82℃にあり、明らかに触媒よりも熱に安定な構造であった。このCBMを融合した酵素CBM22-CM10は、理由は明らかではないが、変性温度が6℃上昇し、80℃になり、温度に対してタンパク質全体の構造安定化を引き起こした。これは、触媒ドメインとCBMとの間の相互作用により、タンパク質全体が熱に対して安定化したと考えられる (図3)23)。
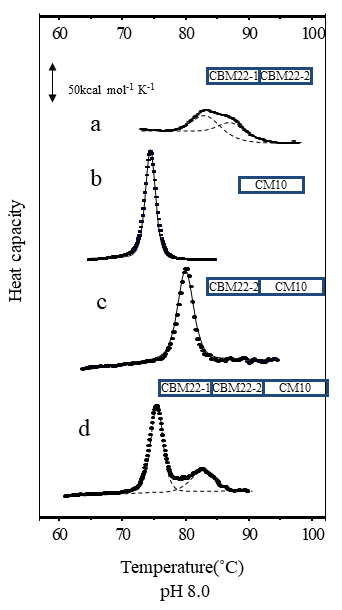
図3 CBMをもつ酵素の変性温度
7.CBMの基質崩壊作用
CBMは、繊維表面に結合するだけでなく、表面の構造を非加水分解的に崩壊させている。セルロースの場合、セロヘキサオースまでは、水に可溶であるが、それ以上の長鎖の場合は水に溶けない。しかし、酵素がセルロースを分解するには、少なくともクレフト内部まで、遊離したセルロース鎖が届く必要がある。そこで、糖質結合モジュールは、結晶面からのセルロース鎖の剥離に効果があると考えられてきた。実際に、糖質結合モジュールと綿繊維をインキュベーションすると、綿繊維表面にささくれのような崩壊が見られる24)。さらに、セルロース繊維の分子内水素結合、鎖間の水素結合が糖質結合モジュールの結合により弱められており25)、この作用をバイオマスの前処理に加えることにより糖化率を上げることができている26)。
8.CBMよる植物病害の応答
CBMは、微生物だけでなく植物にも分布しており、前述のCBM49の他、CBM43は花粉管タンパク質に見つかっている27)。植物病原菌には、セルロースに結合するCBM1をタンデムにつないだタンパク質を生産するものがあり、これらのCBM1は、植物に対して防御応答を引きこすことが知られている28)。そこで、CBM3の溶液をトマトに噴霧し、トマト青枯れ病菌を含んだ培土に移植したところ、CBM3溶液を噴霧したトマトは枯れず、病原菌に対して抵抗性を示した (図4)29)。このように、CBM溶液を噴霧し、CBMが植物に結合することにより、植物側に何らかのシグナルが伝わり、それにより植物が本来もっている病原微生物に対する抵抗性を誘導することがわかった30)。


9.おわりに
CBMは、単に、触媒を基質に近づけるという効果をもつだけでなく、様々な機能をもつことが分かってきた。CBMがつくことで、酵素の活性や基質特異性が影響を受けること、CBM自身が結晶性セルロースに対して崩壊活性をもつことも明らかになってきた。また、データベースには多くの構造の異なるCBMが存在しており、なぜこのような多様なCBMが存在しているかを考えると、我々が知らないような細胞壁の構造の差異に微生物は対応するために、CBMを進化させたとも考えることができる。微生物ゲノムの解析が進むことにより、今後も新たなCBMが発見されると予想される。一方、CBMの応用例のひとつとして紹介したが、生きた植物の細胞壁にCBMが結合することで、植物側に防御応答を誘導できることがわかった。この他にも、タグとしての利用や、CBMを発現した微生物のセルロース繊維への固定化、CBMとの連結により特定の物質をセルロース上に固定するといった応用例もある31)。酵素本体の触媒活性はもちろん重要であるが、こうした付加ドメインのもつ役割も意味があることを自然は教えている。
謝辞
本研究の一部は、科学研究費補助金20580362、伊藤記念財団、三重大学学内COE-Bの助成により行われました。
文献
1) VanTibeurgh, H., Tomme, P., Claeyssens, M., Bhikhabhai, R., Pettersson, G.: FEBS Lett., 204, 223 (1986).
2) Gilkes, N. R., Henrissat, B., Kilburn, D. G., Miller Jr, R. C., Warren, R. A. J.: Microbiol. Rev., 55, 303 (1991).
3) Cantarel, B. L., Coutinho, P. M., Rancurel, C., Berrnard, T., Lombard, V., Henrissat, B.: Nucleic Acids Res., 37, D233 (2009).
4) Herve, C., Rogowski, A., Blake, A. W., Marcus, S. E., Gilbert, H. J., Knox, J. P.: Proc. Natl. Acad. Sci. USA, 107, 15293 (2010).
5) Boraston, A. B., Bolam, D. N., Gilbert, H. J., Davies, G. J.: Biochem. J., 382, 769 (2004).
6) Hashimoto, H.: Cell. Mol. Life Sci., 63, 2954 (2006).
7) Guillen, D., Sanchez, S., Rodriguez-Sanoja, R.: Appl. Microbiol. Biotechnol., 85, 1241 (2010).
8) Tsukimoto, K., Takada, R., Araki, Y., Suzuki, K., Karita, S., Wakagi, T., Shoun, H., Watanabe, T., Fushinobu, S.: FEBS Lett., 584, 1205 (2010).
9) Lehtio, J., Sugiyama, J., Gustavssion, M., Fransson, L., Linder, M., Teeri, T. T.: Proc. Natl. Acad. Sci. USA, 100, 484 (2003).
10) Igarashi, K., Uchihashi, T., Koivula, A., Wada, M., Kimura, S., Okamoto, T., Penttila, M., Ando, T., Samejima, M.: Science, 333, 1279 (2011).
11) Nimlos, M. R., Beckham, G. T., Mtthews, J. F., Bu, L., Himmel , M. E., Crowley, M. F.; J. Biol. Chem., 287, 20603 (2012).
12) Bae, H.-J., Turcott, G., Chamberland, H., Karita, S., Vezina, L.-P.: FEMS Microbiol. Lett., 227, 175 (2003).
13) Knox, J. P.: Curr. Opin. Plant Biol., 11, 308 (2008).
14) Ding, S.-Y., Xu, Q., Ali, M. K., Baker, J. O., Bayer, E. A., Barak, Y., Lamed, R., Sugiyama, J., Rumbles, G., Himmel, M. E.: BioTechniques, 41, 435 (2006).
15) Blake, A. W., McCartney, L., Flint, J. E., Bolam, D. N., Boraston, A. B., Gilbert, H. J., Knox, J. P.: J. Biol. Chem., 281, 29321 (2006).
16) Araki, Y., Karita, S., Tsuchiya, T., Kondo, M., Goto, M.: Biosci. Biotechnol. Biochem., 74, 802 (2010).
17) Kawakubo, K., Karita, S., Araki, Y., Watanabe, S., Oydomari, M., Takada, R., Tanaka, F., Abe, K., Watanabe, T., Honda, Y., Watanabe, T.: Biotechnol. Bioeng., 105, 499 (2009).
18) Kljun, A., Benians, T. A., Goubet, F., Meulewaeter, F., Knox, J. P., Blackburn, R. S.: Biomacromol., 12, 4121 (2011).
19) Gilkes, N. R., Jervis, E., Henrissat, B., Tekant, B., Miller Jr, R. C., Warren, R. A. J., Kilburn, D. G.: J. Biol. Chem., 267, 6743 (1992).
20) Sugimoto, N., Igarashi, K., Wada, M., Samejima, M.: Langmuir, 28, 14323 (2012).
21) Araki, Y., Karita, S., Tanaka, A., Kondo, M., Goto, M.: Biosci. Biotechnol. Biochem., 73, 1028 (2009).
22) Fontes, C. M., Hazlewood, G. P., Morag, E., Hall, J. B., Hirst, H., Gilbert, H. J.: Biochem. J., 307, 151 (1995).
23) Araki, R., Karita, S., Tanaka, A., Suzuki, M., Kimura, T., Sakka, K.: Biosci. Biotechnol. Biochem., 72, 1322 (2007).
24) Din, N., Gilkes, N. R., Tekant, B., Miller Jr, R. C., Warren, R. A. J., Kilburn, D. G.: Bio/Technol., 9, 1096 (1991).
25) Hall, M., Bansal, P., Lee, J. H., Realff, M. J., Bommarius, A. S.: Bioresource Technol., 102, 2910 (2011).
26) Ciolacu, D., Kovac, J., Kokol, V.: Carbohydr. Res., 345, 621 (2010).
27) Barral, P., Suarez, C., Batanero, E., Alfonso, C., Alche, J. D., Rodriguez-Garcia, M. I., Villalba, M., Rivas, G., Rodriguez, R.: Biochem. J., 390, 77 (2006).
28) Gaulin, E., Drame, N., Lafitte, C., Torto-Alalibo, T., Martinez, Y., Ameline-Torregrosa, C., Khatib, M., Mazarguil, H., Villalba-Mateos, F., Kamoun, S., Mazars, C., Dumas, B., Bottin, A., Esquerre-Tugaye, M.-T., Rickauer, M.: Plant Cell, 18, 1766 (2006).
29) 苅田修一、清水将文:植物防疫, 66, 326 (2012).
30) Dumas, B., Bottin, A., Gaulin, E., Esquerre-Tugaye, M.-T.: Trends Plant Sci., 13, 160 (2008).
31) Shoseyov, O., Shani, Z., Levy, I.: Microbiol. Mol. Biol. Rev., 70, 283 (2006).