�y�g�s�b�N�X�z
Microbial Plastic Factory �|�y�f�H�w�������쓮���鑽���|�����_�n���|
�c������
�k��@�E�H
�P�D�͂��߂�
�ߔN�A�������v���b�g�t�H�[������Ղɕt�����l�̍��������E�ޗ��Y����u���������w�v1) �Ƃ����V�������삪���E�I�����ɂȂ��Ă���B�������זE���H��Ɍ����ĂāA�ړI�������������邽�߂̑�ӌo�H�������I�Ƀf�U�C�������ꂽ���ᕉ�׃v���Z�X�\�z��ڎw���Ă���B�ŋ߂ł́A�������H�� (Microbial Factory) �Ƃ������t���s�����āA�o�C�I�R���A�t�@�C���P�~�J���Y�A�|���}�[�f�ނȂǂ�{�i�I�ɐ�������X�e�[�W�ւƈڍs������B
�{�e�ł́A��X�����߂đn���������_�x�[�X�|���}�[�����p�̔������H��ɂ��ďq�ׂ�B�u���_�́A�������̍זE�O�ɔr�o�����B�v�ƁA��ʂ̋��ȏ��ɋL�q����Ă���B�������A2008�N�ɓ��_���זE���Ńo�C�I�|���}�[���Ɏ�荞�܂��Ƃ��������ۂ��N������2)�B�����j�㏉�߂Ă̂��Ƃł���B�|�����_PLA3)�͂��߁u���_�x�[�X�|���}�[�v���������זE���Ő������ł����삯�ƂȂ����o�����ł���B���̌_�@�ƂȂ����̂́A���ɍy�f�H�w�����̐��ʂł���u���_�d���y�f�v (Lactate-Polymerizing Enzyme: LPE) �̊J�� (����)2)�ł������B
�Q�D���_�d���y�f (LPE) �����܂ł̃v�����[�O
�����̔��������|���G�X�e�����������邱�Ƃ͈ȑO����m���Ă���A�ŏ��̕�́A1926�N�Ƀp�X�c�[���������Ŕ������ꂽ�������|���G�X�e�� (polyhydroxybutyrate (P(3HB)) �ł���BP(3HB) �̃��m�}�[3�q�h���L�V���_ (3-hydroxybutyrate: 3HB) �Ɠ��_�́A�u�q�h���L�V�_�v�Ƃ�����{���w�\�������L���Ă���A�卽�Y�f�Ɍ������鐅�_��̈ʒu���قȂ��Ă���B�������|���G�X�e���́A���̍\�����m�}�[���A���݂Ɏ���܂�3HB���܂�150��ވȏオ����Ă���APHA(polyhydroxyalakanoate)4)�Ƒ��̂���Ă���B
PHA�́A�A���R���̓���A�������邢�͓�_���Y�f���璼�ڔ����������ł��邱�Ƃ���A�����z�^�̊����a�f�ނł���APHA��Ζ��n�v���X�`�b�N�̑�֕i�Ƃ��ė��p����A�J�[�{���j���[�g�����Ƃ����ϓ_�Ńv���X�`�b�N�Y�Ƃ̊����ׂ��y���ł���Ɗ��҂���Ă���B���Ƃ��APHA�����̏����ɕ��ꂽ���f��Ralstonia eutropha���͂��ߑ�����PHA���Y�ۂ�P(3HB)) �Y����BP(3HB) �́A�d���ĐƂ������ł��邱�Ƃ���A���p���̂��߂ɂ͕����̉��P���K�v�ł������B�{�ۑ���������邽�߂̐��I�Ȍ����́A3HB���m�}�[�Ɣ�ׂāA�Y�f����1����3-�q�h���L�V�g���_ (3HV) ���m�}�[�����d��������P(3HB-co-3HV) ���d���|���}�[ (�R�|���}�[) �̐������ł�����5)�B����ɁA��m�}�[�����Ƃ���3HV�����������̒���3-�q�h���L�V�w�L�T���_ (3HHx) �₻��ȏ�̒Y�f�����܂�3-�q�h���L�V�A���J���_ (3HA) ���j�b�g�����A���������コ���邱�ƂŁA�j��L�т����サ�����_��ȃ|���}�[�̍����ɐ������Ă����B2011�N5���ɂ́A�����̇��J�l�J���AP(3HB-co-3HHx) ���d���|���}�[ (�o�^���W�F�A�I�j���b�N�X) �̐�g���X�P�[���ł̐��Y���Ƃ��J�n����6)�B�}1�ɁA�����̓��e���܂Ƃ߂Ď����B
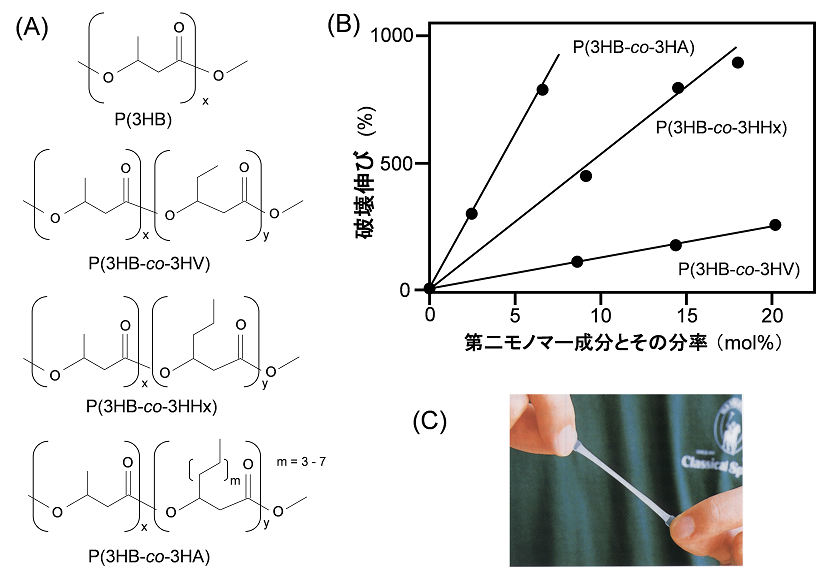

��̓I�ɂ́APHA�d���y�f��l�H�i�������邱�Ƃɂ���āA�]�݂̕����������PHA�|���}�[�����݂ɍ������邱�Ƃ�ڎw���Ă����B���̍ہA���L������ِ���L����Pseudomonas sp. 61-3�R����PHA�d���y�f (PhaC1PS) ���o���y�f�Ƃ��đI�肵��7)�BPHA�d���y�f�́A�}2�Ɏ����悤�Ɋ������SCys���N�_�Ƀ��m�}�[�����[�h����A��ʉ��������̃T�u���j�b�g�Ƃ̊ԂŁA�|���}�[�L���ƃ��m�}�[���[�h�Ƃ����݂ɌJ��Ԃ���Ȃ���d�����郁�J�j�Y��������Ă���BPhaC1PS�́A��������Ȃ�3HA���j�b�g�̎�荞�݂͗e�Ղł��������A3HB���j�b�g�̎�荞�ݔ\�͂�����ł���Ƃ����ۑ肪�������B�����ŁA3HB�ɑ��锽�������������邽�߂̍y�f���ς�P(3HB) �z���|���}�[�̒~�ϔ\�͂̌�����w�W�Ɏ��{�����B�{�y�f�́A�����̌����҂����킵�Ă���̂ɂ�������炸�A���̍\�������𖾂ł���A���ώw�j�𗧂Ă邱�Ƃ�����ł������B�����ŁA�ԗ��I�Ȉ�`�q�ψقƋ@�\�I���ɂ��i���H�w�I�A�v���[�`�����p����8)�B

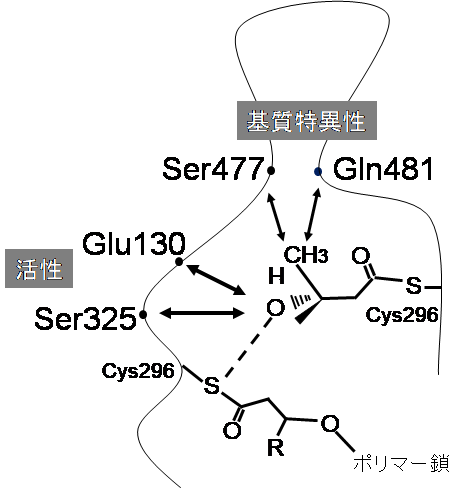
�܂��APHA�d����`�q�̑S��ɑ��āA1�`2�A�~�m�_�u����������悤�Ƀ����_���ψق������B���̏d���y�f��`�q��咰�ۂŃ��C�u�����[�����A�n�C�X���[�v�b�g�X�N���[�j���O�@�ɂ��A���|���}�[�~�ϊ��̎擾��ڎw�����B�X�N���[�j���O�@�́A�|���}�[�������� (3HB�ɑ��锽����) ��咰�ۃR���j�[�̔��F���x�Ŕ���ł���v���[�g�A�b�Z�C�n��p�����B�I�����ꂽ�ψّ̂́A�����ǃ��x���Ŕ|�{��HPLC�ɂ����P(3HB) �~�ϔ\���ʓI�ɒ��ׁA�ψٕ��ʂƒu���A�~�m�_�Ɋւ�������W�ς����B�ŏI�I�ɂ́A3HB�ɍ������������D�Ǖψٍy�f (��ꐢ��) �̎擾�ɐ��������B�}3�́A��������ъ���ِ��Ɋ֗^����4�̕��ʂ̈ʒu�W��͎��I�Ɏ����Ă���B�ψقɔ����@�\�̕ϓ������𗊂�ɕ����яオ���Ă������̐���\�����A������𖾂����ł��낤���̍\���Ƃǂ̒��x�d�Ȃ荇����������y���݂ł���B

����ɁA����ꂽ�ψّ̂̃V���O���D�Ǖψق��n���I�������I�ɑg�ݍ��킹���B���̌��ʁAP(3HB) �~�ϔ\�͂��쐶�^�Ɣ�r���Ė�420�{�ɂ܂Ō��シ����̗D�Ǖψٍy�f��n�����邱�Ƃɐ�������9)�B����ꂽ�����i���^PHA�d���y�f���C�u�����[��p���āA�{��ł��郂�m�}�[�g�����䂪�\�����������B���̌��ʁA���X�̖쐶�^PhaC1PS����������R�|���}�[����3HB������14%�ł������̂ɑ��āA70%�܂ő啝�ɑ��������R�|���}�[���������ꂽ�B�������āA���҂��PHA�d���y�f�̕��q�i���ɂ��y�f�̊���ِ���ϊ����A���d��PHA�̃��m�}�[�g�����L�͈͂����ׂɕω������邱�Ƃɐ��������B���̌����̐��ʂɂ��A�p�r�ɉ������g������Ȃ�R�|���}�[�̍����𑽂��̕ψك��C�u�����[�����p���邱�ƂŁA�g�e�[���[���[�h�h�Ɏ����ł���悤�ɂȂ����B
���́A���̉�������Ɂu���_�|���}�[�����v�Ƃ����n�����˔@�Ƃ��Ėڂ̑O�Ɍ���Ă����̂ł���B�ł͂Ȃ��A���_���d������鎖���d�v�Ȃ̂��Ƃ����ƁA�|�����_ (PLA) ���o�C�I�|���}�[�̒��ł�����������H���ɗD��A�ł����p������Ă���ޗ�������ł���B���ہAPHA�̐������@�\�����p���ă|�����_���������悤�Ƃ��錤���́A�ȑO���琢�E�̒����Ȍ����@�ւōs���Ă����B��ɂ��q�ׂ��悤�ɁA���w�\������l����A���_ (2-hydroxypropionate) ��PHA�t�@�~���[�ɑ������i�\���ƍl�����邪�A�߂��ĉ������݂ł������B���̒��ŁA��X����|�������_���d��������V�X�e�����̉ߒ��ɂ́A�������́g���R�̔����h���������B�������A�ő�̔��� (�����H) �͓��_�d���y�f�ł���B
�R�D���_�d���y�f (LPE) �̔���������_�|���}�[�������H��a���܂�
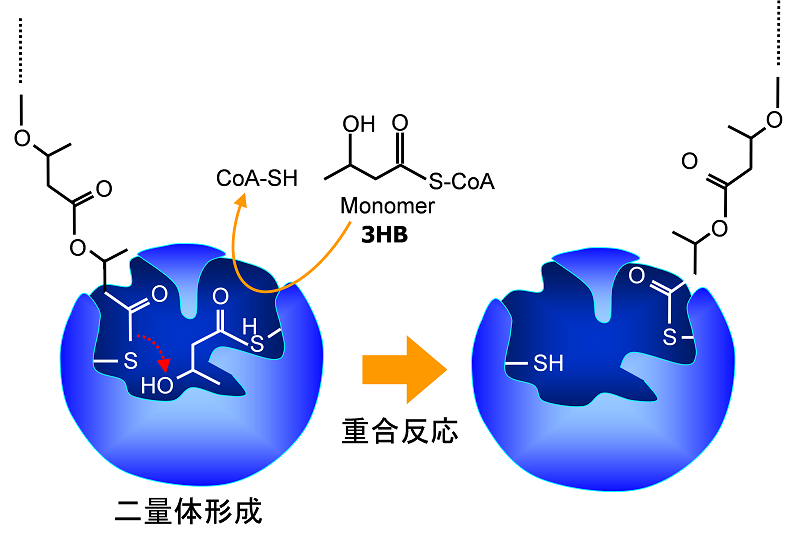
���X�V�R���m�}�[��ł���3HB-CoA���d���ł���PHA�d���y�f���A�������_��CoA�̂ł��郉�N�`��CoA (LA-CoA) �����l�ɏd���ł���A�זE���Ń|�����_�������������ł��낤�B���̉\����������ɂ́APHA�d���y�f�����Ain vitro�ł�LA-CoA�Ƃ̔����𑪒肷��悢�B�������A���̕��@�ł͓��_�̏d���͌����Ȃ������B�����ŁA��X�͊w�����������ň�̍H�v�������B����́ALA-CoA�����łȂ�3HB-CoA���������������Ŋ�����]���������Ƃł���B����ƁA�����܂������ǂ̒����������������B�����A���������|���}�[�͂��Ă݂�ƁA3HB�|���}�[�����ɓ��_���m�}�[���g�ݍ��܂�Ă��邱�Ƃ����������B�܂�A�g����hPHA�d���y�f�́A���_���m�}�[��P�̂ł͏d�����Ȃ����A���d���̂ł�������ł����̂ł���2,10)�B
�ł��d�v�ȓ_�́A�g����hPHA�d���y�f���A�i���H�w�I�ɉ��ς��ꂽ�������ψّ̂ł��������Ƃł���B����6�N�Ԃ����APHA�����y�f�̂������̍������ψّ̂��쐻���Ă����B�����̕ψّ͓̂��_���d�����鎖��ړI�ɍ쐬���ꂽ�킯�ł͂Ȃ������B�������A���ʓI�ɁA���̒��̈�̕ψّ݂̂̂��A��q�������d���̂̍��������ŁA���_�d�������������A����܂ŒN���B���ł��Ȃ��������_�d���y�f (LPE�Ɩ���) �̔����ɂȂ������̂ł���B
�����A�������H��� (�咰��) �ɍ\�z�������m�}�[�������[�g�ɉ����A��������LPE����`�q�������쓮���邩�ׂ��B�܂��A�}4�Ɏ����悤�ȓ��_�|���}�[�̐������o�H��咰�ۓ��ɐv�����B�\�������X�e�b�v�́A(1) ���_�����A(2) ���m�}�[�����y�f�ɂ�郂�m�}�[ (LA-CoA) �̍����A����� (3) LPE�ɂ��d���ł���BLA-CoA���m�}�[�́A���_�E���f�y�f (LDH) �̔������瓾����_���v���s�I�j��CoA�]�ڍy�f (PCT) �ɂ����CoA���t������邱�Ƃō��������o�H��v�����B���ۂɊO��PCT��`�q��咰�ۓ��Ŕ������āALA-CoA��CE/MS���͂Ō��o���邱�Ƃɂ��A���_���m�}�[�����o�H�̍\�z���m�F�����B�܂��A�y�A�ƂȂ�3HB-CoA�����o�H�Ƃ��ẮA����܂ł̋����y�f��`�q��p���������𗘗p�����B����狟�����ꂽ���m�}�[���ŏI�I�ɓ��_�d���y�f���d������Ƃ����f�U�C���ł���B
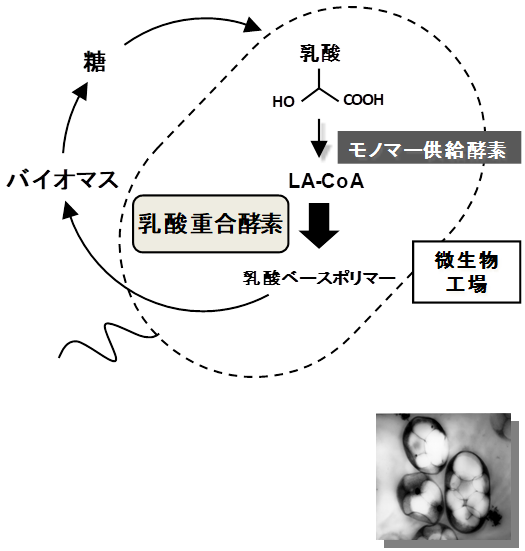

�ʂ����āA�咰�ۑ̓��ō������ꂽ�|���}�[�𒊏o�����͂������ʁALA���j�b�g��6 mol%�������ꂽP (LA-co-3HB) �R�|���}�[�ł��邱�Ƃ����炩�ƂȂ���2)�B�v�ʂ�ɁA�������H�ꂪ�쓮���o�����̂ł���B���n�ʼn��a�ɏd���������i�ޖ{�v���Z�X�́A���w�����ł̏d���������ɗp����L�Q�ȏd�����G�}��L�@�n�}���g�p���Ȃ��ōςނ��Ƃ�A��������������v�����Ȃ����Ƃ��獇���ߒ��ł̊����ׂ̌y�������҂����B
�S�D�����_�����̓��_�|���}�[�̍���
�v���g�^�C�v�������H��ł́A�R�|���}�[���ɑ��݂���LA������6%�ɗ��܂��Ă������A����ɕ��������コ���邱�Ƃ��\�ł��낤���H������X�́A���m�}�[�O��̂ł�����_�̍����ʂ��A���_���������߂�v���ɂȂ�ƍl�����B���ہA���C�|�{�ɂ����_�����̑��i��A���_�������Y�����Ӊ��ϊ��Ȃǂ𗘗p���邱�Ƃɂ��A���_������50%�߂��܂ŏ㏸�����邱�Ƃɐ�������11)�B�����ŁA����ɓ��_���������コ���邽�߂ɁA��X�͉ߋ��Ɏ擾�����d�����������コ����D�Ǖψق�g�ݍ��킹�ĎO�d�ψق�L����V���Ȑi���^LPE��n�o���A�����60%�܂œ��_�������㏸�����邱�Ƃɐ�������12)�B�������Ȃ���A���̍y�f��p���Ă��A�|�����_���������悤�Ƃ���ƁA�|���}�[�������Ȃ����Ƃ���A���_�̏d���ɂ��̑��̃��j�b�g���K�v�ł��邱�Ƃ������������ꂽ�B���Ȃ킿�A�������|���G�X�e���̏d�����J�j�Y���̍����Ɋւ�����N�ƂȂ��Ă���A���ݏڍׂȉ�͂�i�߂Ă���B
�T�D�R���l�ۂɂ�钴�����_�����|���}�[�̐��Y
����܂ł̃|���}�[���������ł́A���쐫�ɗD�ꂽ�咰�ۂ�p���čs���Ă����B��������X�́A�R���l�ۂ��h��Ƃ���P(3HB) �̍����n�����łɍ\�z���Ă����B�R���l�ۂ́A���S�Ȏ��p�A�~�m�_���Y�ۂƂ��Ă̎��т�����A���Z�x�̓��t�������悭�������A�܂������x�|�{���\�ł���Ȃǂ̃����b�g������B�ł́A�R���l�ۂ��g���Ă����l�ȓ��_�|���}�[�̐��Y���ł���̂��낤���H�����́A���̒P���ȓ��@�Ō������J�n�������A����ꂽ���ʂ͋����ׂ����̂������B�Ȃ�Ɠ��_������99%����|���}�[���������ꂽ�̂ł���B�ŏ��A�����������w���́A3HB�̃s�[�N�������Ȃ����߁A�uPLA�������ł��܂����I�v�ƕ��Ă����B�������A3HB���m�}�[�̕K�v���������F�����Ă����M�҂�́A����ɍ������x�ŕ��͂���悤�Ɏw�������B���̌��ʁA�ɔ��ʂł͂��邪�A��͂�3HB���܂܂�Ă��鎖����������13)�B���̂悤�ȍ����_�����̃|���}�[�́A���Ȃ��Ƃ��M�I�����Ɋւ��ẮA�X�e���I�R���v���b�N�X�`���ɂ��Z�_�㏸���܂߁A���w����PLA�Ƃقړ����̒l���������B���������āA�ޗ��Ȋw�̊ϓ_����́APLA���������ł����ƌ����Ă悢�B
�����̌��ʂ���A���������ł�����_�̍����ʂ傳���邱�ƁA���_�d���y�f�̊������������邱�ƂȂǁA�u�v���X�v�̐헪�����łȂ��A�ƂȂ�3HB���m�}�[�̋����͂𐧌�����u�}�C�i�X�v�̐헪 (�����Ă��_���Ȃ�����Ă݂ȁI) ���L���ł��邱�Ƃ��킩��B
�U�D�����̑I����_�d���y�f�ɂ�鍂���w���x�|���}�[�̍���
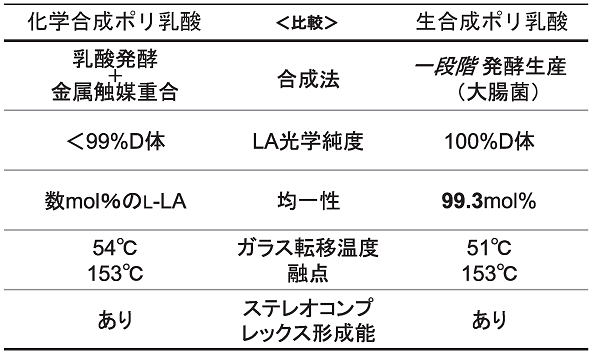
�������H��ɂ�镨�����Y�̑傫�ȃA�h�o���e�[�W�̈�ɁA���ِ̈��̂���ɍ������x�őI��I�ɍ����\�ł��邱�Ƃ��������邾�낤�B��X�������������_�|���}�[�������������ĕ��͂����Ƃ���A�ق�100%D���_����\������Ă���������������11)�B���Ȃ킿�A���ڂ���Ă�����_�d���y�f�̗��̑I�𐫂ɋN������B�|���G�X�e���d���y�f�͔��Ɍ�����D�̑I�𐫂�L���Ă��鎖���m���邪�A���̐����͓��_�̏d���ɂ��p������Ă����B����́A3�q�h���L�V����Ƃ̍\����r����A�G�X�e�������Ɋ֗^���鐅�_��ƃJ���{�L�V����Ƃ̈ʒu�W�����]���Ă��鎖���l����Ƃ��Ȃ苻���[�����ł���B�|�����_�ɂ́APLLA��PDLA�̗����w�ِ��|���}�[�������݂��ċ������������邱�ƂŃX�e���I�R���v���b�N�X���`�����A�M�Z�����x�����シ�邱�Ƃ��m���Ă���A���w�ِ��̂̍�蕪�����d�v�ł���B���݁A�Y�ƓI�ɐ��Y����Ă���PLA�̎嗬��PLLA�ł���A�������H��Ő��Y�����PDLA (�ɋ߂��|���}�[) �̓X�e���I�R���v���b�N�X�̃p�[�g�i�[�Ƃ��Ă̗��p����ϊ��҂ł��邾�낤�B�\1�ɁA���w����PDLA�Ɛ�����PDLA�Ɋւ��āA�\���╨���E�@�\�̊ϓ_�����r�Ƃ��Ă܂Ƃ߂Ď�����14,15)�B��ϋ����[���Δ�ƂȂ��Ă���B
�\1�@���w�����|�����_�Ɛ������|�����_�Ƃ̔�r
�V�D�����|�����_�̍\�����l��
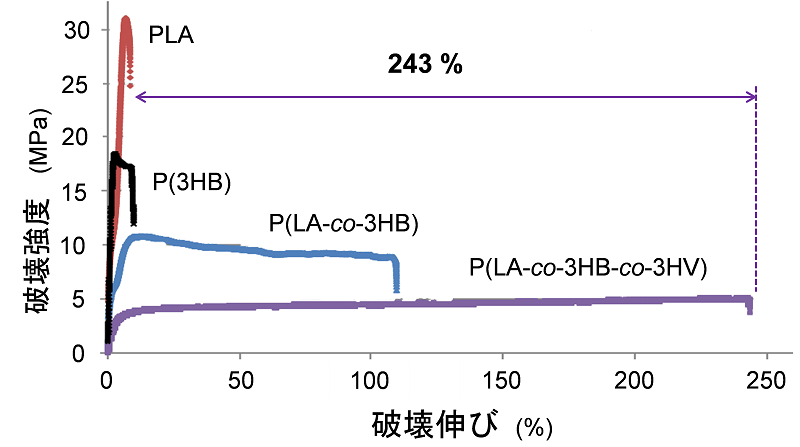
���āA���̂悤�ɂ��č������ꂽP(LA-co-3HB) �₳��ɂ�3�����m�}�[����Ȃ�P(LA-co-3HB-co-3HV) �R�|���}�[16) �́A�z���|���}�[�ł���PLA�Ƃ�P(3HB) �Ƃ��قȂ�V���ȃo�C�I�|���}�[�ł���B�ł́A���̃R�|���}�[�͂ǂ̂悤�Ȑ����������Ă��邾�낤���H��X�́A�t�@�[�����^�[�Ŕ|�{���Y�����|���}�[���t�B���������A�@�B�I�����ׂ��B���̌��ʁA�ʐ^ (�}5) �Ɏ����悤�ȓ����ȃt�B�������ł��A����ɂ��̃t�B�����́A��������ƐL������_��������Ă����B���̐����́APLA��P(3HB) ���قƂ�ǐL�����������Ȃ��̂ƑΏƓI�ł������B�}6�ɁA���̓T�^�I�Ȍ��ʂ������B���̌��ʂ́A���_�|���}�[�������������A�P�ɉ��w�����v���Z�X����̒u������ (�v���Z�X�C�m�x�[�V����) �ɗ��܂炸�A�V���ȍޗ��ݏo������ (�ޗ��C�m�x�[�V����) ���ł��邱�Ƃ������Ă���B

![]()
�}6�@���_�x�[�X�|���}�[�̋@�B�I����
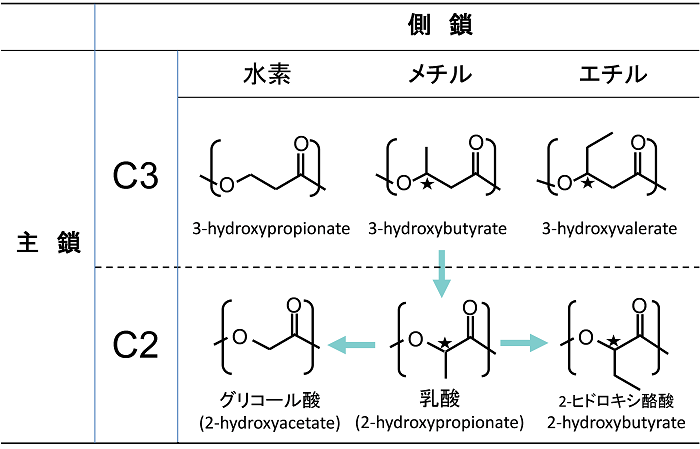
���̎��_����A�u�|�����_�v����u�����|�����_�v�Ƃ����V���Ȃ�J�e�S���[�����܂�Ă����B���Ȃ킿�ALPE�̕��L������ِ��𗘗p���āA���_���x�[�X�ɑ��l�ȃ��m�}�[�����d�������ꂽ�R�|���}�[�̍������\�ƂȂ�B���ہA3HB�ȊO�̑��탂�m�}�[�Ƃ̋��d���̂̑n���ɐ������Ă���B�\2�ł́A���m�}�[�ɂ�����卽�\���̑��l���ɒ��ڂ��Ă���B����܂ŁA3�ʂɐ��_���L���鎉�b�_�ɑ��ẮA150��ȏ�̃��m�}�[�ɑ��ďd���\�ł������B���̂��Ƃ́A�����̍\�����l���ɂ�PHA�d���y�f�͋ɂ߂čL�����������������ƈӖ�����B����A���_�̂悤��2�ʂɐ��_���L���鎉�b�_���m�}�[�ɑ��Ă͑S���d������������PHA�d���y�f������邱�Ƃ͂Ȃ������B����̓��_�d���y�f�̊J������ɁA���m�}�[����O���R�[���_17) ��2-�q�h���L�V���_ (2HB)18) �ɑ��Ă��d�������������ψّ̂��������Ă��Ă���B���������āA�����|�����_�̍\�����l���̊g���ɑ傫�Ȋ�^�������炵���B���݁A���̈�Íޗ��Ƃ��ĉ��p����Ă���|���O���R�[���_���邢�͓��_�ƃO���R�[���_�̃R�|���}�[�Ȃǂ��A�{�i�I�ɔ������H��ɂ���ă����|�b�g�����ł��鎞�オ�n�܂����B
�\2�@�����|�����_�̎卽�̍\�����l��
�W�D�����W�]
LPE�̔����ɂ��A���̓��_�|���}�[���Y�p�̔������H����\�z�ł����B�������v���b�g�t�H�[���́A�咰�ۂ���X�^�[�g���A���݂̓R���l�ۂȂǑ���������ւƗp�r�ɉ����Ċg�����Ă���B����́A�������H��̋��݂ŁA�|���}�[�������p�̃\�t�g�E�G�A���^�[�Q�b�g�������ɈڐA���邱�ƂŃt���L�V�u���ɉ^�p�ł���B����͌����ƂȂ��H�o�C�I�}�X�����Ƃ̑g�ݍ��킹���l�����A�o�C�I�}�X�����������������h��Ƃ̑g�ݍ��킹�ɂ��A�Đ��\�ȒY�f�����獂�t�����l�ȃo�C�I�|���}�[�Y���邱�Ƃ��ڕW�ł���19)�B�Ⴆ�A������������_���Y�f�Ȃ�A������Ђ��Ă͐A���̂��|���}�[���Y�v���b�g�t�H�[���ɂȂ�20)�B
�K���A���N�x����̐V�v���W�F�N�g (JST-CREST) �u��_���Y�f�̌����I�������̎����̂��߂̐A���������@�\��o�C�I�}�X�̗����p�Z�p���̊�ՋZ�p�̑n�o�v�̈�ۑ肪�J�n���邱�ƂɂȂ����B��_���Y�f�����C�ʊтɍ����\�E���@�\�o�C�I�v���X�`�b�N��n�����閲�̃v���W�F�N�g���i�Ɍ����A�y�f�H�w�̊ϓ_������v���ł�����K���ł���B
�ӎ�
�{�e�ŏЉ���������ʂ̑����́A�k���������������уg���^�����ԁE�L�c�����Ƃ̎Y�w�A�g���������ɂ���ē���ꂽ���̂ł���B�{�e�̎������ʂ́A��ɓ��������̏��{����Y�y�����A�R�c���a���m (������w����)�A���������m (�����̑f�Ќ�����)�A�v��z (�����������m�ے��w��) �ɂ����̂ł���B�܂��A(��) �Ȋw�Z�p�U���@�\ (JST) ��CREST�v���W�F�N�g�u��_���Y�f�̎������v�̈�Ƃ��čs��ꂽ�B
����
1) Carothers, J. M., Goler, J. A., Keasling, J. D.: Curr. Opin. Biotechnol., 20, 498 (2009).
2) Taguchi, S., Yamada, M., Matsumoto, K., Tajima, K., Satoh, Y., Munekata, M., Ohno, K., Kohda, K., Shimamura, T., Kambe, H., Obata, S.: Proc. Natl. Acad. Sci. USA, 105, 17323 (2008).
3) Tsuji, H.: Biopolymers, Wiley-VCH, 4, 129 (2002).
4) Madison, L. L., Huisman, G. W.: Microbiol. Mol. Biol. Rev., 63, 21 (1999).
5) Taguchi, S., Tsuge, T.: Protein Engineering Handbook, Edited by S. Lutz, U.T. Bornschuer, WILEY-VCH, 877 (2009).
6) �O�؍N�O�F�����q, 61, 197 (2012).
7) Matsusaki, H., Manji, K., Taguchi, K., Kato, M., Fukui, T., Doi, Y.: J. Bacteriol., 180, 6459 (1998).
8) Taguchi, S., Doi, Y.: Macromol. Biosci., 4, 145 (2004).
9) Nomura, C. T., Taguchi, S.: Appl. Microbiol. Biotechnol., 73, 969 (2007).
10) Tajima, K., Satoh, Y., Satoh, T., Itoh, R., Han, X., Taguchi, S., Kakuchi, T., Munekata, M.: Macromolecules, 42, 1985 (2009).
11) Yamada, M., Matsumoto, K., Nakai, T., Taguchi, S.: Biomacromolecules, 10, 677 (2009).
12) Yamada, M., Matsumoto, K., Shimizu, K., Uramoto, S., Nakai, T., Taguchi, S.: Biomacromolecules, 11, 815 (2010).
13) Song, Y., Matsumoto, K., Yamada, M., Gohda, A., Brigham, C. J., Sinskey, A. J., Taguchi, S.: Appl. Microbiol. Biotechnol., 93, 1917 (2012).
14) Shozui, F., Matsumoto, K., Motohashi, R., Sun, J., Satoh, T., Kakuchi, T., Taguchi, S.: Polym. Degrad. Stab., 96, 499 (2011).
15) Yamada, M., Matsumoto, K., Uramoto, S., Motohashi, R., Abe, H., Taguchi, S.: J. Biotechnol., 154, 255 (2011).
16) Shozui, F., Matsumoto, K., Nakai, T., Yamada, M., Taguchi, S.: Appl. Microbiol. Biotechnol., 85, 949 (2010).
17) Matsumoto, K., Ishiyama, A., Sakai, K., Shiba, T., Taguchi, S.: J. Biotechnol., 156, 214 (2011).
18) Han, X., Satoh, Y., Satoh, T., Matsumoto, K., Kakuchi, T., Taguchi, S., Dairi, T., Munekata, M., Tajima, K.: Appl. Microbiol. Biotechnol., 92, 509 (2011).
19) Nduko, J. M., Matsumoto, K., Taguchi, S.: Biobased Monomers, Polymers, and Materials, ACS Symp. Ser., Chapter 14, 213 (2012).
20) Matsumoto, K., Murata, T., Nagao, R., Nomura, C. T., Arai, S., Arai, Y., Takase, K., Nakashita, H., Taguchi, S., Shimada, H.: Biomacromolecules, 13, 686 (2009).