【トピックス】
バクテリアの新規ヘム合成関連遺伝子
加登一成
日本曹達 小田原研究所
1.はじめに
ヘムやクロロフィルなどのポルフィリン化合物は、呼吸や光合成において重要な役割を担っている。ヘムはさまざまなタンパク質 (酵素) に結合して、いろいろな酸化還元反応を触媒する。ヘムが関与する酸化還元反応の中で、特に顕著なのは呼吸と光合成であるが、それ以外にも活性酸素の消去や解毒作用などさまざまな反応に関与している。植物の場合は全遺伝子の2%以上がヘム結合タンパク質をコードしていると見積もられている1)。ラン藻の場合も少なくとも全遺伝子の1%以上はヘム結合タンパク質をコードしていると思われ、これらの数字からもヘムが細胞の中でいかに重要かが伺える。一方、クロロフィル (葉緑素) は、光合成に必須の分子で、光を吸収する、光のエネルギーを使って電子を移動させるなどの機能を持っている。
今回発見したのは、ヘムやクロロフィルの合成に必要な遺伝子である (図1)。ポルフィリン生合成経路のうち、5-アミノレブリン酸から、プロトポルフィリンIXまでの6段階の酵素反応は、全ての生物に共通の反応であると考えられている2)。プロトポルフィリノーゲンⅨ酸化酵素 (Protox) は、プロトポルフィリンIX生合成の最終段階において、プロトポルフィリノーゲンⅨ (Protogen IX) から6個の電子を奪ってプロトポルフィリンIX (Proto IX) を合成する鍵酵素である3)。これまで、真核生物および好気性細菌はhemY遺伝子にコードされるおよそ55kDaのProtoxを2,4,5)、大腸菌やサルモネラ菌はhemG遺伝子にコードされるおよそ21 kDaのProtoxを持っていることが明らかになっている6,7)。しかし、ゲノム配列の解析されたラン藻の多くの種においてはhemYまたはhemGのホモログは見つかっていない2,8)。
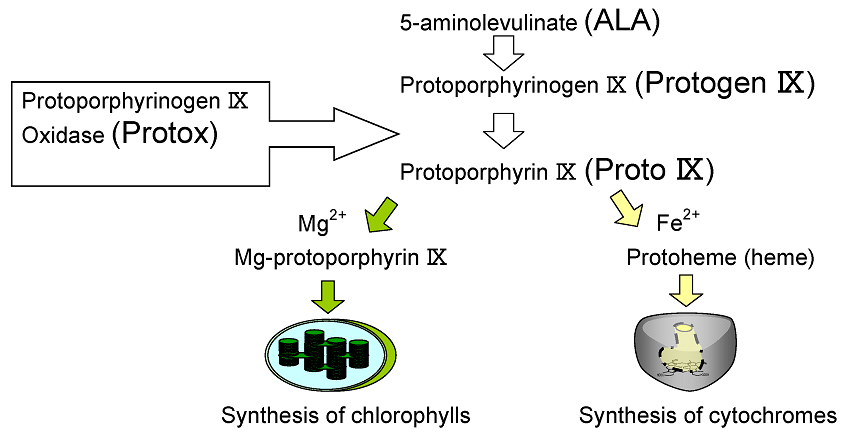
図1 ヘムとクロロフィルの共通合成経路
これはクロロフィルやヘム合成の研究者にとっては予想外のことであった。なぜなら、もともとは光合成能力がなかった植物の祖先が、光合成能力を持つラン藻の遺伝子を取り込んで光合成能力を獲得したというのが定説であり、クロロフィル合成に必要な遺伝子は全てラン藻から受け継がれていると考えられていたからである9)。
実際になぜ、ラン藻にはヘム/クロロフィル合成酵素のうち、Protoxの遺伝子だけが見つからないのか?我々は、ラン藻には、植物とは全く異なるタイプのProtoxが存在して、この反応を担っているからではないかと考えた。本稿では、ラン藻Synechocystis sp. PCC 6803のProtox遺伝子に関する研究について紹介したい。
2.ラン藻Protox遺伝子の同定を目指して
我々は遺伝学的手法を用いてラン藻Protox遺伝子を同定する計画を立てた。遺伝学的手法とは簡単にいうと以下のような方法である。ラン藻はおよそ3000個の遺伝子を持っているため、これらの遺伝子にトランスポゾンが入ったラン藻の変異株のプールを作り出し、次にこれらの様々な変異株の中からProtoxの活性がなくなったラン藻の変異株を見つけ出す。この変異株のどこにトランスポゾンが入っているのかを調べるとProtoxの活性に必要な遺伝子がわかるはずである。このような方法は最近の生物学では極めて一般的な手法であるが、今回の目的では単純にこのような手法を利用することはできなかった。なぜならProtoxの活性がなくなったラン藻はヘムやクロロフィルを合成することができず、死滅してしまうはずで、そうなれば変異株を見つけ出すことは不可能と考えられるからである。そこで、我々は以下のようなストラテジーを考えた (図2)。
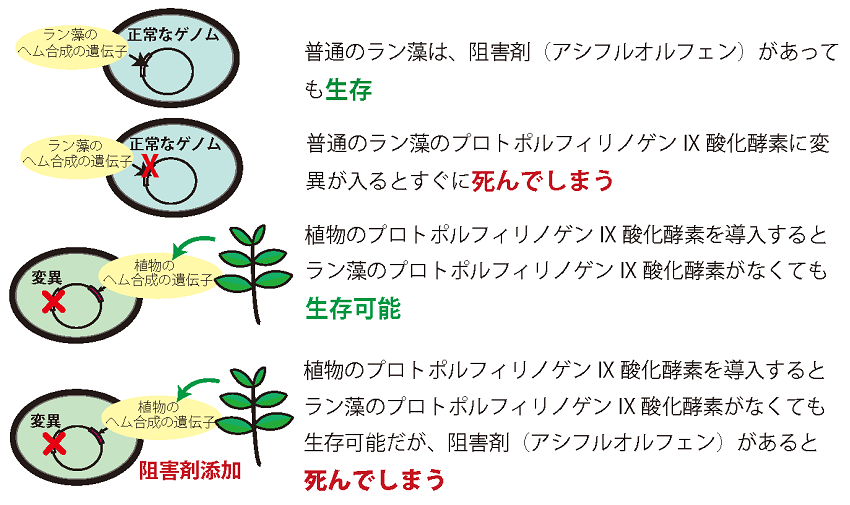
図2 ラン藻Protox遺伝子同定のストラテジー
1. あらかじめシロイヌナズナ由来Protox遺伝子を導入したラン藻形質転換株を作成する (AT株と命名)
2. AT株に対してトランスポゾンを用いたランダムな遺伝子破壊を行い、ラン藻変異株のプールを用意する10)。もし、ラン藻のProtoxの遺伝子に変異があっても、植物のProtoxの遺伝子があるので、ラン藻は死滅しないはず。
3. ここで、植物のProtox阻害剤 (ジフェニルエーテル系除草剤acifluorfen) をラン藻変異株に処理する11)。もし、ラン藻のProtoxの遺伝子に変異があって、しかも植物のProtoxが阻害剤で阻害されれば、ここで初めてラン藻のProtoxの変異株は死滅するはず。
簡単にまとめると、上記のようなストラテジーでスクリーニングを行い、通常は問題なく生育するが、植物のProtox阻害剤の存在下では死滅する変異株を調べればラン藻のProtoxに変異が入っているはずである。このようなストラテジーは、理屈の上ではうまくいくはずであるが、実際には「植物のProtoxがあればラン藻の本来のProtoxがなくても生きていけるのだろうか?」「植物のProtoxを持つラン藻に対して阻害剤がうまく効果を示すだろうか?」などとクリアしなければならない条件が多くある。このようなストラテジーを使って変異株を探索した、という報告はなくリスクを伴う方法であった。
3.Acifluorfenを利用したラン藻変異株スクリーニング
先述のストラテジーに従い、acifluorfenに対する感受性を指標にラン藻変異株スクリーニングを実施した。その結果、acifluorfenの存在下で特異的に死滅するラン藻の変異株を単離することに成功した。トランスポゾンタグの挿入位置を解析したところ、ラン藻ゲノムの256677番目のTから256685番目のG間にタグが挿入されていることがわかった。この位置はORF領域には含まれていないが、下流にある推定タンパク質slr1790 (256698-257279, 193aa) の転写調節領域の可能性が考えられた。その他、単離した変異株としては呼吸鎖のNADH dehydrogenase I chain Lをコードするslr0844の変異株が該当した。この変異株はacifluorfenの存在下で生育遅延を示す程度であり、先述のslr1790遺伝子の上流にタグが挿入された変異株の致死的な表現系とは明らかに異なった。
4.ラン藻slr1790遺伝子破壊株の解析
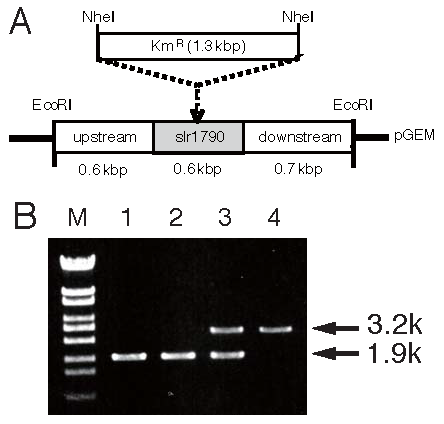
slr1790遺伝子のコードするSlr1790タンパク質の機能を解析するために、ラン藻野生株 (WT株)、シロイヌナズナProtox遺伝子を導入したラン藻形質転換株 (AT株) についてslr1790遺伝子のコード領域中央部をカナマイシンカセットで分断するコンストラクトを利用して破壊株の作成を試みた (WTSK株、ATSK株と命名。図3A)。ラン藻は細胞内に多コピーでゲノムDNAが存在しているため、得られた形質転換株についてカナマイシンを含んだBG11 寒天培地で数回継代を繰り返し、置換された遺伝子のコピーの割合をPCR法にて見積もった。プライマーとしてはslr1790遺伝子の前後数百bp付近より設計したものを使用した。この設計では、野生型ゲノムの場合は1.9kbpのバンドが、カナマイシンカセットが挿入された変異型の場合、カナマイシンカセットのサイズ (1.3kbp) だけ大きな3.2kbpのバンドが増幅される。結果、WTSK株に関しては1.9kbpと3.2kbpのバンドが増幅され遺伝子破壊は不完全に、ATSK株に関しては3.2kbpのバンドが増幅され遺伝破壊が完全に進行したことを確認した (図3B)。この結果は、slr1790遺伝子は必須遺伝子であり、その機能欠損は事前に導入したシロイヌナズナ由来Protox遺伝子により相補されることを意味する。

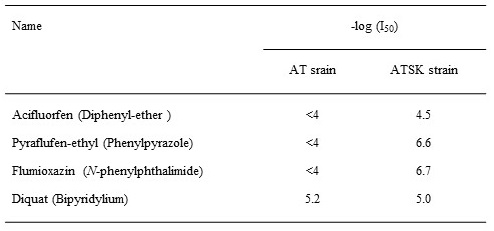
次に作成した遺伝子破壊株について、acifluorfenの存在下、非存在下での生育比較を行った。その結果、WTSK株はacifluorfenの有無に関わらず、WT株と比較して生育遅延する傾向が認められた (図4A)。また、ATSK株に関してはacifluorfenの非存在下では生育に変化は認められないものの、acifluorfenの存在下ではWT株、AT株、WTSK株と比較して特異的に生育が抑制さることを確認した (図4B)。これらの結果は、slr1790の完全な遺伝子破壊は、ラン藻内在のProtox活性を完全に消失させたことを示す。また、acifluorfenとは構造の異なるpyraflufen-ethylやflumioxazinといった他のProtox阻害型除草剤を処理した場合においても、acifluorfen処理と同様にAT株は生育に影響を受けず、ATSK株は生育阻害を受けた (表1)。なお、Protox阻害とは作用が異なるdiquatを処理した場合は、AT株、ATSK株ともに同濃度で生育阻害が認められた。
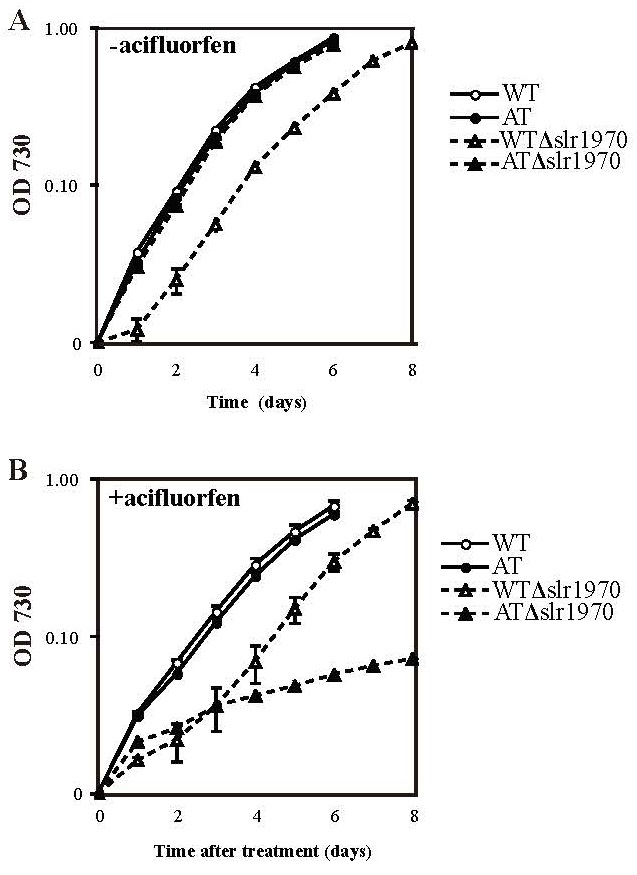

表1 ラン藻slr1790遺伝子破壊株に対するProtox阻害型除草剤およびdiquatの効果
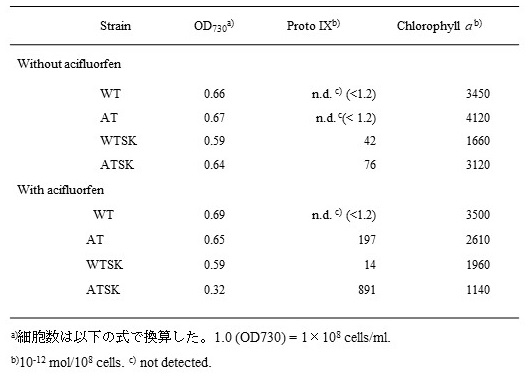
次に作成した遺伝子破壊株について、HPLCにて色素分析を実施した。その結果、WTSK株において、Protox変異株に特徴的なProto IXの異常蓄積が認められ、最終産物であるクロロフィルaの量はWT株と比較してacifluorfen処理の有無に関わらず、約半分に低下した (表2)。一方、ATSK株においてはacifluorfenを処理した場合にのみ他の株には認められないProto IXの異常蓄積が認められた (表2)。
表2 ラン藻slr1790遺伝子破壊株の色素分析
以上、変異株の解析によりslr1790遺伝子がラン藻のProtoxの本体あるいはサブユニットをコードすることが明らかとなった。
5.光合成細菌slr1790ホモログのProtox酵素活性
Protox活性をin vitroで確認するため、ラン藻Synechocystis sp. PCC 6803のslr1790遺伝子について、大腸菌やバキュロウイルス等の様々なタンパク質発現系を用いて検討したが、いずれもタンパク質の発現量が極めて低く酵素活性の測定には至らなかった。Slr1790タンパク質は膜貫通構造を持つことが予想され、タンパク質発現から酵素活性の確認に至るまでには試行錯誤を繰り返したが、最終的には光合成細菌Rhodobacter spaeroidesのslr1790ホモログ (Rs-slr1790) を使って、Protox活性を示すことに成功した12)(図5)。
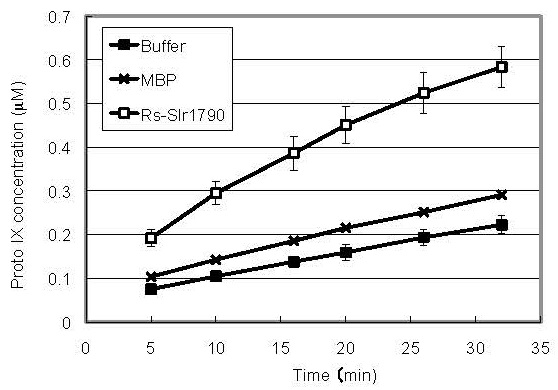

この結果が得られたことより、slr1790遺伝子はラン藻のProtoxをコードしていると最終的に証明することができ、我々はこの遺伝子をhemJ遺伝子と名付けた。
6.slr1790 (hemJ) ホモログの系統発生解析
slr1790遺伝子は193アミノ酸をコードし、5回膜貫通構造を有することが予想された。相同性検索の結果、呼吸に関連するタンパク質NADH dehydrogenase complex IのサブユニットのひとつであるNuoM (ND4) と類似していた (図6)。NuoMは膜間におけるプロトン転移を担うと推定される13)。しかしながら、Slr1790はNuoMの約半分の長さであり、NuoM間で保存性の高い領域がSlr1790とは異なっており、Slr1790とNuoMの機能は異なるものと考えられた (図6)。

![]()
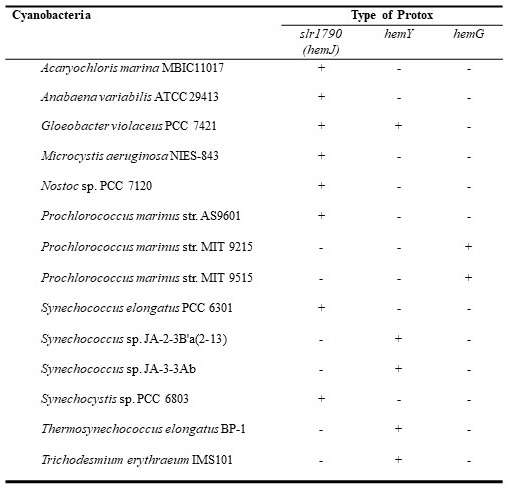
また、slr1790 (hemJ) ホモログの生物間分布をラン藻について調査した結果、ラン藻の多くはhemJホモログを持つことがわかった (図7、表3)。hemJホモログを持たないラン藻はhemG、あるいはhemYホモログを持ち、興味深いことに原始ラン藻であるGloeobacter violaceus PCC 7421 はhemJホモログとhemYホモログの両方を持っていた。ラン藻間においてhemJ、hemY、hemGの分布パターンがこのように排他的であることからも、それぞれが同じ機能を有し、hemJがProtoxをコードすることを支持する結果と考えられる。

![]()
表3 ラン藻におけるhemJ, hemY, hemGホモログの分布
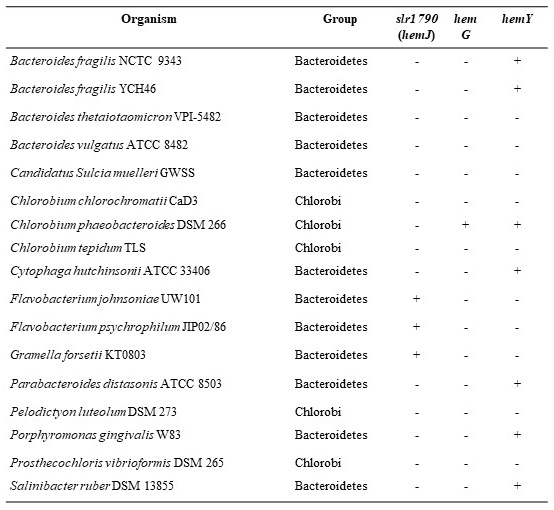
更に、ゲノム配列が公開されている全生物を対象に相同性検索を行った結果、hemJホモログはバクテロイデスやプロテオバクテリアに広く分布し、それらの多くはこれまでProtox遺伝子が不明の生物であった (図8)。γ-プロテオバクテリアを除くプロテオバクテリアにおいてはhemJホモログを持つ生物がhemYもしくはhemGホモログを持つものより多かった。対照的にクロロフレクサスはhemYホモログを持つものが多かった。γ-プロテオバクテリアの多くはhemGホモログを持ち、hemJホモログとhemGホモログは排他的に分布していた。同様にバクテロイデス/クロロビウムにおいては、hemJホモログとhemYホモログは排他的に分布していたが、このグループの中でChlorobium phaeobacteroides DSM 266はhemGホモログとhemYホモログを持っていた (表4)。解析したバクテロイデス/クロロビウムの17種類のうち7種類において、hemJ、hemY、hemGのいずれのホモログを持たなかったことより、少なくとも、あと₁種類は未同定のProtoxが存在するものと考えられた。
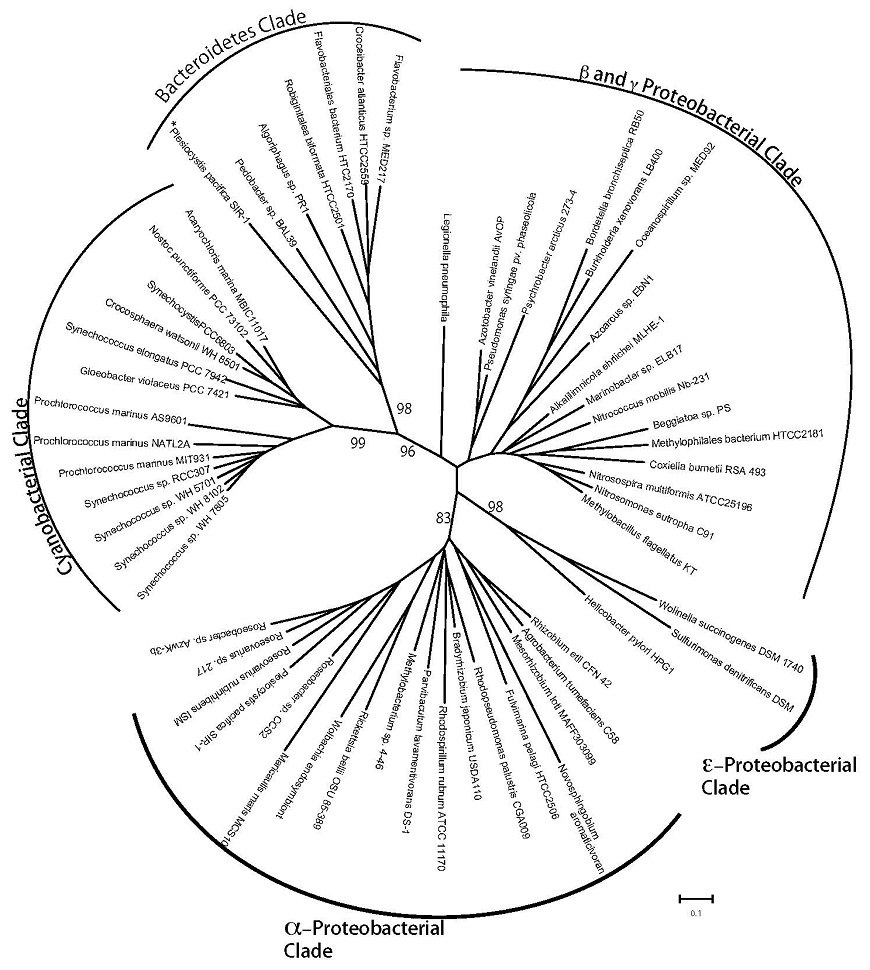
図8 hemJ(slr1790ホモログ)の系統樹解析
表4 バクテロイデス/クロロビウムにおけるhemJ, hemY, hemGホモログの分布
7.おわりに
遺伝学的手法と特異的阻害剤を組み合わせた手法により、不明であったラン藻Protox遺伝子 (hemJ) の同定に成功した。この探索手法は、適切な阻害剤があれば、バクテリアの代謝に関わる未知の遺伝子の同定に利用することが可能であると思われる。今回発見したhemJ遺伝子はラン藻だけでなくこれまでProtoxが不明であった多くの生物に分布していた。我々の解析では、さらにもう₁種類の未同定Protoxが存在することが予想される。Protoxのような生育に必須である遺伝子が、生物間においてこのように多くの種類を持つのか大変興味深い点であり、ヘムのように、すべての生物に必要な物質であっても、生物は、進化の段階で次々に新しい酵素を作り出して、よりよい方法でそれを合成する手段を模索していると考えられる。
また、hemJホモログは真核生物の持つhemYホモログとは全く構造が異なることから、例えばhemJを高等植物へ導入することによる除草剤耐性植物の創出や、hemJを標的酵素としたバクテリア剤の探索研究等、今後の産業面での展開にも期待できる。
謝辞
本研究は、低温科学研究所においては、田中歩教授、田中亮一准教授のもとで行われた。また、低温科学研究所の平島真澄氏 (現独立行政法人花き研究所)、田中佐知子氏、加藤由佳子氏のご協力に紙面をお借りして感謝したい。
文献
1) Mochizuki, N., Tanaka, R., Grimm, B., Masuda, T., Moulin, M., Smith, A. G., Tanaka, A., Terry, M. J.: Trends Plant Sci., 15, 488 (2010).
2) Oborník, M., Green, B. R.: Mol. Biol. Evol., 22, 234 (2005).
3) Koch, M., Breithaupt, C., Kiefersauer, R., Freigang, J., Huber, R., Messerschmidt, A.: EMBO J., 23, 1720 (2004).
4) Narita, S., Tanaka, R., Ito, T., Okada, K., Taketani, S., Inokuchi, H.: Gene, 182, 169 (1996).
5) Lermontova, I., Kruse, E., Mock, H. P., Grimm, B.: Proc. Natl. Acad. Sci. USA, 94, 8895 (1997).
6) Boynton, T. O., Daugherty, L. E., Dailey, T. A., Dailey, H. A.: Biochemistry, 48, 6705 (2009).
7) Xu, K., Delling, J., Elliott, T.: J. Bacteriol., 174, 3953 (1992).
8) Panek, H., O’Brian, M. R.: Microbiology, 148, 2273 (2002).
9) McFadden, G. I.: Plant Physiol., 125, 50 (2001).
10) Bhaya, D., Takahashi, A., Grossman, A. R.: Proc. Natl. Acad. Sci. USA, 98, 7540 (2001).
11) Hirai, K.: Peroxidizing Herbicides (Böger, P., Wakabayashi, K., eds.) , Springer-Verlag, 2, 15 (1999).
12) Gomelsky, M., Kaplan, S.: J. Bacteriol., 178, 1946 (1996).
13) Torres-Bacete, J., Nakamaru-Ogiso, E., Matsuno-Yagi, A., Yagi, T.: J. Biol. Chem., 282, 36914 (2007).