【トピックス】
クロロフィル生合成系において機能するニトロゲナーゼ類似酵素、暗所作動型プロトクロロフィリド還元酵素の構造と機能
野亦次郎
東工大・資源化学研究所
1.はじめに
クロロフィル (Chl)、バクテリオクロロフィル (Bchl) は光合成に必須のテトラピロール性色素であり、光エネルギーを化学エネルギーに変換する上で重要な機能を担う。従属栄養生物をはじめ、この地球上の多くの生物が光合成生物によって変換された太陽の光エネルギーに依存して生きていることを考えると、Chl、Bchlの生合成系は非常に重要な代謝系の1つであることがわかる。
Chl、Bchlの生合成経路は、15以上の酵素反応により構成される、極めて複雑な系である。この生合成系は、ビタミンB12 やヘムの生合成系と一部を共有している。出発物質の5-アミノレブリン酸からウロポルフィリノーゲンIIIまではビタミンB12 とシロヘムの、プロトポルフィリンIXまではプロトヘムの生合成系と共有している。プロトポルフィリンIXにMg2+が挿入される過程以降がChl、Bchlの生合成経路に特有であり、Mg-ブランチと呼ばれる1)。Mg-ブランチにおいて、プロトクロロフィリド (Pchlide) からクロロフィリドが生成する反応は、Chl、Bchl生合成の律速段階と言われる。多くの光合成生物において、クロロフィリドの生成反応は二つの異なる酵素、光依存型プロトクロロフィリド還元酵素 (LPOR; NADPH-Light-dependent Pchlide oxidoreductase)、または、暗所作動型プロトクロロフィリド還元酵素 (DPOR; Dark-operative Pchlide oxidoreductase) によって触媒される (図1)。LPORは短鎖デヒドロゲナーゼ/リダクターゼスーパーファミリーに属し、単一の蛋白質から構成される。反応にはNADPHおよび光を必要とするなど、ユニークな特徴を示す2)。一方、DPORは二つの蛋白質、L-蛋白質 (BchLホモ二量体) とNB-蛋白質 ((BchN)2(BchB)2ヘテロ四量体) から構成され、それぞれ窒素固定酵素ニトロゲナーゼのコンポーネント、Fe-蛋白質 (NifH ホモ二量体)、およびMoFe-蛋白質 ((NifD)2(NifK)2ヘテロ四量体) と構造的に類似していることが示されていた3)。筆者らは、光合成細菌を利用したDPORの大量発現系の構築をはじめ、嫌気チャンバーを活用し、DPORの生化学的解析を行う基盤を築いた。一連の解析によって、DPORの諸生化学的性質を明らかにし、DPORが構造的、機能的にもニトロゲナーゼと相同であることを明らかにした4,5,6)。最近私達は、DPORの触媒コンポーネントであるNB-蛋白質の結晶化を試み、基質Pchlideが結合した状態の蛋白質結晶を得ることに成功した。得られた構造情報に基づき生化学的解析を行い、ニトロゲナーゼとの構造的相違や、推定される反応機構について考察を行った。本稿では、この一連の解析について紹介したい。
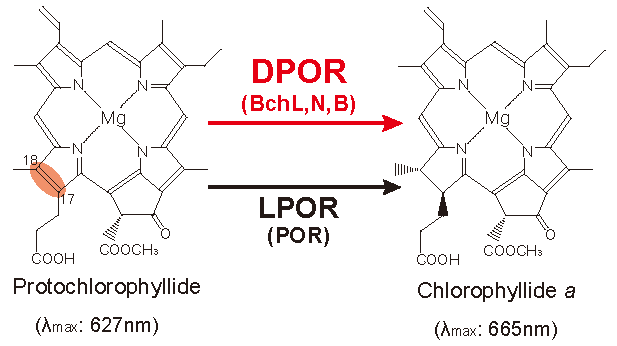
図1 暗所作動型プロトクロロフィリド還元酵素 (DPOR) または光依存型プロトクロロフィリド還元酵素 (LPOR) によるtrans還元反応
2.NB-タンパク質の結晶構造解析
結晶化に充分な量の精製蛋白質を効率よく得るために、紅色非硫黄細菌Rhodobacter capsulatusを用いて、NB-蛋白質の大量発現系を構築した6)。また、DPORはニトロゲナーゼ同様、酸素感受性酵素である。そこで、蛋白質を嫌気状態に保つため、全ての実験は嫌気チャンバーを用いて行った。光合成的に培養したR.capsulatusから精製したNB-蛋白質は緑色を呈しており、その吸収特性からPchlideを結合していることが確認された。この基質結合型のNB-蛋白質を用いて結晶化を行い、結晶構造を明らかにした7)(図2)。NB-蛋白質は (BchN)2(BchB)2からなるヘテロ四量体構造であり、これまで予想されていたモデルと一致した。各サブユニットの構成や全体構造はニトロゲナーゼの触媒コンポーネント、MoFe-蛋白質と非常に良く似ていた。その一方で、金属クラスターや触媒部位には大きな違いが見られた。本研究では、それらの詳細な解析を行った。
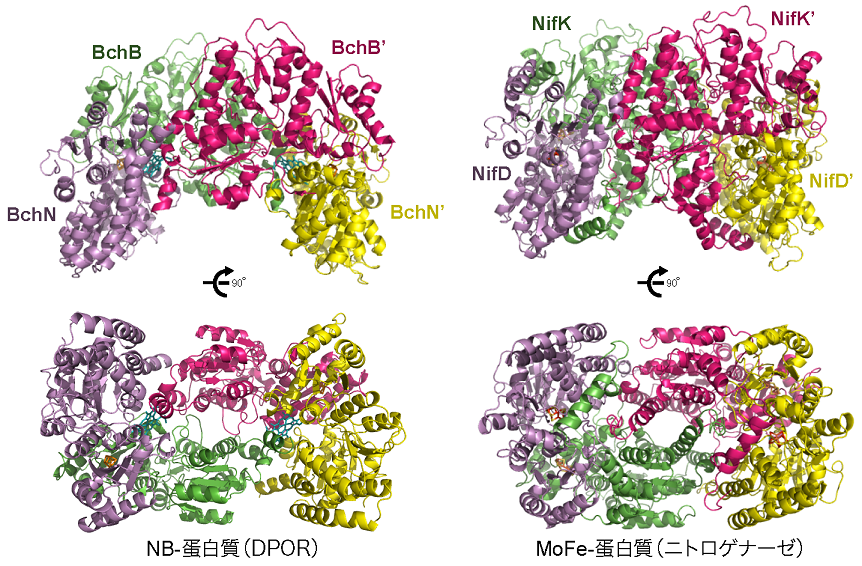
図2 NB-蛋白質とMoFe-蛋白質の結晶構造
3.鉄硫黄クラスターの解析
NB-蛋白質の2つの[4Fe-4S]型鉄硫黄クラスターはBchN-BchBの境界面に保持されていた (図3a)。これらの鉄硫黄クラスターはBchNに由来する3つのシステイン残基 (Cys26、Cys51、Cys112 ) と、BchBに由来する1つのアスパラギン酸残基 (Asp36) によって保持されるというユニークな特徴を持っており、私達はこれを“NB-cluster”と名付けた。NB-clusterのようにアスパラギン酸残基が鉄硫黄クラスターを配位している例は極めて珍しく、他には古細菌Pyrococcus furiosusとThermococcus profundusのフェレドキシンで報告されているだけである8,9)。このアスパラギン酸配位の重要性を調べるため、部位特異的変異導入によりAsp36をシステイン、アラニン、セリンへと置換した変異型NB-蛋白質 (それぞれD36C、D36A、D36S) を作製し、DPOR活性を測定した。その結果、D36CとD36Sは活性を失っていたが、D36Aは野生型NB-蛋白質と比較して13%の活性を保持していた (図4)。D36C、D36Aの結晶化を行ったところ、D36Cの鉄硫黄クラスターは4つのシステイン残基 (Cys26、Cys51、Cys112 とCys36) により保持されていた (図3b)。一方で、D36Aの鉄硫黄クラスターは3つのシステイン残基 (Cys26、Cys51、Cys112 ) によってのみ保持されており、配位子のない鉄原子には水分子または水酸化物イオンのいずれかが配位していた (図3c)。この構造は、活性型アコニターゼの鉄硫黄クラスターと非常に良く似ていた10)。以上の結果から、アスパラギン酸配位子は、NB-clusterの保持には必ずしも必要ないが、Pchlide還元活性には必要であることが明らかとなった。おそらく、Pchlideの還元に十分な、高い酸化還元電位を生み出す上で重要であると推察される。
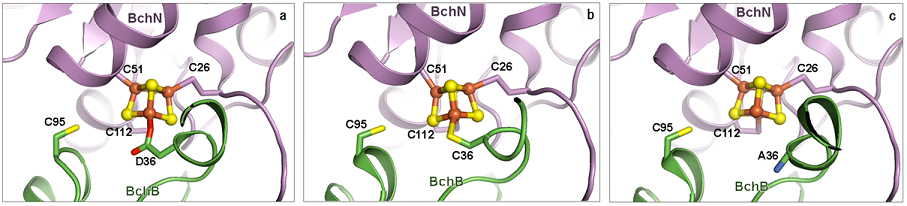
![]()
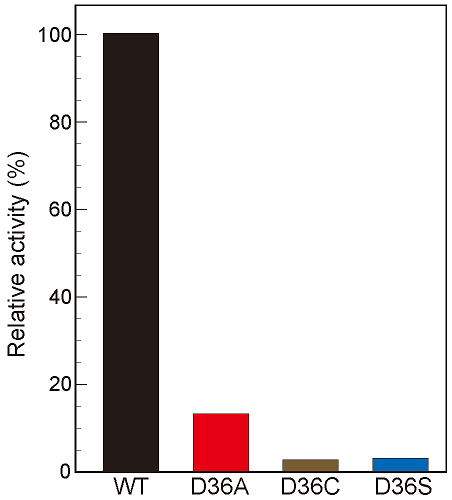
図4 野生型および変異型NB-蛋白質の活性評価
4.DPORによるPchlide還元反応機構の解析
NB-蛋白質の基質結合ポケットは、BchNとBchB、さらに、もう1つのプロトマーのBchB (BchB’) によって形成されている (図5a)。PchlideはNB-clusterから約10.0 Åの距離に位置し、PchlideとNB-clusterの間には疎水的な環境が形成され、その中間地点にはBchNに由来するフェニルアラニン残基 (Phe25) が配置している。基質を結合していない構造 (図5b) と比較すると、興味深い相違がある。基質の結合に伴い、BchB’ のヘリックス構造の一部がほどけ、ヘリックスが基質結合ポケットに“蓋”をするような配置へと変化している。この構造変化に伴い、ヘリックスを構成するロイシン残基 (Leu410) とメチオニン残基 (Met408) が基質から各々7.9 Å、7.6 Åの距離まで接近し、PchlideのC17側鎖のプロピオン酸が押し曲げられ、ポルフィリン平面に対しほぼ垂直な構造をとる。
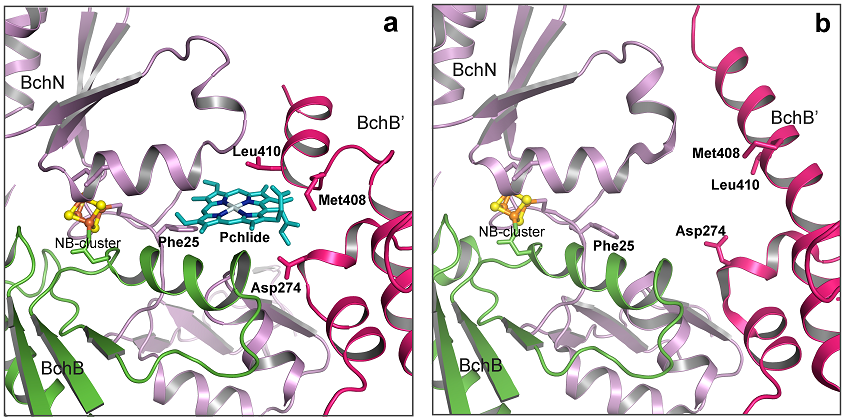
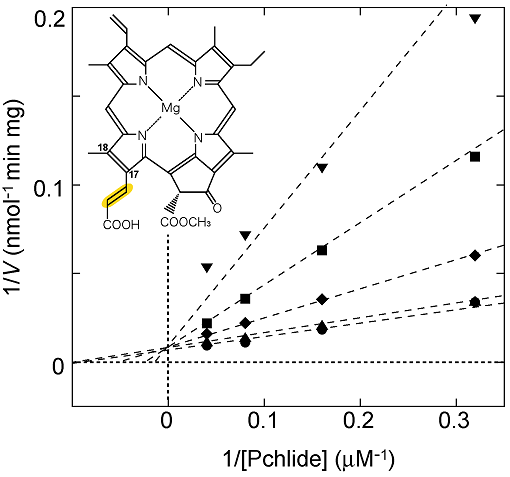
DPORによるPchlide還元反応では、2つの炭素原子、C17とC18間の炭素間二重結合が還元され、2つのプロトンがtrans位に付加される (図1)。このtrans還元を行うためには、2つのプロトンはポルフィリン平面に対し各々、C17には上方から、C18には下方から付加される必要がある。Pchlide近傍の構造を見ると、Pchlide のC17から4.9 Åの距離に、BchB’ を構成するアスパラギン酸残基 (Asp274) が位置しており、このAsp274がC17へのプロトンドナー候補と考えられた。そこで、部位特異的変異導入法によってAsp274をアラニンへと置換した変異型NB-蛋白質、D274Aを作製しDPOR活性を測定したところ、活性を失っていた (図6)。従って、Asp274がC17へのプロトンドナーであることが示唆された。一方、C18の近傍には極性のアミノ酸残基は見あたらない。ここでPchlide分子の構造に注目すると、C17側鎖のプロピオン酸が大きく湾曲し、そのカルボキシル基はC18から4.8 Åの近距離に位置している。そこで、このプロピオン酸をC18へのプロトンドナー候補と考え、検討を行った。Pchlideのアナログ基質であるクロロフィルc (Chl c) は、プロピオン酸に代わりアクリル酸をC17側鎖に持つ。そこで、Chl cをDPOR反応に供したところ、還元されなかった。また、Chl cはDPORによるPchlide還元反応を競合的に阻害した (図7)。これらの結果は、Chl cがPchlide同様に基質結合ポケットに結合するが、還元はされないことを意味する。従って、PchlideのC17側鎖のプロピオン酸がC18へのプロトンドナーであることが示唆された。さらに、DPOR反応におけるPhe25とMet408、Leu410の役割を検討した。部位特異的変異導入法によりPhe25、Met408、Leu410を各々アラニンへと置換した変異型NB-蛋白質、F25A、M408AおよびL410Aを作製し、DPOR活性を測定した (図6)。F25Aは50%の活性を示し、電子伝達に適した疎水的環境の形成に関わると推測された。一方、M408Aは80%以上の活性を維持していたが、L410Aは活性を失っていたことから、Leu410はDPOR活性に大きく寄与していることが示唆された。おそらくLeu410はC17側鎖のプロピオン酸の湾曲を引き起こすと考えられる。
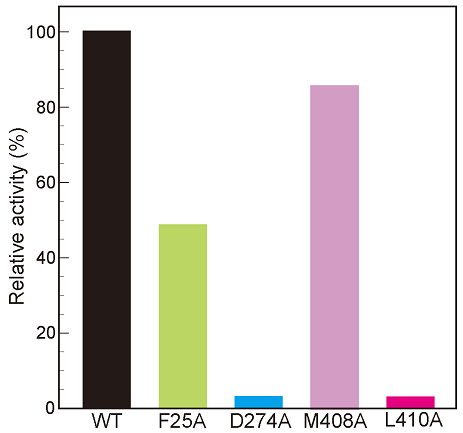
図6 野生型および変異型NB-蛋白質の活性評価

以上の結果から、DPORによるtrans還元の反応機構を推定した (図8)。まずPchlideの結合に伴いBchB’ヘリックスがPchlideへ接近し、Leu410がプロピオン酸の湾曲を引き起こす。次に、電子がL-蛋白質からNBclusterを介してPchlideのπ電子雲へと伝達され、Asp274がC17へ、プロピオン酸がC18へ各々プロトンを付加し、カチオンラジカルが生じる。続いて2つめの電子が伝達され、クロロフィリドが生成する。
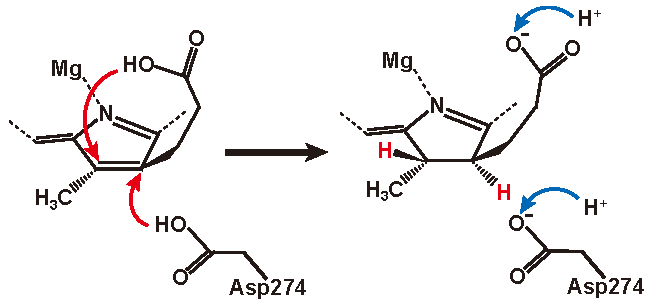
図8 推定されるtrans還元反応機構
5.DPORとニトロゲナーゼに保存される共通構造
興味深いことに、NB-蛋白質におけるNB-clusterとPchlideの位置は各々、MoFe-蛋白質におけるP-clusterと鉄モリブデンコファクター (FeMo-co) の位置と非常によく一致し、DPORとニトロゲナーゼの間には全体構造のみならず触媒要素の配置までが保存されていることが明らかになった。この保存された共通構造のもと、電子は還元蛋白質 (L-蛋白質、Fe-蛋白質) の鉄硫黄クラスターから触媒蛋白質 (NB-蛋白質、MoFe-蛋白質) の鉄硫黄クラスター (NB-cluster、P-cluster) へ、ATPの加水分解に依存して伝達される。さらに電子は触媒蛋白質の基質結合ポケットへ伝達され、触媒部位に結合した基質 (Pchlide、FeMoco-N2) の多重結合が還元される (図9)。一次構造の類似性から、DPORとニトロゲナーゼは進化的起源を同じくしており、共通の祖先酵素から分岐したと考えられている11)。DPORはポルフィリンの還元を、ニトロゲナーゼはN2の還元を行うために各々が独自の金属クラスター (NB-cluster、P-cluster) や触媒ポケットを持つように進化を遂げてきたと考えられる。しかし、その進化の中でも、保存された共通構造はポルフィリンやN2の非常に安定な多重結合を還元する上で不可欠であり、共通の祖先酵素から両酵素に引き継がれ、保持され続けてきたのかもしれない。
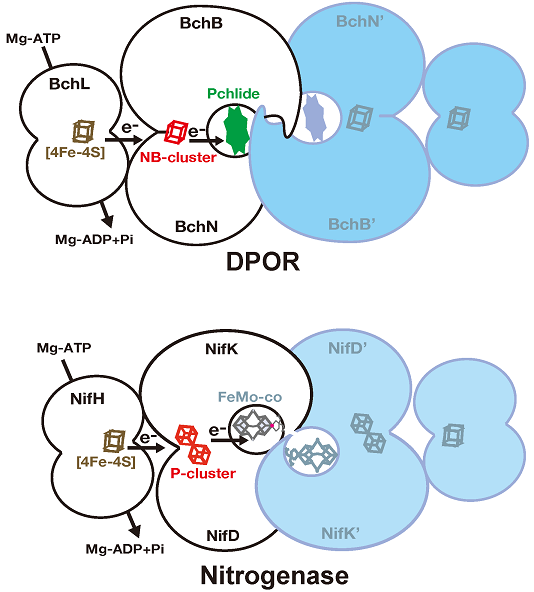
図9 DPORとニトロゲナーゼに共通した構造と反応機構
6.おわりに
本稿では、クロロフィル生合成における鍵酵素、DPORの触媒蛋白質の結晶構造解析からその反応機構の提唱に至るまでの、一連の解析を紹介した。DPORによるtrans還元反応の構造的基盤が明らかとなり、DPORとニトロゲナーゼが保持する共通構造が見出された。そしてこの構造が安定な多重結合の還元に必須であると推察された。
最近、メタン生成細菌や紅色非硫黄細菌を含む幾つかの窒素固定細菌のゲノム中に、DPORやニトロゲナーゼに類似した遺伝子が複数見つかり、注目を集めている12,13)。しかし、これらのニトロゲナーゼ類似遺伝子がどのような機能を担っているのか、明らかにされていない。これらニトロゲナーゼ類似酵素の機能や構造、反応機構を明らかにすることは、ニトロゲナーゼ類似酵素群がどのように進化してきたのか、光合成 (クロロフィル合成) や窒素サイクルをはじめとする、生物の重要な代謝系の誕生にどのように関わってきたのかを解き明かす手がかりを与えると期待される。興味は尽きることが無い。
謝辞
本研究は名古屋大学生命農学研究科の藤田祐一准教授のもとで行なわれたものである。蛋白質の結晶化は大阪大学蛋白質研究所の栗栖源嗣教授、村木則文博士との共同研究により行なわれた。また、クロロフィルcは立命館大学の民秋均教授、溝口正教授にご提供頂いた。本研究は、日本学術振興会および科学技術振興機構のサポ−トを得て行なわれたものである。
文献
1) Fujita, Y.: Chlorophylls, in Encyclopedia of life science, 4, 324 (2002).
2) Wilks, H., Timko, M.: Proc. Natl. Acad. Sci., 92, 724 (1995).
3) Fujita, Y., Bauer, C. E.: J. Biol. Chem., 275, 23583 (2000).
4) Nomata, J., Swem, L. R., Bauer, C. E., Fujita, Y.: Biochim. Biophys. Acta, 1708, 229 (2005).
5) Nomata, J., Kitashima, M., Inoue, K., Fujita, Y.: FEBS Lett., 580, 6151 (2006).
6) Nomata, J., Ogawa, T., Kitashima, M., Inoue, K., Fujita, Y.: FEBS Lett., 582, 1346 (2008).
7) Muraki, N., Nomata, J., Ebata, K., Mizoguchi, T., Shiba, T., Tamiaki, H., Kurisu, G., Fujita, Y.: Nature, 465, 110 (2010).
8) Calzolai, L., Gorst, C., Zhao, Z., Teng, Q., Adams, M., La Mar, G.: Biochemistry, 34, 11373 (1995).
9) Imai, T., Taguchi, K., Ogawara, Y., Ohmori, D., Yamakura, F., Ikezawa, H., Urushiyama, A.: J. Biochem., 130, 649 (2001).
10) Beinert, H., Kennedy, M., Stout, C.: Chem. Rev., 96, 2335 (1996).
11) Fujita, Y., Bauer, C. E.: Porphyrin Handbook, 13, 109 (2003).
12) Raymond, J., Siefert, J. L., Staples, C. R., Blankenship, R. E.: Mol. Biol. Evol., 21, 541 (2004).
13) Staples, C. R., Lahiri, S., Raymond, J., Herbulis, V. L., Mukhophadhyay, B., Blankenship, R. E.: J. Bacteriol., 189, 7392 (2007).