【トピックス】
相同DNA組換えの機構理解から新規蛋白質創出へ
柴田武彦、美川 務
理研基幹研究所、放射光科学総合研究センター
1.はじめに
遺伝学で「乗換え」を学んだことがありますか。これが、我々が「交叉、crossing-over」と呼ぶ相同 (DNA) 組換えの一つの型である。性決定因子と目の色を決定する遺伝子などの連鎖が見つかり、それらの遺伝子の間で起きる組換え現象を現わすために、約百年前T. H. Morgan1) が初めて用いた。Morganらは、交配で観察される交叉の率で遺伝子の間の距離を現わすと、遺伝子が一直線上に並ぶことを見つけた。変異遺伝子間の距離を交叉率1%を単位とするcM (センチモルガン) で現わす遺伝子地図 (連鎖地図) の作成は、前ゲノム時代には個々の遺伝子を突きとめる手段であった。この四半世紀の研究で相同組換えの分子レベルでの仕組みがかなり分かり、近年は、その知識をもとに酵素工学分野でも役立つ強力な新技術が実現してきた。
2.相同組換え
2-1 DNA二重鎖切断修復と相同 (DNA) 組換え (相同組換え)
ウイルスを除く全生物のゲノム情報はA, T, G, Cの4種の塩基を文字として二重鎖DNAに記録され子孫に伝えられる。Aは必ずTと、Gは必ずCと水素結合で対になるため、一方のDNA鎖の塩基配列が決まると相手のDNA鎖の塩基配列も一つに決まる。よって、互いに相補配列と呼ばれる構造を持つ二重鎖DNAは同じゲノム情報を2重に記録しており、活性酸素種 (ROS) などによってDNAが傷ついても、また複製エラーが起きても、相補鎖の配列を鋳型として正確に修復される。二重鎖DNAが膨大なゲノム情報を誤りなく遺伝できるのは、このためである。ところが、二重鎖DNAの2つのDNA鎖が同時に切られる二重鎖切断もかなりの頻度で起きる (図1)。高等動植物の細胞では、ゲノムDNAのごく一部しか遺伝子として使わないため、両切断末端をそのまま再結合する非相同末端接合 (non-homologous end joining, NHEJ) とよばれる修復もあるが、必ずその部分で配列の欠落や挿入を伴いもと通りにはならない (図1)。切断末端と同じ塩基配列をもつ二重鎖DNAを鋳型として、そのコピーで二重鎖切断の周辺を置き換えることでDNA二重鎖切断を正確に修復するのが相同組換えによる二重鎖切断修復である。
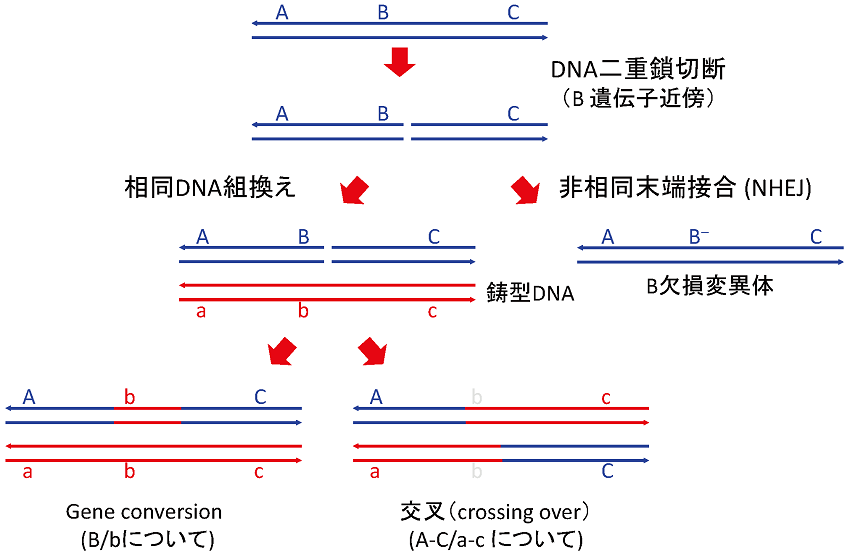
図1 DNA二重鎖切断の修復
その仕組みを遺伝子A-B-Cを載せたDNAの遺伝子B近傍に起きた二重鎖切断の修復を例に説明したい。先ず、二重鎖切断末端で5’端から一方のDNA鎖が削り込まれて3’突出の単鎖DNA領域ができる (図2;第2段階)。その単鎖DNAに相同DNA対合蛋白質が働き、鋳型となる二重鎖DNA (組換えが観察できるようにA-B-Cの対立遺伝子*1 a-b-cを載せているとする) の中の相補配列を探し出し、その単鎖DNAを割り込ませ、A-TまたはG-Cの塩基対合によりヘテロ二重鎖を作る (図2;第3段階)。この反応を特に相同 (DNA) 対合という2,3)。割り込んだ単鎖DNAの3’端がプライマーとして始まる修復DNA合成で鋳型DNAの遺伝子bを含む塩基配列をコピーして切断で失われたDNA配列を回復する (図2; 第4段階)。
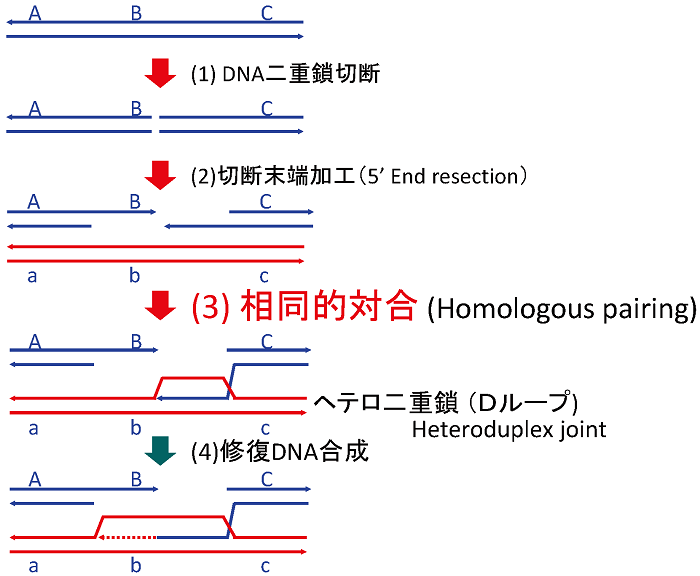
図2 相同DNA組換えの初期過程:DNA二重鎖切断からヘテロ二重鎖形成、修復DNA合成まで
回復したDNA配列をもつ単鎖DNA部分が鋳型から離れて、切断の相手端にできた単鎖DNA部分の相補配列とアニールすると切断の両側の連続性を回復する (図3;左側第5段階)。残った単鎖ギャップが第2の修復DNA合成で埋められるとDNA二重鎖切断修復が完了する (図3;左側第6段階)。鋳型DNAをコピーした部分が対立遺伝子bなのでA-TまたはG-Cではない不整対 (mismatch) が残るが、それが対立遺伝子bの配列に従って修復される (図3;左側第7段階) と鋳型DNAの遺伝情報が組み込まれた組換え体 (A-b-C) として識別される。このように一対の親DNA (A-B-C : a-b-c) の一方だけが相手の遺伝情報で置き換えられる組換え (A-b-C : a-b-c) をgene conversion*2という (図1)。一対の親DNAの間でDNA部分を交換する (A-C : a-c →A-c: a-C) 交叉 (crossing over; 図1) は、ホリデー中間体を経て行われる (図3;右側段階5から7)4,5)。
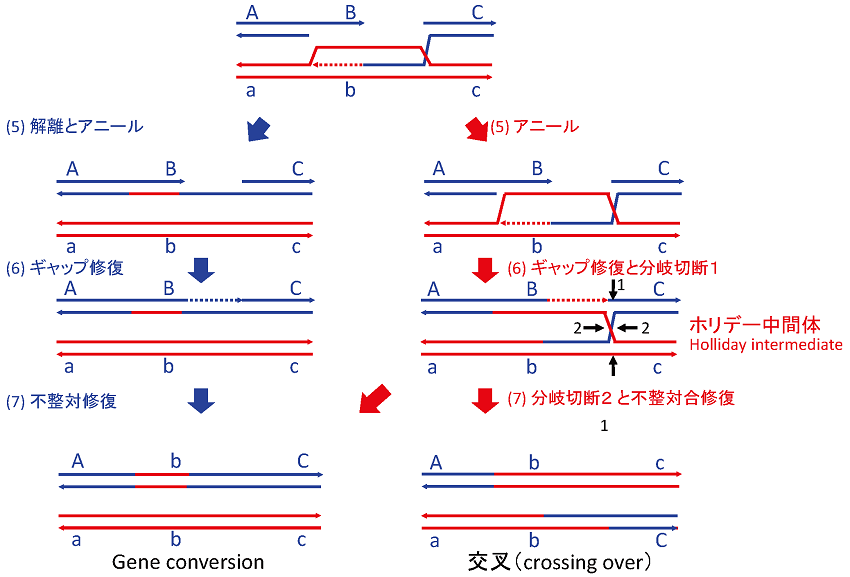
図3 相同DNA組換えの後期課程:gene conversionと交叉
相同組換えはランダムに起きる傷としての二重鎖切断の修復に働く他、半数体の配偶子細胞ができる過程で起きる減数分裂では相同染色体の分離に欠かせない。これは、交叉によってできるキアズマの働きである。減数分裂期の組換えは、DNA上の決まった場所に決まったタイミングで起きるプログラムされた二重鎖切断で誘導される。ゲノム上の指定した部位で相同組換えを誘導させる標的組換え技術の実現には、二重鎖切断の制御が重要な手掛かりだ。
*1. 対立遺伝子:同一の遺伝子であるが、機能的に区別できるDNA配列の違い (遺伝子型) をもつ遺伝子群を対立遺伝子 (allele) という。
*2. gene conversionの和訳として「遺伝子変換」という用語が有るが、むしろ別の意味に広く使われているので、本稿ではgene conversionを用いる。
2-2 相同組換えに働く2種の相同対合蛋白質
ATP存在下で大腸菌のRecA蛋白質が単鎖DNAに働いて相補配列をもつ二重鎖DNAと相同対合させることが発見されて2,3) から相同組換えの分子レベルでの理解が急速に進んだ。RecAと構造と機能がよく似たATPを補助因子として相同対合とDNA鎖交換反応を行う蛋白質が、真正細菌 (RecA)、古細菌 (RadA)6)、真核生物 (Rad51, Dmc1)7,8) から次々に見つかった。相同対合自身にはATP分解は不要だが、DNA鎖交換反応はATP分解に依存するヘテロ二重鎖伸長として見つかった。
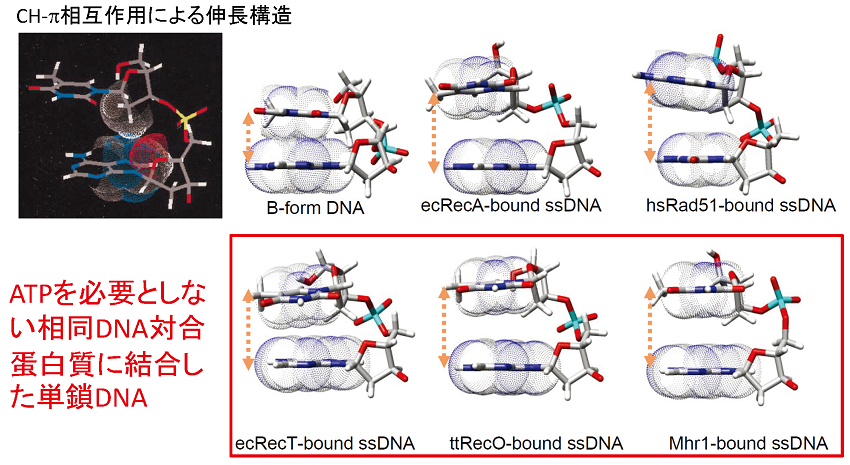
胡桃坂らや凌らはATPを必要としないRad52 (ヒト)、Mhr1 (酵母ミトコンドリア) など一群の相同対合蛋白を明らかにした9,10)。Rad52は、酵母では相同組換えに欠かせない蛋白質で、組換えメディエーターの原型である11)。単鎖DNA部分ができると、細胞内に豊富にある単鎖DNA結合蛋白質 (真正細菌のSSB、真核生物のRPA) が直ちに結合しRecA/Rad51が相同対合することを妨害する。この状況でRecA/Rad51を単鎖DNAに載せるのが、組換えメディエーターである11)。興味深いことに、RecA/Rad51を用いる相同組換え系は全てATP不要型相同対合蛋白質を組換えメディエーターとして必要とする。ATPを必要とするRecA/Rad51による相同対合とATPを必要としないRad52やMhr1による相同対合は、化学反応として共通であるかが議論になったが、最近、我々が特殊なDNAの立体構造が相同対合反応共通な中間体であることを示したことで、DNAから見たときにはATPの要求性に関わりなく相同対合反応は共通機構で起きるといえる12)。一方、蛋白質側からみると異なる特徴が見えてくる。Mhr1による相同対合反応産物を精査したところ、それがDループではなく、ワトソン・クリック型塩基対によるヘテロ二重鎖を含む並行三重鎖13) であることが示された14)。並行三重鎖からDNA鎖交換反応で第3のDNA鎖が分離するとDループになる。
3.相同対合蛋白質を活用したノイズフリーPCR
真正細菌や真核生物の核での相同対合は全てRecA/Rad51を必要とするが、ミトコンドリア (ヒト 16 kbp;出芽酵母 75-85 kbp) やDNAウイルスでは、RecA/Rad51型蛋白質を必要とせず、ATP不要型相同対合蛋白質だけで相同組換えが起きる例が知られている。これらは共通にゲノムサイズが小さいことを考慮すると、真正細菌 (4 Mbp) 以上の大きなゲノムDNAでは、ATP不要型相同対合蛋白質が先ず荒く相補塩基配列を探し出し、RecA/Rad51が、ATP分解によるエネルギーを利用した反応で不整対をもつヘテロ二重鎖を解離することで、高精度相同対合を行っているのだろう。以下の2つのRecAの機能はこの仮説を支持する。先ず、は相同領域長が十分に無い (数kb以下) とATP分解に依存した反応で、できたDループを解離する (Dループサイクル)15)。また、PCRによってDNAを増幅する時に、反応系へ熱耐性RecAを添加するとそのATP分解に依存した活性で非特異的副産物生成が効果的に抑制できる (図4)16,17)。
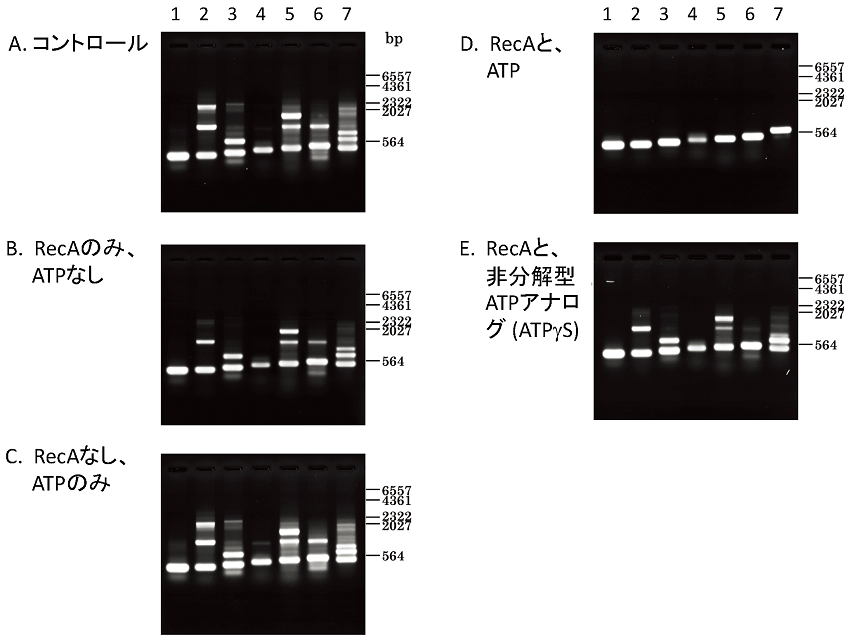
図4 RecAによるPCR副産物の効果的な抑制
4.相同組換えによる酵素工学の思わぬ可能性
4-1 DNAゲノムとRNAゲノムと相同組換え
先に述べた相同対合反応の中間体として共通に誘導される特殊なDNAの立体構造とは、各ヌクレオチドのDNAを特徴づけるリボース環2’位のメチレン基と、その次位の塩基の間で起きるCH-π相互作用によって塩基間が引き延ばされ塩基間スタッキングがない構造のことである。そのため二重鎖DNAであっても塩基の自由回転で相手の単鎖DNAとの相補配列を探しだせる18)。この伸長構造は、NMR技法で解かれた (図5)12,19)。CH-π相互作用によって安定化される伸長構造が相同対合の中間体ということは、2’OHの存在でこの構造をとれないRNAは、大容量のゲノム情報保持に必要な二重鎖構造をとると相同組換えができないことを意味する。事実、RNAゲノムをもつウイルスでは相同組換えは、突然変異や非相同組換えに比べても稀な現象であり、鋳型スイッチ (template switch) とよばれる単鎖DNA同士のアニーリングを基礎にした反応でしか起きない。突然変異や非相同組換えに比べはるかに高頻度で、普遍的に相同対合を基に相同組換えを行うDNAゲノムとは対照的である。ダーウインの進化論以来、伝統的に突然変異が新規遺伝子創出の原動力とみなされている。しかしながら、変異率はRNAの方がDNAよりもはるかに高いにもかかわらず、ゲノム情報の複雑さでは、RNAゲノムはDNAゲノムの足元にも及ばない。さらに、ミトコンドリアゲノムは核ゲノムより10倍も高い率で突然変異を起こす一方で、ヒトと酵母のミトコンドリアにコードされた蛋白質を比較すると、まるで進化していないことが分かる。ミトコンドリアゲノムは二重鎖DNAであるが、厳密な母系遺伝やホモプラスミー*3により相同組換えが起きても痕跡が残らない。これらの例は、突然変異というよりも,むしろ相同組換えの方が新規の遺伝子創出の強力な手段であることの反映ともみなせる20)。
*3. ホモプラスミー:それぞれの細胞で数百から数千コピーも存在するミトコンドリアDNAが単一の遺伝子型を保っている状態。変異等で生じた対立形質は、栄養増殖過程で急速に分離する。

4-2 DNAシャッフリングという分子進化工学
ゲノム情報は文字列なので文章と見做せる。作文は、実際には頭の中で既知の文章の部分をつなぎ合わせたり、単語や文節単位の入れ替えで行っている。塩基配列が共通な個所があれば異なる遺伝子間のgeneconversion型相同組換えで遺伝子部分を取り換えるだけ、染色体異常を伴わずに効率よく新規遺伝子を創出できる、言い換えると新遺伝子機能を作文できるはずである。相同組換えはある程度の長さの塩基配列の共通性を糊代にしているので、蛋白質をコードしているDNA領域はやはり組換え相手に蛋白質をコードしている領域を、しかもアミノ酸を規定する3文字単位のフレームを合わせて選ぶ。この点で文法無視で並べ替える非相同組換えと違う。この仮説は、StemmerらのDNA シャッフリング 21) によって部分的には支持される。二重鎖DNAにランダムに単鎖切断を入れてから変性して、プライマーを加えずに、エラーが起こりやすい条件でPCRを行うと単にエラーが起こりやすい条件でのPCRに比べてはるかに効果的に蛋白質の改質ができた。DNAシャッフリングでは、単鎖DNA同士のアニーリングでDNA部分の編集を行っているので、RNAでもできる相同組換えの範囲内であり、二重鎖DNAでの組換えに秘められているメリットは生かされていない。
4-3 相同組換え活用による新規天然型抗体生産
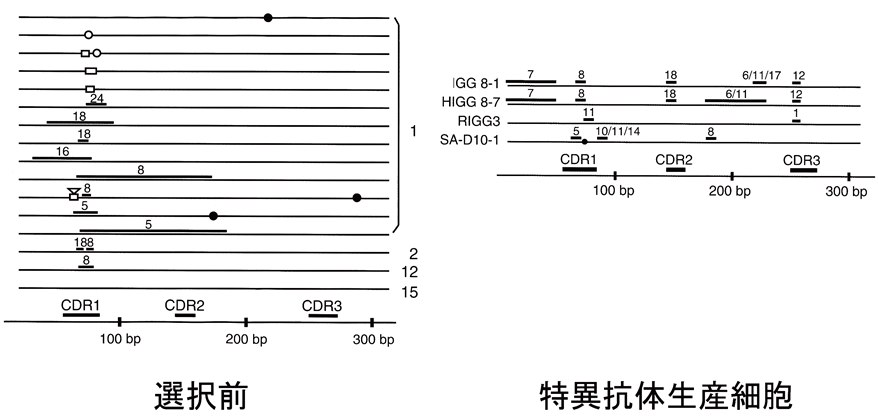
相同組換えによる新機能をもつ蛋白質創出への第一歩がADLib法の発明である。相同組換え率は、高い減数分裂期酵母細胞でも10-2レベル以下であり、高等動物細胞の相同組換えはそれよりはるかに低い。酵母の相同組換え開始制御の仕組みについての研究から、組換え開始に先立って、二重鎖切断が起きるDNA部位でヒストンのアセチル化が亢進して染色体が再編成されることが分った22)。瀬尾と太田らは、奇想天外な発展として、ニワトリのB細胞由来株化細胞をヒストン脱アセチル化酵素阻害剤で処理してみた。驚いたことに、核細胞当たり、ほぼ100%という超高頻度で免疫抗体遺伝子可変領域の相同組換えを誘導できた。さらに、抗原を結合させた磁気ビーズによって、その抗原に特異的に結合する抗体を生産する細胞を、組換え誘導処理細胞群の中から釣り上げ、1週間程度でクローン化とその抗体の多量生産ができた (ADLib法: http://www.jst.go.jp/pr/announce/20050530/index.html)23,24)。動物体を使わないこの手法は自己抗原など異物と認識されにくい困難抗原の抗体を取得する実績も挙げている。トリは、ヒトと異なり可変領域のカセットが軽鎖、重鎖それぞれに一つしかなく、代わりに可変領域に似た塩基配列を数十も含むクラスターがありそれを鋳型とするgene conversion型相同組換えによって多様な抗原特異性を獲得するといわれていた。ところが、実際に相同組換えの誘導で新規の特異性をもつ抗体を作らせたのは、これが初めてである。ヒストン脱アセチル化酵素阻害剤処理では相同組換えと共に、突然変異も同程度に誘導されるが、特異抗体を生産する細胞の可変領域はほぼ全て相同組換えでできており、突然変異の寄与はほとんどなかった (図6)23)。この相同組換えは、ヘテロ二重鎖に10%程度の不整対を含む必要があり、RecA/Rad51型蛋白質による不整対排除機能は抑制されていると考えられる。ADLib法の原理は抗体を超えて広く新規蛋白質創出に応用できるであろう。

5.おわりに:試験管内相同組換え系構築に向けて
知られている蛋白質の限界は生物進化での選択の結果であり必ずしも蛋白質の物性に根差したものではない。生体の制約を外れて思いのままの形や機能を持った蛋白質の創出が可能になれば、多様なナノスケールの構造物、様々な蛋白質触媒 (酵素) の創出が実現する。その技術は世の中を一変するほどのインパクトを持つであろう。現時点では機能をもつ蛋白質を一からデザインすることは極めて困難であるが、既知の機能を持った構造ユニットを組合せることにより、新たな機能を持った蛋白質を創出することはより実現性が高いであろう。事実、生命は様々な遺伝子を組合せることにより複雑な構造と機能を持った新しい蛋白質を創出して進化してきた。それを実現してきたのが相同組換えなのである。もし、試験管内で相同組換えを高度に制御して利用できるようになれば、人工的に新規蛋白質を創出する基盤技術の確立につながる。さらに、生命が行うのと同じ相同組換えで行う新規遺伝子創出は、他の方法を圧倒する可能性も高い。今後、相同組換えに参加する数多くの蛋白質を試験管内で再構築することにより試験管内相同組換え系の構築を目指していきたい。
文献
1) Morgan, T. H., Cattell, E.: J. Exp. Zool., 13, 79 (1912).
2) Shibata, T., DasGupta, C., Cunningham, R. P., Radding, C. M.: Proc. Natl. Acad. Sci. USA, 76, 1638 (1979).
3) McEntee, K., Weinstock, G. M., Lehman, I. R.: Proc. Natl. Acad. Sci. USA, 76, 2615 (1979).
4) Holliday, R.: Genetic Res., Camb., 5, 282 (1964).
5) Szostak, J. W., Orr-Weaver, T. L., Rothstein, R. J., Stahl, F. W.: Cell, 33, 25 (1983).
6) Sandler, S. J., Satin, L. H., Samra, H. S., Clark, A. J.: Nucleic Acids Res., 24, 2125 (1996).
7) Shinohara, A., Ogawa, H., Ogawa, T.: Cell, 69, 457 (1992).
8) Bishop, D. K., Park, D., Xu, L. Z., Kleckner, N.: Cell, 69, 439 (1992).
9) Kagawa, W., Kurumizaka, H., Ikawa, S., Yokoyama, S., Shibata, T.: J. Biol. Chem., 276, 35201 (2001).
10) Ling, F., Shibata, T.: EMBO J, 21, 4730 (2002).
11) Sung, P.: J. Biol. Chem., 272, 28194 (1997).
12) Masuda, T., Ito, Y., Terada, T., Shibata, T., Mikawa, T.: J. Biol. Chem., 284, 30230 (2009).
13) Vlieghe, D., Vanmeervelt, L., Dautant, A., Gallois, B., Precigoux, G., Kennard, O.: Science, 273, 1702 (1996).
14) Ling, F., Yoshida, M., Shibata, T.: J. Biol. Chem., 284, 9341 (2009).
15) Shibata, T., Ohtani, T., Iwabuchi, M., Ando, T.: J. Biol. Chem., 257, 13981 (1982).
16) Shigemori, Y., Mikawa, T., Shibata, T., Oishi, M.: Nucleic Acids Res., 33, e126 (2005).
17) 美川 務: BIO INDUSTRY, 26, 58 (2009).
18) Nishinaka, T., Shinohara, A., Ito, Y., Yokoyama, S., Shibata, T.: Proc. Natl. Acad. Sci. USA, 95, 11071 (1998).
19) Nishinaka, T., Ito, Y., Yokoyama, S., Shibata, T.: Proc. Natl. Acad. Sci. USA, 94, 6623 (1997).
20) Shibata, T., Nishinaka, T., Mikawa, T., Aihara, H., Kurumizaka, H., Yokoyama, S., Ito, Y.: Proc. Natl. Acad. Sci. USA, 98, 8425 (2001).
21) Stemmer, W. P. C.: Nature, 370, 389 (1994).
22) Yamada, T., Mizuno, K.-i., Hirota, K., Kon, N., Wahls, W. P., Hartsuiker, E., Murofushi, H., Shibata, T., Ohta, K.: EMBO J, 23, 1792 (2004).
23) Seo, H., Masuoka, M., Murofushi, H., Takeda, S., Shibata, T., Ohta, K.: Nat. Biotechnol, 23, 731 (2005).
24) 太田 邦史, 瀬尾 秀宗: Yakugaku Zasshi, 127, 81 (2007).