【トピックス】
L-ノルバリンの製法開発
林 素子、山本浩明
ダイセル化学
1.はじめに
EVAL (N-[1-(S)-(ethoxycarbonyl)butyl]-(S)-alanine) は、ACE阻害剤であるPerindoprilの重要中間体であり、非天然アミノ酸であるL-ノルバリン (L-Nva、(S)-2-aminopentanoic acid) から合成される。アミノ酸は、キラルシントンとして古くから用いられてきた。1,2) これは、アミノ酸が天然に大量に存在する光学活性化合物であり、容易に入手可能であることが理由である。しかし、非天然アミノ酸は、通常の代謝経路を用いた発酵法やタンパク質の加水分解物の精製で得ることは、ヒドロキシプロリンなど一部の例外を除いて困難である。そこで、EVALの原料たるL-Nvaの製法開発を行った。
光学活性な非天然アミノ酸を合成するには、いくつかの方法が考えられる。中でも、対応するα-ケト酸のアミノ化が容易に考えうるが、L-Nvaの原料であるα-ケト吉草酸 (KVA、2-oxopentanoic acid) は経済性、安定性に問題があり、且つ工業的なスケールで入手が困難であった。それに対してラセミのNvaはストレッカー法で容易に合成でき、比較的安価に必要量を入手できたため、ラセミ体を原料として使用することにした。
ラセミ体を原料とした光学活性L-アミノ酸の合成には、ラセミのN-アセチルアミノ酸をアシラーゼ等で光学分割する方法が広く用いられている。しかし、この方法は必要な立体と不要な立体を分割するため、理論収率は 50%にとどまる。収率を改善するには、不要な立体のラセミ化、再反応が必要であり、多工程の反応となってしまう。それに対して、不要な立体を必要な立体に反転させるデラセミ化は、ラセミ体を原料としながらも理論収率が100%となり、コスト面からも資源の有効活用という面からも好ましいルートと考えられたことから、デラセミ化ルートの検討を開始した。
アミノ酸のデラセミ化3,4) は、①不要なD体をケト酸に酸化、②生成したケト酸をアミノ化、という2つの工程からなる (図1)。その過程で原料に含まれるL体はそのまま残存するため、最終的に理論収率は100%となる。この2つの工程を、当社の光学活性化合物を得る主要手段のひとつである組換え酵素ライブラリー5) (Cloned EnzymeLibrary) を用いて実施することとした。この際、培養及び反応工程を1つの反応器で行えるよう、必要な酵素遺伝子を1種類の組換え菌で全て発現する、ワンポットデラセミ化を目標に、検討を開始した。
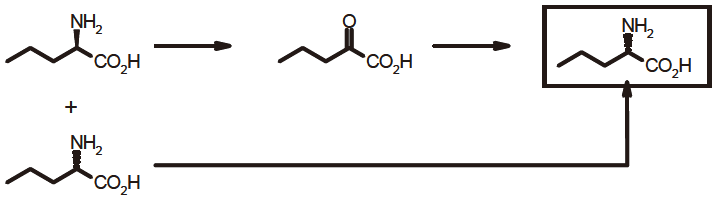
図1 デラセミ化によるノルバリンの合成
2.酵素スクリーニング
2-1 D-アミノ酸酸化酵素のスクリーニング
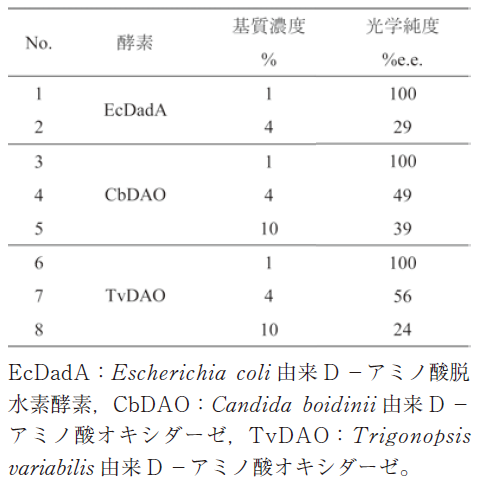
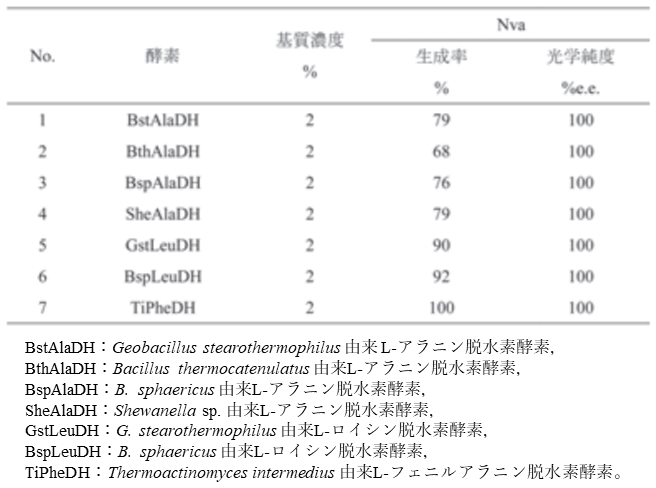
まず、各工程に必要な酵素のスクリーニングを行った。D体をケト酸に酸化する酵素のスクリーニングは、2種類のD-アミノ酸オキシダーゼと1種類のD-アミノ酸脱水素酵素を用い、ラセミのNvaを基質として反応することによって行った。また、反応は、酵素遺伝子を組み込んだプラスミドによって形質転換した大腸菌を用いて実施した。反応の進行に伴い、D体が消失して光学純度が上昇するため、光学純度をスクリーニングの指標とした。その結果、酵母の一種であるCandida boidinii由来のD-アミノ酸オキシダーゼ (CbDAO)6) が高基質濃度で最もNvaの光学純度が高かったため、これを選択した (表1)。
表1 ラセミNvaを基質とした第1段階酵素のスクリーニング
2-2 アミノ化酵素のスクリーニング
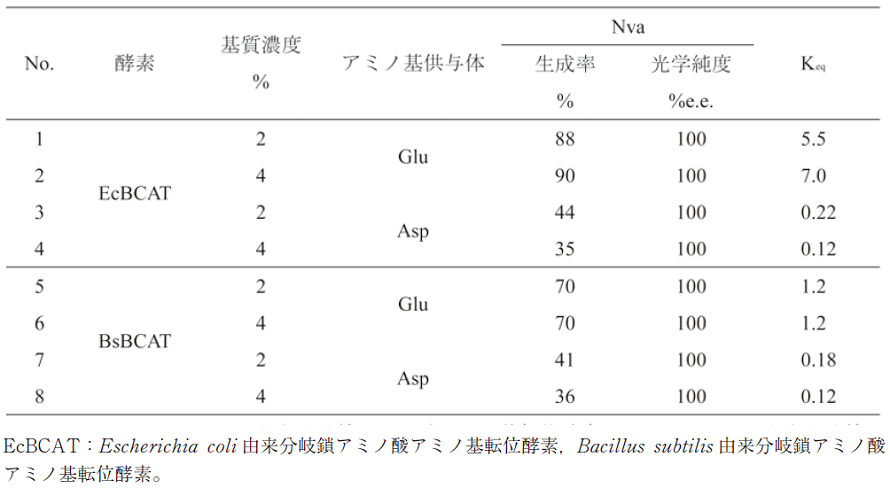
次いで、ケト酸をL-アミノ酸にアミノ化する酵素のスクリーニングを行った。まず、KVAを基質として、アミノ基転位酵素のスクリーニングを実施した。アミノ基転位反応は本質的には平衡反応であるため、溶解度の高いものを生成物とする場合には不利な反応である。そこで、アミノドナーには、通常用いられるL-グルタミン酸とL-アスパラギン酸の2種類を用いた。L-アスパラギン酸はアミノ基転位によりオキサロ酢酸を生成するが、これが大腸菌の内在酵素で代謝されてオキサロ酢酸濃度が低下し、平衡がL-Nvaの生成側にずれることを期待したものである。しかし、表2に示したように、L-Nvaの生成率はむしろL-グルタミン酸をアミノドナーとした方が高かった。アミノドナーをL-グルタミン酸、酵素をEscherichia coli由来分岐鎖アミノ酸アミノ基転位酵素 (EcBCAT) とした場合に平衡定数を求めるとおよそ6となり、KVAの変換率を99%とするためには、17当量のL-グルタミン酸が必要であると算出された。これではKVAの初濃度も上げられないこと、大量のL-グルタミン酸と目的物であるL-Nvaの分離操作が必要となることから、アミノ基転位酵素によるKVAからL-Nvaへの変換を諦めた。
表2 KVAを基質としたアミノ基転位酵素のスクリーニング
そこで、同じ反応を触媒する酵素として、アミノ酸脱水素酵素のスクリーニングを実施した。対象としたアミノ酸脱水素酵素は、L-アラニン脱水素酵素4種類、L-ロイシン脱水素酵素2種類、L-フェニルアラニン脱水素酵素1種類である。この場合も、KVAを基質とし、L-Nvaの生成率と光学純度を測定した。
アミノ酸脱水素酵素は、アンモニアをアミノドナーとし、補酵素NADHに依存的な還元的アミノ化反応を触媒する。補酵素NADHは基質と当量必要となるが、これは非常に高価であるために、当量を反応系に添加することは非現実的である。そこで、スクリーニング対象となるアミノ酸脱水素酵素とギ酸脱水素酵素 (McFDH)7) を共発現する組換え大腸菌を構築し、それを用いてスクリーニングを実施した。この系を用いると、ギ酸脱水素酵素で補酵素が再生され、菌体内に含まれる触媒量の補酵素で反応が進行することから、高価な補酵素を添加せずに反応を行うことができる (図2)。
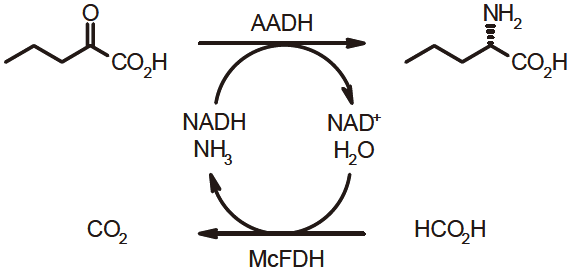
![]()
その結果を表3に示した。生成したNvaの光学純度はどの酵素でも100%e.e.であったため、光学純度の面からは外れる酵素はなかったが、Nvaの生成率が最も高かったThermoactinomyces intermedius由来のL-フェニルアラニン脱水素酵素 (TiPheDH)8) を選択した。
表3 KVAを基質としたL-アミノ酸脱水素酵素のスクリーニング
3.3遺伝子共発現プラスミドによる組換え菌の反応
ここで選択された3遺伝子を共発現するプラスミドpSFTPCO1、pSFTPCO2、及びpSFTPCO3を構築した (図3)9)。これらの違いは、遺伝子の連結順である。元となる3遺伝子発現プラスミドpSE420U8) はひとつのプロモーターで複数の遺伝子をオペロン状に発現させるのが特徴である。そこで、遺伝子の順序の最適化を図るため、これら3種類のプラスミドを構築した。上流に位置するほど発現量が多くなることが予想されたことから、遺伝子順を入れ替えることによって各酵素の活性比を変化させ、反応に与える影響を検討した。
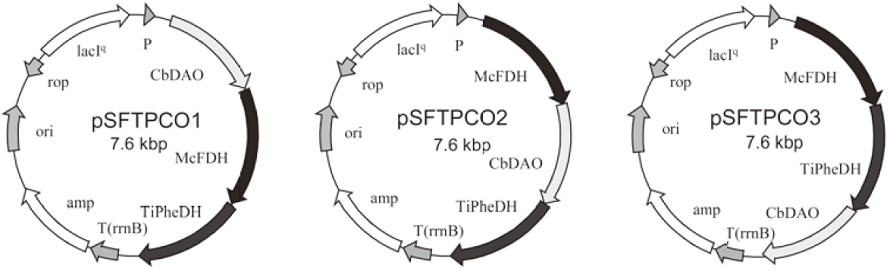

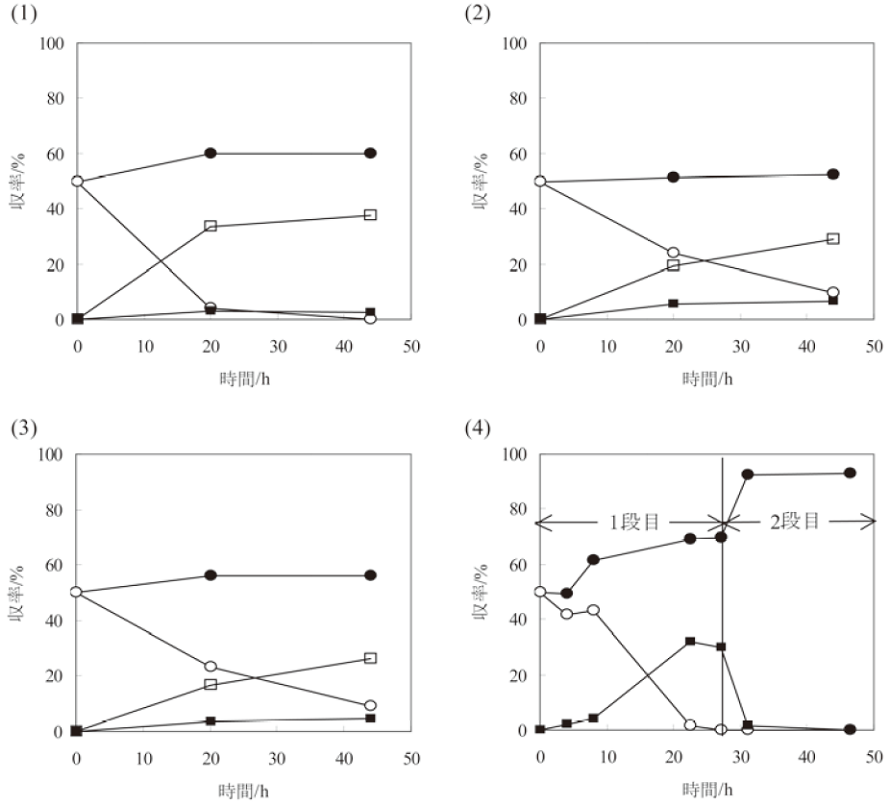
これらのプラスミドを含む組換え大腸菌を構築し、各酵素の活性を測定すると、予想した通り各酵素のバランスは変化していた。実際にラセミのNvaを原料とし、初濃度を12%として菌体反応を実施した結果を図4に示す。D-アミノ酸オキシダーゼCbDAO、ギ酸脱水素酵素McFDH、L-フェニルアラニン脱水素酵素TiPheDHの順に遺伝子を並べた3遺伝子発現組換え菌E. coli HB101 (pSFTPCO1) を使用した場合にD-Nvaが検出されず、L-Nvaの収率が最も高いことが示されたが、L-Nvaの収率は63%にとどまり、KVAは殆ど検出されなかった。収率が50%を超えていることによりデラセミ化は一部進行していることは示されたが、KVAの残存が見られないことから、NvaまたはKVAが分解されていることが示唆された。そこで詳細に反応液を分析したところ、反応液からは酪酸が検出された。この酪酸は、D-アミノ酸オキシダーゼの反応で副生する過酸化水素によってKVAが酸化分解を受けて生成したものと考えられ、NvaとKVA及び酪酸を合計すると化学量論的にも収支が合っていた。つまり、収率向上のためにはKVAの分解を抑えることが必要となった。

4.4遺伝子共発現プラスミドによる組換え菌の反応
4-1 4遺伝子共発現プラスミドによる組換え菌
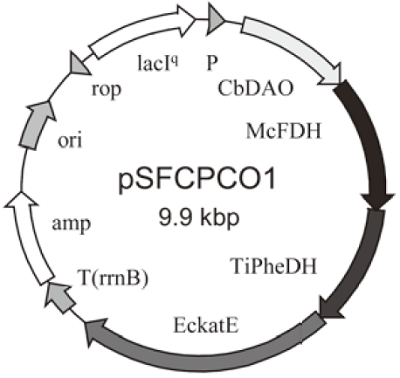
過酸化水素を分解する酵素として、カタラーゼが知られている。カタラーゼは大腸菌が元々生産する酵素であるが10)、更なる活性の増強を目的に、前述の3遺伝子共発現プラスミドpSFTPCO1の第4シストロンにEscherichia coli由来のカタラーゼ遺伝子を挿入したpSFCPCO1を構築した (図5)。このプラスミドは大きさが10 kbpに及ぶため、組換え菌を構築できるか、構築できたとしても各酵素が発現するかが危惧されたが、このプラスミドを含む組換え菌は4種類の酵素を安定に十分量発現していた。
![]()
そこで、4遺伝子共発現プラスミドによる組換え菌E. coli HB101 (pSFCPCO1) によるデラセミ化反応を実施した。ところが、L-Nvaの収率は73%までしか上昇せず、酪酸の生成は抑えられたものの、KVAが未反応のまま残存する結果となった (図4(4))。D-Nvaの酸化に必要な酸素を供給するための好通気条件下では、ギ酸脱水素酵素によりNAD+から再生させた NADH が宿主の電子伝達系により NAD+へ再酸化されるために、KVAからL-Nvaへの還元的アミノ化反応が進行しにくい条件になっていると考えられた。
4-2 反応条件の最適化
残存するKVAのL-Nvaへの変換反応を促進するため、2種類の反応条件を設定し、反応の進行に伴って条件の切り替えを実施した。即ち、反応の前半は好通気条件でD-Nvaの酸化を行い、D体の消失を確認した所で通気を停止し、KVAの還元的アミノ化を促進するというものである。同時にpHも第一段階ではpH 8.0に、第二段階ではpH 7.0に調整し、各段階でD-Nvaの酸化、KVAのアミノ化に至適のpHで反応させるように変更した。また、還元的アミノ化の際には、ギ酸脱水素酵素によるギ酸を基質としたNADHの再生に伴ってpHが上昇するため、pH調整を行う必要があるが、これをギ酸の添加で行うこととした。これには、2つの意味がある。ひとつは、恐らくはNADH酸化酵素により再生されたNADHが酸化され、ギ酸が当量よりも多く必要であるという知見が得られたため、NADH再生用基質を補う目的である。そして、精製の際に貧溶媒を添加して晶析するが、晶析母液組成で高濃度に溶解する塩として、硫酸塩等よりはギ酸塩の方が適当であったためである。
カタラーゼの導入によってKVAの分解を抑制し、更に反応条件を最適化することによって、反応収率は93%に達した。また、D体は非検出であった。このラセミ体を原料としたNvaのデラセミ化反応は、D体の酸化、KVAのアミノ化、補酵素の再生という複数の反応、更には副反応の抑制までを「単一の細胞」内で実施するものであり、ワンポットと言うよりもむしろCell Factoryと呼ぶ方が適当であると考えている (図6)。
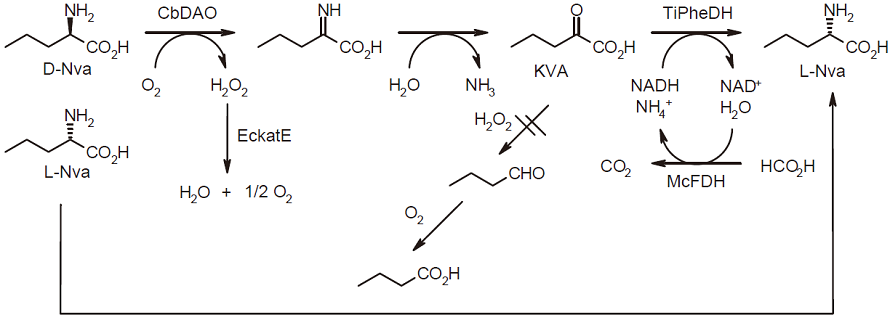
図6 Cell Factory内でのNvaのデラセミ化
5.おわりに
酵素によるデラセミ化反応は、ワンポットで理論的には100%収率を達成することができ、例えば光学分割反応のように、収率をあげるために不要な立体の誘導体を取り出し、ラセミ化を実施した後に再度分割を行う必要はない。デラセミ化は、机上では非常に美しい反応式として表されるが、実際に行うと様々な副反応が絡んでくる。しかし、複数の合成段階を経て得られる非天然化合物を、あたかも天然化合物のように細胞内で合成できるCell Factoryのシステムが成立することが示されたことは、酵素による物質生産の限界をひとつ取り払ったといえるのではないだろうか。
文献
1) Nakanishi, K., Takeuchi, A., Matsuno, R.: Appl. Microbiol. Biotechnol., 32, 633 (1990).
2) Nagel, U.: Angew. Chem. Int. Ed., 23, 435 (1984).
3) Nakajima, N., Esaki, N., Soda, K.: J. Chem. Soc. Chem. Commun, 13, 947 (1990).
4) For a review, see: Patel, R. N.: Biomolecular Engineering, 17, 167 (2001), and references cited therein.
5) 山本浩明, 小林良則:ファインケミカル, 36, 92 (2007).
6) Yurimoto, H., Hasegawa, T., Sakai, Y., Kato, N.: Yeast, 16, 1217 (2000).
7) Karzanov, V. V., Correa, C. M., Bogatsky, Y. G., Netrusov, A. I.: FEMS Microbiol. Lett., 81, 95 (1991).
8) Takeda, H., Yoshimura, T., Ohshima, T., Esaki, N., Soda, K.: J. Biochem., 109, 371 (1991).
9) Hayashi, M., Yamamoto, H., Kimoto, N.: WO patent, 132145 (2006).
10) Nakagawa, S., Ishino, S., Teshiba, S.: Biosci. Biotech. Biochem., 60, 415 (1996).