【トピックス】
核酸工学 ーFluorescent RNA : 光るRNAを目指してー
山東信介、青山安宏
京大院・工
1.はじめに
生命を構成する基本要素 "細胞" の機能は、核酸、蛋白質、脂質、糖、更には、低分子にいたる様々な機能性分子が複雑に連動しあい生み出されている。中でも、優れた酵素能を有する蛋白質は、細胞機能の中心的な役割を担っていることが知られている。また、細胞内での役割に留まらず、現在では多種多様な工学的応用が実現され、様々な物質生産にも応用されている。
一方、核酸はそれほど注目されてこなかった。極論すれば、細胞内での核酸の役割は、遺伝情報を蓄積し (DNA)、蛋白質翻訳系へと運搬する (RNA) 役割であると考えられてきた。また、リボザイムなど画期的な核酸酵素が発見されてはいるが、細胞内での重要さは蛋白質酵素に比べて非常に低く見積もられているといっても過言ではない。しかし近年、RNAなどの核酸が細胞機能の制御に非常に重要な役割を担っていることが明らかになりつつある。自分自身の翻訳頻度をコントロールし (riboregulator/riboswitch)、また、自分以外のRNA機能を制御する (microRNA/siRNA) ことも可能である。我々は "個々の細胞" に着目、均一と考えられている核酸の細胞間勾配・相違に焦点を当て、システムとして機能する細胞に対する核酸の貢献を明らかにしたいという目標を持ち研究を実施している。本稿では、核酸の理解を目指し、核酸工学的アプローチから開発された機能性核酸モジュールに関する研究を紹介させていただきたい1-6)。
2.光るRNA = Fluorescent RNA
重要な機能を担っていることが明らかになりつつある核酸であるが、細胞内での機能・機構・役割は驚くほどわかっていない部分が多い。その原因の一つとして、細胞内での核酸挙動を解析する技術の欠如が挙げられる。これは先述の通り、もともと細胞内での核酸の配列・転写・移動などを解析する必要がないと思われていたことに起因する。従来の配列診断・転写モニタリング技術はすりつぶした細胞より抽出した核酸を用いて実施することを前提としており、変異、発現状況は同一種とみなした対象細胞群の平均値として取り扱われてきた。一方、生きた細胞内で機能しているRNAの配列や挙動を解析するには、多様性の根源である細胞膜を破壊しない解析法が必須である。FISH (Fluorescence In Situ Hybridization) 法に代表されるように、蛍光分子を物理的に連結させた人工オリゴヌクレオチドを用いて細胞膜内のDNA/RNAを検出することは可能である。しかし、当然のことながら非特異的に結合するプローブを洗い流すためには細胞を固定化する必要があり、生細胞へは適応できない7)。
核酸に比べ、重要な標的と思われてきた蛋白質の解析は非常に進んでいる。例えば、細胞内で特定の蛋白質の発現・移動・局所化などを見たければ、最も簡単な方法は光る蛋白質 (Fluorescent Protein, 蛍光蛋白質GFPなど) を目的とする蛋白質にゲノムレベルで連結し、融合蛋白質として発現することである (図1a)8)。融合した蛍光蛋白質を蛍光顕微鏡で追跡することで、細胞内での蛋白質翻訳・移動・局所化などの解析が可能である。また、ガラクトシダーゼ・ルシフェラーゼなど酵素能を有する蛋白質を融合蛋白質として利用すれば、触媒的にシグナルを増幅し、微量蛋白質の発現を検出することも可能である。
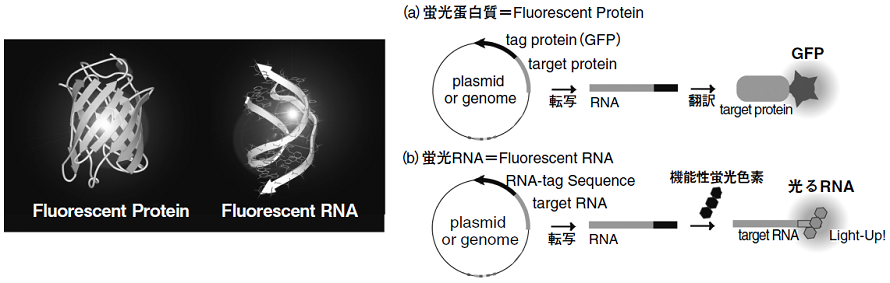
図1 新しい核酸モジュール:光る蛋白質と光るRNA
そこで近年、核酸に対しても同様に "光る配列" を創製し、核酸の局所化解析・発現検出、さらには、配列解析に応用しようとする試みがある9)。蛍光RNAをタグ配列として目的RNAにゲノムレベルで融合して細胞内で転写し、目的RNAの転写・移動・局所化を蛍光顕微鏡で観察しようとするものである。これは汎用されているFluorescent Proteinならぬ、Fluorescent RNAに他ならない。ただし、このアイデアには大きな問題がある。蛍光蛋白質は天然に存在するが、蛍光RNAは天然には存在しないのである。主要な酵素などを構成する蛋白質に比べ、非修飾核酸に蛍光シグナル発生などの機能を持たせることは難しい。この引き出しの少なさも核酸解析、また、その応用が難しい要因の一つである。
3.どのように蛍光RNAモジュールを作るか?
最も好ましい "蛍光RNA" は、蛍光蛋白質がそうであるように、自分自身の配列のみを用いて蛍光発色団を形成し、自己完結的に蛍光モジュールを形成するものである。しかし、天然に "光るRNA" が存在していないことを考えると、現実的にはこのアイデアの実現は難しい。そこで、最も形成が難しい蛍光発色団のみを外から加えてやり、RNAモジュールがその発色団を取り込んで蛍光を発するという "半人工/半天然" 型アプローチが注目を集めている。そのためには、①通常は無蛍光性であるが、②RNAモジュール配列を特異的に認識/結合して蛍光を発し、③膜透過性があり、かつ、④細胞の他のものには結合しないという恐ろしく困難な制約を克服できる蛍光発色団=機能性蛍光色素が必要不可欠となる。GFPなどの蛍光蛋白質はアミノ酸の分子内反応によって構築される蛍光発色団を蛋白質膜で覆って蛍光を発するのに対し、"蛍光RNA" モジュールでは、外から加えた膜透過性の蛍光発色団をRNA配列が覆って蛍光を発する仕組みである (図1b)。
4.RNAタグ配列に結合して蛍光を発する機能性蛍光色素
上述の通り、"光るRNA" を構成する機能性蛍光色素は、標的RNA配列に対する結合に伴って初めて蛍光量子収率を増大させるのみでなく、細胞膜透過性、生体直交性、低細胞毒性などの生細胞応用に必須となる項目も満足させる必要がある。本点は配列特異性の付加などに比べ、合理的設計による実現が非常に難しい部分であり、大きな問題となる。マラカイトグリーン/RNAタグ配列ペアなど可能性のある候補ペアは幾つか報告されているが10-13)、現状では実際の生体反応モニタリングを可能にした例はない。一方、現在細胞内核酸染色に使用されている蛍光色素群は、長期にわたる研究の結果得られたものであり、上記の点も克服済みである。我々はこのような優れた生細胞染色蛍光色素群を改変し、目的とする "特定の核酸タグ配列/構造に結合した場合にのみ蛍光を発する機能性蛍光色素" へと進化させる戦略を構築した5,6)。詳細は省略するが、Step1:分子モデリングに基づく環境応答性蛍光色素の化学修飾ライブラリの創製とStep2:核酸の試験管内進化法による結合核酸配列の探索を利用した有機化学/分子生物学を融合した戦略である。特に問題となるのは、Step2で得られた蛍光色素/核酸タグ配列の蛍光増幅能であるが、色素の周辺微視環境依存性を指標とした光化学的性質と融合させることで、本戦略に基づいたペアの探索を可能とした (図2a)。
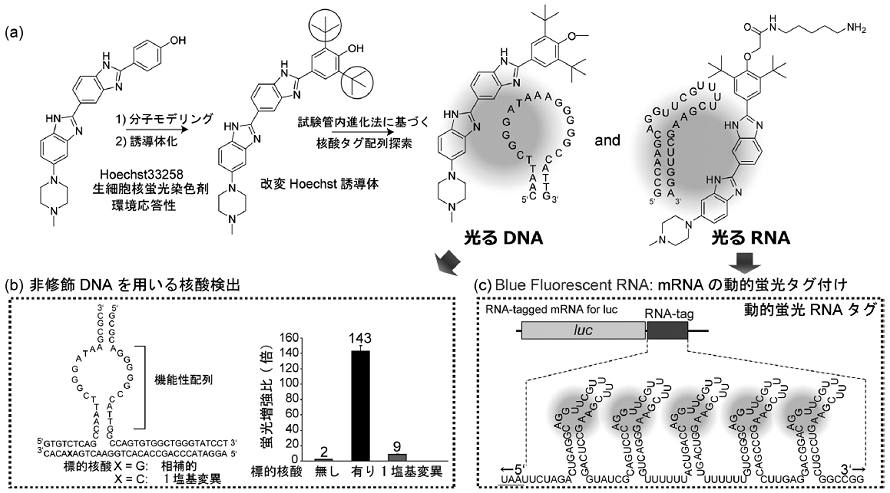
図2 青色蛍光DNA/RNAの創製と応用
5.光るDNA
実際、生細胞核染色に用いられるHoechst色素を用いて実験を行い、特定のDNA配列に結合した場合にのみ蛍光を発する "機能性蛍光色素" へと進化させることに成功した5)。その1例を図2に示している。新たに合成された色素ライブラリから選択されたこの改変Hoechstは、もともとの標的である2本鎖DNAに対する蛍光発光が抑制されているが、新しく選択された25-merのDNA配列に結合すると約190倍の蛍光増強 (200 nM, ex. 345 nm, em. 460 nm) を誘起した (蛍光量子収率0.37) (図2a)。また、核酸プローブをBinary 型 (標的核酸があるときにだけ、必須となる25-merの蛍光発光必須構造を形成する) に設計することで、1塩基精度での標的核酸検出に応用可能であった (図2b)。本結果は、天然DNAプローブを用いた核酸検出・配列診断系の構築が可能であることを示している。
6.光るRNA
RNAタグ配列の単離にも成功している (蛍光量子収率 = 0.26)6)。このRNAタグ配列を標的となるmRNAに融合した場合、その転写に伴いインテリジェント蛍光色素がRNAタグ配列に結合し、蛍光を発する。すなわち、RNA版の蛍光モジュール「光るRNA」である。実際、我々はluciferasemRNA末端に新しく創製した光るタグRNA配列を融合させ、in vitro T7-RNA転写系中におけるmRNAの選択的蛍光ラベル化が可能であることを実証した。この "光るRNA" が青色波長蛍光を呈することから、本系は世界で初めて実現された青色蛍光RNA=Blue Fluorescent RNAであるといえる (図2c)。
7.おわりに
活性分子として注目されてこなかった核酸に着目し、動的な挙動を解析する新しいモジュール開発について紹介させて頂いた。"蛍光RNA" の実用化が進めば、未だ不明な部分も多い機能性分子としての核酸に対する理解が進んでいくと期待される。今回紹介した戦略は様々な蛍光色素への展開が可能であり、Green、Blue、Red Fluoresncet RNAなど生細胞内での実応用可能な多色核酸ラベル化、レシオ測定を指向した様々な "光るRNA" の創製に挑戦している。また究極的には、外部添加蛍光発色団なしに蛍光を発するRNA配列を探索することも大きな目標である。そのようなRNAが見つかれば、真の意味で "Fluorescent RNAを作り出した" といえるであろう。
また、光るRNAモジュールの創製にとどまらず、核酸は複雑な蛋白質翻訳システムの中核 (=リボソーム) を担うなど、実は蛋白質に負けない機能を持っている。我々も翻訳系を改変し、複雑な非天然蛋白質合成に応用するなど種々のアプローチにも挑戦している14-16)。酵素=蛋白質という壁を打ち破れるよう、核酸を用いた様々な応用 (核酸工学) に向け少しでも貢献できればと思っている。
謝辞
本研究の主な部分はNEDO産業技術研究助成事業において実施しました。ご支援に感謝いたします。
文献
1) Sando, S., Abe, H., Kool, E. T.: J. Am. Chem. Soc., 126, 1081 (2004).
2) Sando, S., Narita, A., Abe, K., Aoyama, Y.: J. Am. Chem. Soc., 127, 5300 (2005).
3) Narita, A., Ogawa, K., Sando, S., Aoyama, Y.: Angew. Chem. Int. Ed., 45, 2879 (2006).
4) Narita, A., Ogawa, K., Sando, S., Aoyama, Y.: Nat. Prot., 2, 1105 (2007)
5) Sando, S., Narita, A., Aoyama, Y.: ChemBioChem, 8, 1795 (2007).
6) Sando, S., Narita, A., Hayami, M., Aoyama, Y.: Chem. Commun., in press.
7) Silverman, A. P., Kool, E. T.: Trends in Biotechnol., 23, 225 (2005).
8) Tsien, R. Y.: Annu. Rev. Biochem., 67, 509 (1998).
9) Famulok, M.: Nature, 434, 976 (2004).
10) Babendure, J. R., Adams, S. R., Tsien, R. Y.: J. Am. Chem. Soc., 125, 14716 (2003).
11) Sparano, B. A., Koide, K.: J. Am. Chem. Soc., 127, 14954 (2005).
12) Sparano, B. A., Koide, K.: J. Am. Chem. Soc., 129, 4785 (2007).
13) Constantin, T. P., Silva, G. L., Robertson, K. L., Hamilton, T. P., Fague, K., Waggoner, A. S., Armitage, B. A.: Org. Lett., 10, 1561 (2008).
14) Sando, S., Kanatani, K., Sato, N., Matsumoto, H., Hohsaka, T., Aoyama, Y.: J. Am. Chem. Soc., 127, 7998 (2005).
15) Sando, S., Abe, K., Sato, N., Shibata, T., Mizusawa, K., Aoyama, Y.: J. Am. Chem. Soc., 129, 6180 (2007).
16) Sando, S., Masu, H., Furutani, C., Aoyama, Y.: Org. Biomol. Chem., in press.