【トピックス】
色素性乾皮症バリアント群の責任遺伝子がコードするDNAポリメラーゼηの相同DNA 組換えにおける役割
川本卓男
京大・RI センター
1.はじめに
遺伝子DNAが安定に保持されることは、種の存続を保証するために必須である。しかし、われわれの遺伝子DNAは、太陽光中の紫外線 (UV) や環境中の化学物質、さらには、細胞自身の代謝物である活性酸素などにより、常にさまざまな損傷を受け続けている。そして、これらの損傷は、細胞死や突然変異をもたらす危険性を持っている。突然変異は、DNA複製時のエラーによっても生じ、老化、ガン化や、広範な病気の原因となる。ヒトを含めた地球上のすべての生物は、こうした好ましくない状態を回避するために、遺伝子DNAが受けた損傷を見つけて修復し、遺伝子DNAの情報を安定に保つための多様な機構を進化の過程で獲得してきた。したがって、こうした修復機構の何らかの要因による欠損は、ヒトに、早期老化、種々の神経症状、発育遅滞、高発ガン性、免疫異常などの重篤な病態をもたらす。正常人においても加齢による修復能力の低下が、老化や発ガン性に影響すると考えられる。しかしながら、通常の生命活動の中で恒常的に生じ続けるDNA損傷のすべてを、転写や複製の支障となる前に完全に修復するのは不可能といっても過言ではない。
近年、この不可能を補う巧妙な機構が明らかになってきた。DNA損傷による転写の阻害については、転写を阻害しているDNA損傷を優先的に修復する "転写と共役した修復機構" が存在する1)。一方、DNA複製の阻害に関しては、損傷を直接修復するのではなく、損傷をひとまず乗り越えてDNA複製を完了させてしまうという機構が存在することが明らかとなってきた1,2)。DNAの複製は、本来、非常に正確に行われなければならず、したがってDNAの複製を担当するDNAポリメラーゼは、非常に忠実にDNAを複製し、誤った塩基を取り込んだ場合にはそれらを取り除く、いわゆる "校正活性" をも備えている。こういった忠実度の高いDNAポリメラーゼの場合は、DNA複製の最中にDAN損傷があれば、そこから先のDNA複製を進行させることが出来なくなってしまう。しかし、近年、DNA損傷を乗り越えてDNAを複製できる新しいタイプの一群のDNAポリメラーゼが次々と発見され、DNA複製の最中にDNA損傷があった場合、こういったタイプのDNAポリメラーゼが、ひとまず損傷を乗り越えてDNA合成を行い (これを損傷乗り越え合成、translesion synthesis; TLSという)、DNA複製の損傷による阻害を回避することがわかったのである。DNAポリメラーゼη (polη) はこのTLSを行うDNAポリメラーゼで、マウス、ヒト、ニワトリ、酵母などでホモログが報告されており、種を超えてよく保存されている。ヒトにおいて、polηの欠損が、色素性乾皮症 (xeroderma pigmentosum; XP) のバリアントタイプ (XP-V) の原因であることがわかっている3-6)。XPは常染色体劣性遺伝様式をとる遺伝性疾患で、約10万人に1人の割合で発症し、日光露出部での色素沈着、高頻度ガンなどを特徴とする2)。インビトロの実験で、XP-V群の細胞は、polηが欠損するため、UVによるDNA損傷を乗り越えてDNAを複製することが出来ないことが示されている7)。
このようなDNAの損傷によるDNA複製の阻害は、相同DNA組換え (homologous DNA recombination; HR) によっても回避できることが知られている1)。後で説明するように、HRにはDNAを合成し伸長するステップが存在するはずだが、このHRにおいてDNA合成にかかわっているDNAポリメラーゼは明らかになっていなかった。そこで、我々は、polηがHRにおいてもDNAの合成にかかわっている可能性があると考え、HRにおけるpolηの役割を調べることにした。
2.相同DNA組換え (HR)
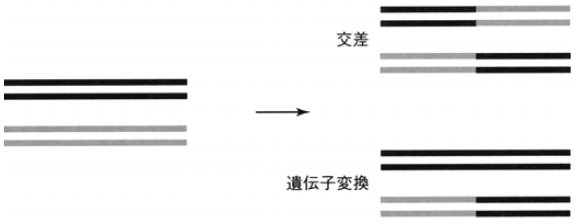
HRは、DNAの塩基配列がよく似た部位 (相同部位) で起こる組換えである。このHRによって、一対の相同DNAの間で、お互いに塩基配列がほぼ同じ部分を相互交換した形になるのが交差で、一方の相同DNAの部分が、相手の塩基配列がほぼ同じ部分で置き換えられる (相手の部分をコピーしたように見える) のが遺伝子変換である (図1)。HRは減数分裂時には高頻度で起こっており、このHRがうまくいかないと配偶子が形成されない (図2)。また、ニワトリ、ウサギ、ウシなどのBリンパ細胞の抗体遺伝子座では、HRによって複数個ある鋳型遺伝子配列の一部が、活性化している対応する遺伝子座の配列と置き換わること (遺伝子変換: gene conversion) によって、抗体遺伝子可変領域の一次構造が多様化する (Ig gene conversion)。たとえば、ニワトリの抗体軽鎖遺伝子は、VλJλとその上流領域に存在する25個のΨVλよりなる。HR (遺伝子変換) によって、VλJλの塩基配列が、対応するΨVλ中の塩基配列に置換される (図3)8,9)。

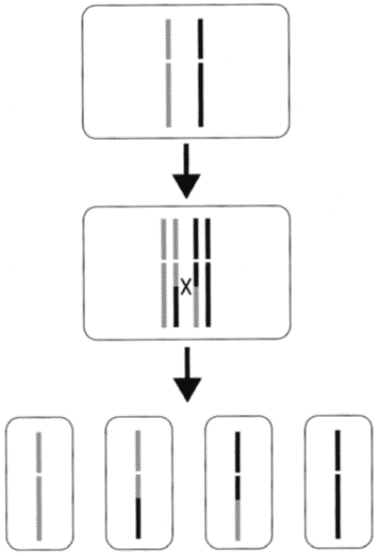

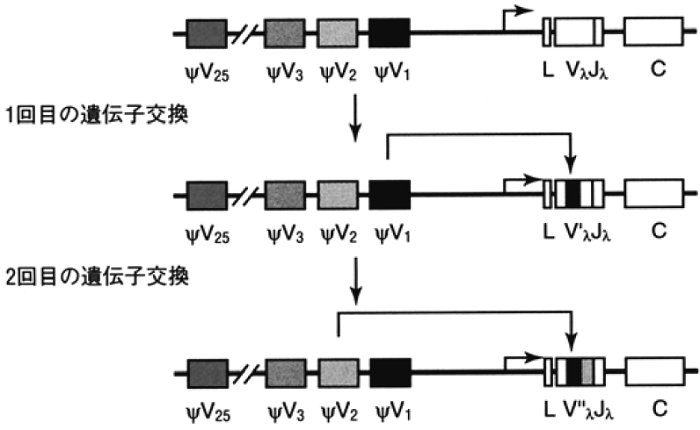
図3 ニワトリ抗体軽鎖遺伝子でのHRによる多様化の例
様々な化学物質や放射線により切断されたDNAの修復や、損傷によるDNAの複製の阻害の回避においてもHRが重要な役割を果たしていることが知られている1)。このようにHRは生物界で普遍的な現象で、それに働くタンパク質は細菌からヒトまで機能、構造ともよく保存されており、DNAの修復や減数分裂時のHRには多くの共通の分子が関与することが解明されている。これまでにわかっている酵母のHRの分子機構を放射線によるDNAの二重鎖切断の修復を例にして模式的に示したのが図4である。二重鎖断裂の結果生じた末端がMre11複合体の関与する反応によって処理され、一本鎖が形成される。この一本鎖DNA上にRPA、Rad51、Rad52などの分子が結合し、このDNA/タンパク質複合体が、相同な塩基配列を持つ別のDNAをサーチ (ホモロジーサーチ) する。そして、DNA/タンパク質複合体が相補的な塩基配列と対合し、ヘテロ二重鎖を形成し、DNA/タンパク質複合体のDNA 3’末端より相補的塩基配列を鋳型としたDNA合成が始まる。出来上がった二つの二重鎖がエンドヌクレアーゼにより切断され解離し、反応が完結する。多くのHRに関与する因子が、大腸菌、酵母、動物細胞により単離され、分子機構のモデルが提案されているが、いまだ明らかになっていない部分も多い。たとえば、前にも述べたように、ホモロジーサーチ後形成されたヘテロ二重鎖において、どのようなDNAポリメラーゼがDNA合成に関与しているのかはまったくわかっていなかった。我々はこのDNA合成に、TLSを行うDNAポリメラーゼが関与しているのではないかと考え研究を行ってきた。
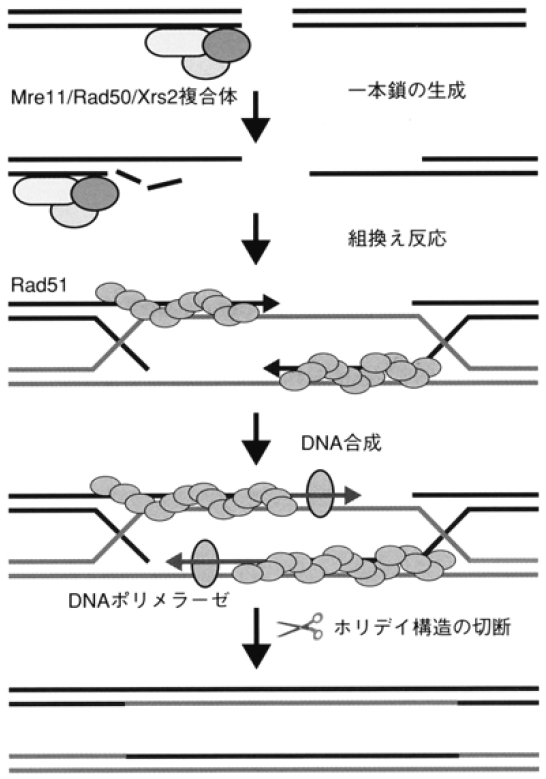
図4 酵母のおける二重鎖切断のHRによる修復の分子機構の模式図
近年、遺伝子ターゲティングがよく行われているが,これはHRを利用したものである。HRの分子機構に対する理解を深めることは、遺伝子ターゲティングの効率を上げるための手法を開発するためにも重要なことである。
3.DNAポリメラーゼη (polη) 欠損株の樹立
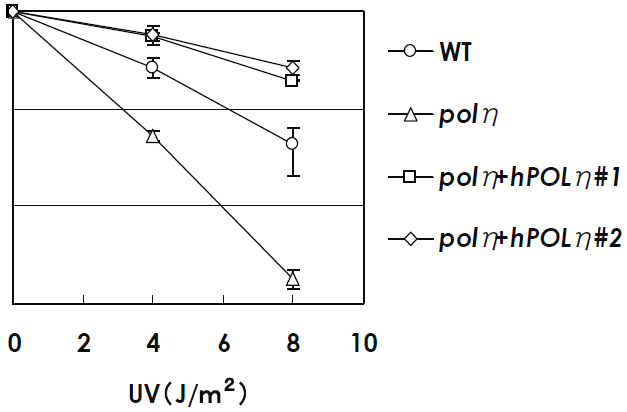
HRにおけるpolηの役割を調べるために、高等動物細胞の中で比較的効率よく遺伝子ターゲティングを行うことが出来るニワトリBリンパ細胞由来のDT40細胞株を用いて、polη欠損細胞株 (polη) の樹立を試みた10)。まず、ニワトリのPOLη cDNAを単離しアミノ酸配列を調べたところ、種を超えてよく保存されている五つの領域が、ニワトリにもあることが確認され、ヒトのその部分との相同性は約78%であった。全体として、ヒトpolηとの相同性は約78%だった。このニワトリPOLη cDNAの情報を元に、ニワトリpolηのノックアウト用コンストラクトを設計した。目的のPOLη遺伝子座への遺伝子ターゲッティングがおきているかどうかはサザンブロット解析により、また、遺伝子ターゲッティングの結果、確実にPOLηの転写が阻害されていることはRNAプロテクションアッセイによって確認した。3株のpolηが得られ、それらpolηの増殖速度は野生型 (wt) とほぼ同等であった。UVに対する感受性は、UV照射後のコロニー形成率を、UVを照射しない場合のコロニー形成率と比較することによって調べることが出来る。この方法を使ってpolηのUVに対する感受性を調べてみたところ、wtより明らかに感受性が高まり、UVに対する耐性が低下していた。また、polηにヒトPOLη遺伝子を導入し発現させることにより、polη欠損によるUVに対する耐性の低下を補うだけではなく、さらに耐性を上げることも出来た (図5)。これらのことにより、得られたpolηは、ヒトXP-V細胞と似た表現型を持つことが確認された。

4.二重鎖切断の修復にかわるHRにおける役割
動物細胞が、二重鎖切断の修復をHRによって行う能力をどれだけ持っているかは、以下に説明する細胞内で人工的に二重鎖切断を誘導しその修復能を測定する方法 (図6) によって評価することが出来る11)。I-SceIメガエンドヌクレアーゼの切断認識サイトを導入した薬剤耐性遺伝子 (これにより薬剤耐性遺伝子は発現されなくなっている) とその上流に野生型の耐性遺伝子 (プロモーター配列がないので発現しない) 配列を導入したDT40株は、I-SceIによって、二重鎖切断を誘導できる。I-SceIは普通のエンドヌクレアーゼとは異なり、18塩基配列にも及ぶ長い塩基配列を認識して切断する。したがって、人工的に認識サイトを導入したもの以外は、この酵素によって切断される可能性はないに等しい。I-SceIを発現させることによって誘導された二重鎖切断は、上流にある野生型の耐性遺伝子配列を用いたHR (遺伝子変換) によって修復され薬剤耐性を得る。したがって、薬剤存在下における薬剤耐性細胞のコロニー出現頻度によってこのHR能を評価できる。
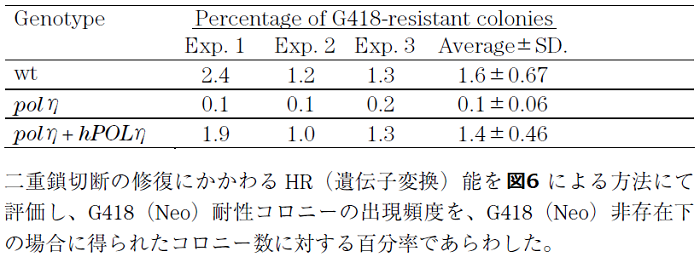
この評価法を用いてpolηのHR能を評価したところ、wtに比べその能力が低下していることが判明した。また、polηにヒトPOLη遺伝子を導入し発現させると、ほぼwtのレベルまで能力が回復することもわかった (表1)10)。
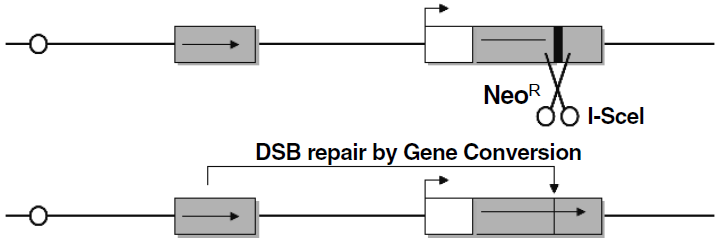

表1 I-SceIによる二重鎖切断によって誘導されたHR (遺伝子変換)
これらのことにより、polηが二重鎖切断の修復に関わるHRにて重要な役割を果たしていることが判明した。
5.抗体遺伝子可変領域の多様化にかかわるHR (Ig gene conversion) における役割
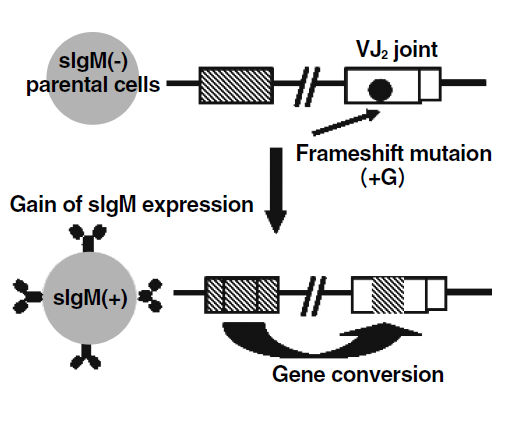
ある一定時間内に、抗体遺伝子可変領域の多様化にかかるHR (Ig gene conversion) がどれだけおきるかを測定する評価法を用いて、Ig gene conversionにおけるpolηの役割を検討した10)。我々がpolηを得るために用いたwtのDT40細胞株は、抗体遺伝子可変領域にグアニン (G) 一塩基分によるフレームシフトを起こしており、sIgMを産生することが出来ない12)。このフレームシフトを持つ領域が、Ig gene conversionによって置き換われば、sIgMを発現するようになる (図7)。よって一定期間の培養中に、sIgMを発現するようになった細胞を、イムノアッセイを利用してカウントすればIg gene conversionを起こす能力が評価できる13)。我々が単離した3株のpolηも上記のようなフレームシフトが保存されていることが確認できた。そこで、wtと3株のpolηについてそれぞれsIgMを発現していない細胞から30~40のサブクローンを得、それらを別々に3週間培養し、それぞれのサブクローンについてsIgMを発現している細胞がどれだけの割合で存在するかカウントした。その結果、polηについては、その割合がwtの1/2~1/6のレベルに減少することがわかった。また、polηにヒトPOLη遺伝子を導入し発現させることによって、割合をwtのレベルに近づけることが出来ることも確認できた (図8)。そこで、Ig gene conversionにpolηのポリメラーゼ活性が実際に必要かどうかを確認するため、2箇所の変異が入ったヒトPOLη遺伝子をpolηに導入し、活性を失ったpolηを発現させ、同様に試験をしてみた。その結果、polηに活性を失ったpolηを発現させても、sIgMポジティブな細胞の割合は低下したまま (図8) であり、Ig gene conversionにおいてpolηはDNA合成に直接関わっていると考えることが出来る。

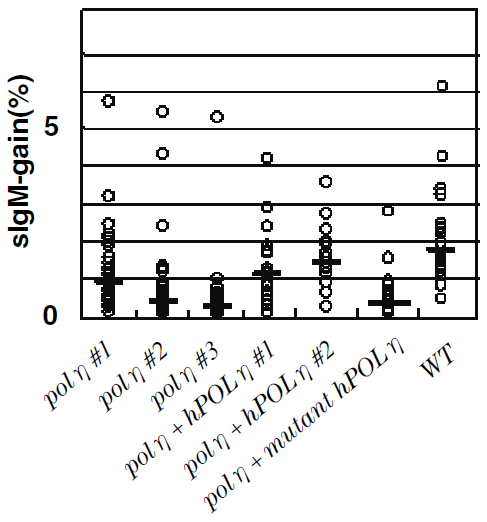

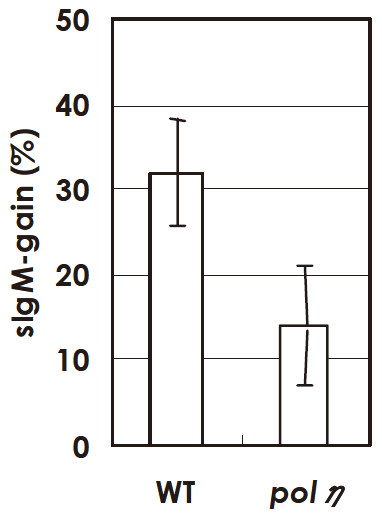
ここで用いた評価法では、wtにおいてもsIgMポジティブな細胞に変化したものの割合は、約1ヶ月間にわたって培養しても1~2%程度の低い値である。ヒストンデアシラーゼインヒビターであるトリコスタンチンA (TSA) 存在下で培養すると、Ig gene conversionの起きる頻度を約50倍上げることが出来ることがわかっている14)。そこで、同様な評価法をTSA存在下で培養することによって行ったところ、図9に示したようにwtに比べpolηの場合sIgMポジティブな細胞に変化したものの割合が減少することを確認することが出来た。

6.おわりに
色素性乾皮症バリアント群の責任遺伝子がコードするpolηはTLSにおいてだけでなくHRにおいても重要な役割を担っていることが示された。しかし、polηにおいてもHRが起こらなくなるわけではないので、HRにおいてpolηをバックアップするDNAポリメラーゼが存在するのであろう。今のところ、バックアップしているDNAポリメラーゼが何であるか詳しいことはわかっていない。また、HRにおいてpolηが実際どのようにDNA合成に関与しているのかも明らかになっていない。HRの分子機構は、明らかになっていないことが多く、今後、さらなる研究が必要である。
本研究は、京都大学大学院医学研究科放射線遺伝学武田研究室で行ったものであり、ご指導いただいた武田俊一教授、数々の貴重な助言をいただいた園田英一郎先生ならびに協力いただいた研究室の皆様方に、改めてお礼申し上げます。
文献
1) 安井明、花岡文雄、田中亀代次 (編) : DNA修復ネットワークとその破綻の分子病態, 共立出版 (2001).
2) Lehmann, A. R.: FEBS Lett., 579, 873 (2005).
3) Cordonnier, A. M., Fuchs, R. P.: Mutat. Res., 435, 111 (1999).
4) Johnson, R. E., Prakash, S., Prakash, L.: J. Biol. Chem., 274, 15975 (1999).
5) Masutani, C., Kusumoto, R., Yamada, A., Dohmae, N., Yokoi, M., Yuasa, M., Araki, M., Iwai, S., Takio, K., Hanaoka, F.: Nature, 399, 700 (1999).
6) Masutani, C., Kusumoto, R., Iwai, S., Hanaoka, F.: EMBO J., 19, 3100 (2000).
7) Masutani, C., Araki, M., Yamada, A., Kusumoto, R., Nogimori, T., Maekawa, T., Iwai, S., Hanaoka, F.: EMBO J., 18, 3491 (1999).
8) Haber, J. E.: Trends Genet., 8, 446 (1992).
9) Reyaud, C. A., Bertocci, B., Dahan, A., Weill, J. C.: Adv. Immunol., 57, 353 (1994).
10) Kawamoto, T., Araki, K., Sonoda, E., Yamashita, Y. M., Harada, K., Kikuchi, K., Masutani, C., Hanaoka, F., Nozaki, K., Hashimoto, N., Takeda, S.: Mol. Cell, 20, 793 (2005).
11) Fukushima, T., Takata, M., Morrison, C., Araki, R., Fujimoto, A., Abe, M., Tatsumi, K., Jasin, M., Dhar, P. K., Sonoda, E., Chiba, T., Takeda, S.: J. Biol. Chem., 276, 44413 (2001).
12) Buerstedde, J. M., Reynaud, C. A., Humphries, E. H., Olson, W., Ewert, D. L., Weill, J. C.: EMBO J., 9, 921 (1990).
13) Hatanaka, A., Yamazoe, M., Sale, J. E., Takata, M., Yamamoto, K., Kitao, H., Sonoda, E., Kikuchi, K., Yonetani, Y., Takeda, S.: Mol. Cell Biol., 25, 1124 (2005).
14) Seo, H., Masuoka, M., Murofushi, H., Takeda, S., Shibata, T., Ohta, K.: Nat. Biotechnol., 23, 731 (2005).