【トピックス】
メタゲノムを利用した新規酵素の探索
宮崎健太郎
産総研・生物機能工学
1.はじめに
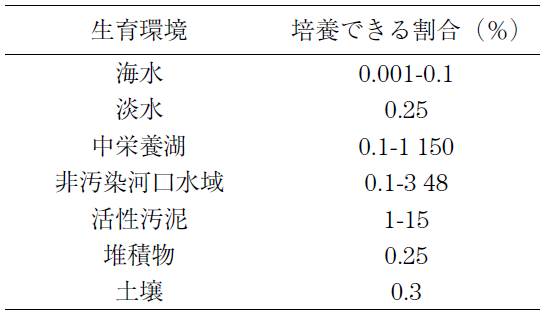
生物機能を活用した物質生産プロセスを構築するためには、自然界から有用な酵素をいかに探し出すかが鍵となる。従来、目的とする酵素を取得するには、まずその生産菌を環境から単離することにより行われてきた。しかし、近年、分子生物学的手法を駆使した微生物生態研究により、環境中に生育する微生物の大部分は、通常の方法では実験室で分離培養することが困難な「難培養性」であることがわかってきた1)。表1に〈培養できる細菌の割合〉をまとめたが、微生物スクリーニング源として最も一般的な土壌で1%未満、海洋では0.1%未満となっており、ほとんどの微生物は、実は培養などできないことがわかる。このため、目的酵素の生産菌を単離するという従来の手法は、環境中に生育する微生物のうちのごく一部、氷山の一角を探索しているに過ぎないことになる。もちろん、自然界の誇る多様性は十二分に広く (土壌1 gにも数千~数万種類、数にして数億の微生物が存在すると言われている)、「一角」のみの探索でもちっとも目減りしたことにならないという意見もあろう。現時点では、純粋培養が容易な微生物とそうでないものとの個体としての違い、保有する遺伝子の違いについて不明な点が多いが、このような中、我々は難培養性微生物の生物資源としての新たな可能性を模索すべく、メタゲノミクス (metagenomics) と呼ばれる手法により遺伝子スクリーニングを行い、得られた遺伝子の新規性について解析した。本稿では、メタゲノミクスの概略とそれを用いて我々が行った芳香族分解系遺伝子のスクリーニングを紹介する。
表1 さまざまな環境と培養できる細菌の割合
2.メタゲノム手法とは?
「メタゲノム」とは、環境中に含まれる微生物ゲノムの混合体である2)。「ゲノム」に対して、「複数の」という意味を表す「メタ」という接頭語がついている。環境DNA (environmental DNA, eDNA) とほぼ同義であるが、最近ではメタゲノムという言葉の方が多く用いられるようになっている。この方法では、微生物を純粋分離せずに、環境に含まれる多種多様な微生物から直接ゲノムを抽出し、そこに存在するすべての微生物ゲノムを混合物として手に入れる。環境には多くの夾雑物が含まれるため、この操作は簡単ではないが、一度DNAとして獲得してしまえば、その後は比較的容易に取り扱いことができる。物質としてのDNAは安定であり、ゲノムDNAを細切れに断片化してベクターに連結し、大腸菌等に保持させれば、元の微生物環境を遺伝子という形で保存・複製・利用できる。いささか荒っぽくはあるが、微生物遺伝子を網羅する、ありのままの姿を捉えるための方法論として、最近高い注目を浴びている2)。
メタゲノムを利用することの最大のメリットは、当然、培養法ではアクセスし得ない微生物の遺伝子を利用できることである。研究室で培養すると待ちきれないほど生育の遅い微生物も、他の微生物との共存関係の中ではじめて生きていける微生物も、十把一絡げにして取り扱えばよい。第二のメリットは、個々の遺伝子を生理的なネットワークから解放できる点である。遺伝子を断片化することで、whole cellとしての機能を復元することは諦めざるを得ないが、逆にこれが細胞生理のしがらみから逃れるのに役に立つ。ゲノムにコードされている遺伝子は、のべつ発現しているわけではない。大抵の遺伝子は、ある条件、あるタイミングで指令がくだり、それに従い機能する。微生物個体を取り扱う場合には、この発現制御の実態を知らなければ、たとえゲノム中にコードされていても機能発現させることはできない。一方、異種宿主を用いて小さな遺伝子断片を発現させることは、本来の生理的なコンテクストとかかわりなく、また宿主の干渉も極小に抑えた中で実行できる。本来は特殊な条件でしか発現しない遺伝子も、組換えシステムの中では強制発現され、速やかにその実態を表すことであろう。単一酵素による物質変換や、ごく少数の遺伝子で実現される生物機能をスクリーニングの対象とする際には、これが大きなメリットとなる。
デメリットについても挙げておこう。先述の通り、whole cellとしての機能は再現できない。また、いくつもの遺伝子が関わる複雑な生合成経路などは、それがオペロン化されてない限り、その機能を捉えることはできない。さらに、DNA断片の微生物起源が分からなくなってしまうことも問題である。素性のわからない遺伝子を取り扱うことは、遺伝子発現などの点で戦略を立て難く、また実利用する際にも (用途によっては) パブリックアクセプタンスを受け難いことも懸念される。研究室での扱いも当初は「未同定実験」であり、若干敷居は高い。
これらのメリット・デメリットがある中、まずメタゲノムの新規性・有効性を実証しなければ将来的な展開も予測できない。そこで我々は、まずは様々な酵素・蛋白質をターゲットとしてメタゲノムライブラリーからの遺伝子スクリーニングを行い、我々なりの解答を得ることを試みた。本稿では、とくにエクストラジオールジオキシゲナーゼ3) を標的としたスクリーニングを例にとり、取得された遺伝子の特徴、新規性、さらには環境適応機構について述べてみたい。
3.新規芳香族分解系遺伝子の同定
微生物による芳香族化合物の好気的分解は、カテコールを中間体とするルートが主要である。エクストラジオールジオキシゲナーゼ (extradiol dioxygenase; EDO) は、カテコールのメタ位を開裂する酵素で、開環化合物は黄色を呈する (図1)。今回我々は、メタゲノム源として工業廃水処理に使われる活性汚泥を利用した。廃水中には芳香族化合物が豊富に含まれており、通常の環境よりも分解微生物が多く存在するだろうというもくろみもあった。汚泥よりDNAを抽出し、平均鎖長35 kbとなるようにDNAを断片化し、フォスミドベクターに連結し、大腸菌を宿主としてライブラリーを構築した。メタゲノムDNA断片長を合計すると3.3 Gbサイズに及び、微生物ゲノムの大きさを5 Mbとすれば、これは600-700 匹分の微生物スクリーニングを行っていることに相当する。我々は、こうして構築したライブラリーを96穴フォーマットのマイクロプレートにアレイ化し、以下、全ての実験をこのハイスループットのフォーマットで一貫して行えるようにした。ただし、実際のクローン数が96,000と膨大であったため、まともに扱うと (1穴あたり1クローンでは) マイクロプレート1,000枚分となってしまう。現実的には我々の実験キャパシティを大きく超えてしまうため、次善の策として1穴あたり100クローンを混合する方法をとった (96穴マイクロプレート10枚の処理)。もちろん、感度低下は避けられないが、スループットとの兼ね合いからこのような妥協点とした。より一般的には、発色基質や不溶性基質を含む寒天プレートを用いて、呈色あるいはクリアゾーンにより陽性クローンを選別する方法もあるが、我々はメタゲノムゆえの活性の低さを懸念し、多少の手間はかかるが、培養から酵素活性の検出までの工程を、段階的に至適化できる溶液系でスクリーニングをセットアップした。
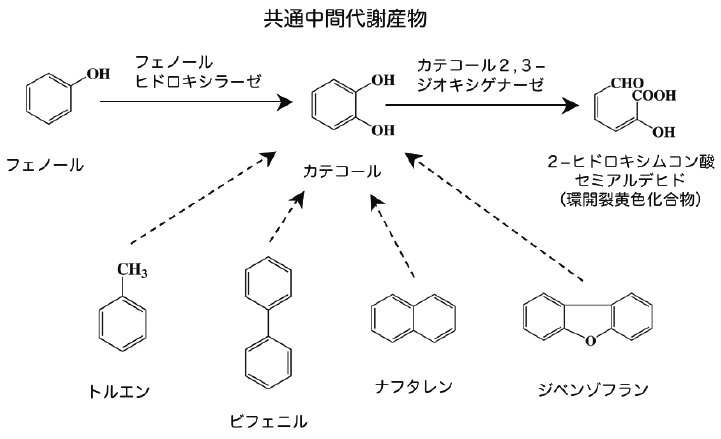
図1 微生物による芳香族化合物の好気的分解
大腸菌を培養し、粗酵素抽出液を調製し、基質カテコールと混合後、黄色反応産物の有無を指標に活性スクリーニングを行った。当初、余り工夫なく実験を行っていた際には陽性クローンは一つも得られなかったが、大腸菌の培養スケールの変更、ベクターコピー数の増強、添加基質濃度の向上、検出タイミングの改良を行った結果、最終的には85もの活性クローンが得られた。この数字は、論文として公開されているメタゲノムからの何らかの遺伝子をスクリーニングしたときの「数」としてはトップランクである。この数字に達するには各種パラメータの至適化が必要であったが、逆に言うと、固定条件ではなかなか陽性クローン数が伸び悩むということで、溶液系の利点を確認した。次に、至適化された液体評価系の感度を最も一般的な寒天培地を用いたプレートアッセイと比較してみた。強弱2種類の活性クローンを寒天培地上に生育させ、カテコールを与えてみると、液体評価系では強く黄色を呈したクローンですら、寒天培地上ではようやくそれと認識出来る程度であった。一方、反応溶液中で弱い黄色しか示さなかったクローンに至っては、ネガティブクローンとの判別は困難であった。
4.メタゲノムらしさの評価
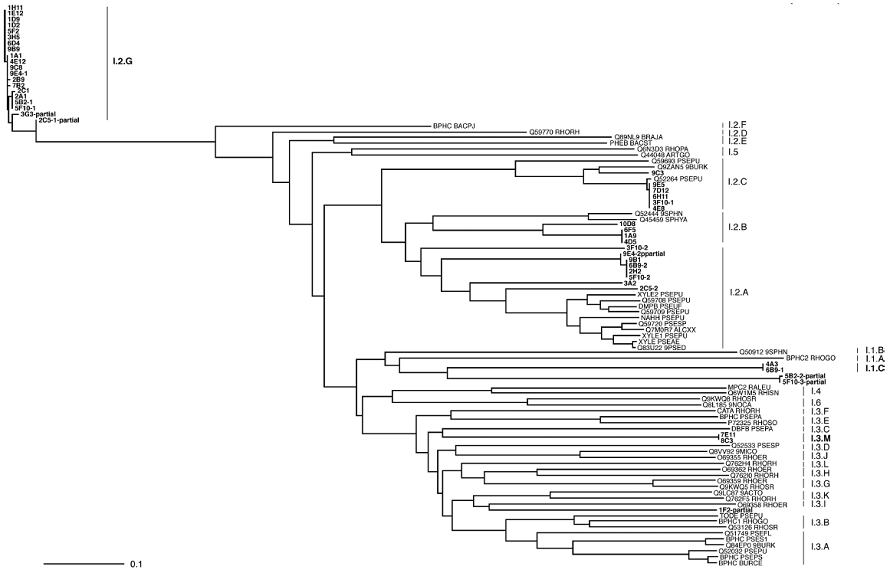
このように、我々はメタゲノムライブラリーから多くのEDO遺伝子を得ることに成功した。では、「メタゲノムらしい結果」とはどういうものであろうか?多くのクローンを得たことで何が明らかになったのか?このメタゲノム研究の根源的問いに答えるべく、まず獲得した91のEDO活性フォスミドクローンのうち、38クローンのショットガン解析を行い (全てを解析しなかったのは経済的理由による)、43のEDO遺伝子を同定した。これらの中には、培養に基づく方法で高頻度に得られるサブファミリーに属する遺伝子がいくつか含まれるのと同時に、既存のサブファミリーに分類されないEDO遺伝子が半数以上存在した。これを受け4つの新規なサブファミリーを提唱した (図2)。カテコールは微生物による様々な芳香環代謝において共通の中間代謝産物であり、EDO酵素はバイオレメディエーションにおけるキーエンザイムとして研究歴も長い。にもかかわらず、今回一度のスクリーニングで多くの新規遺伝子の獲得できたこと、さらにはこれら新規遺伝子の一群が環境中のメインプレーヤーである可能性が示唆されたことは、微生物培養に依存してきた我々の知識、経験が根底から覆されかねないことを意味する。これら43クローンについては、カテコールアナログに対する反応性も検証し、機能の点からも様々な新規酵素が含まれていることが確認された。
![]()
5.EDOの環境適応戦術
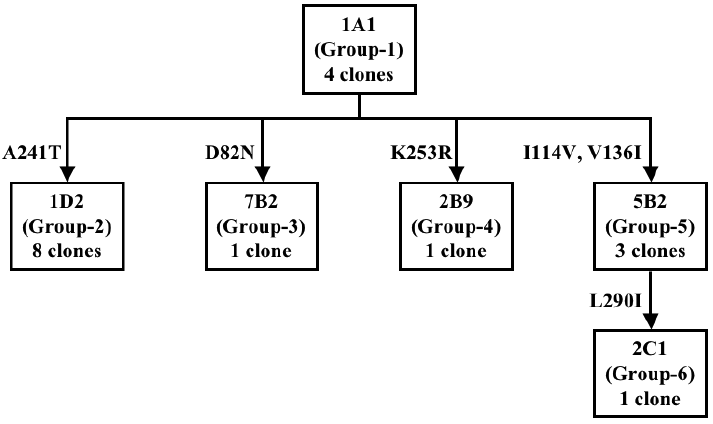
次にメタゲノムライブラリーより得られたEDOの環境適応について検討した。先の図2においても示したとおり、ライブラリーより得られたEDOのうち、新規サブファミリーに属するI.2.G EDOが最も環境中で優占化していることが示唆された。スクリーニングで得られた陽性クローン数が、実環境中での存在量を素直に反映しているかについては若干怪しいところもあるが、今はこれに目をつぶり議論を進める。なお、I.2.Gサブファミリーは均一なEDO集団ではなく、塩基変異により6種類の多型が存在する。図2の系統樹をI.2.Gの部分のみ抜き書きしたのが図3である。そこで、6種類のI.2.Gの酵素とともに、I.3.MおよびI.3.NサブファミリーのものをHis-タグ付き酵素として大腸菌内で過剰発現し、均一に精製し、各種解析に供した。
![]()
まずI.2.G EDOとその他のサブファミリーとの最も大きな違いは、金属要求性である。既知のEDO、また今回得られたI.2.G以外のサブファミリーに属する酵素は全てFe依存的な酵素であったが、I.2.G酵素についてはMn依存性であった (図4)。我々は、メタゲノムが得られた環境 (工業廃水) のことを考え、Mn要求性が環境適応において何らかの役割をそていおるのではないかと考え、様々な実験を行った。その結果、I.2.G酵素は同じ環境から得られたFe依存性のI.3.MやI.3.N EDOと比較し、シアンや過酸化水素に対して耐性があることを見出した (図5)。中でもシアンは金属結合の阻害剤であり、酵素としてはシアンによる阻害を受けないためにも、よりキレート力の強いものを使いたい。その結果、通常はFeであるところ、Mnを利用できるように進化してきたと考えられた。
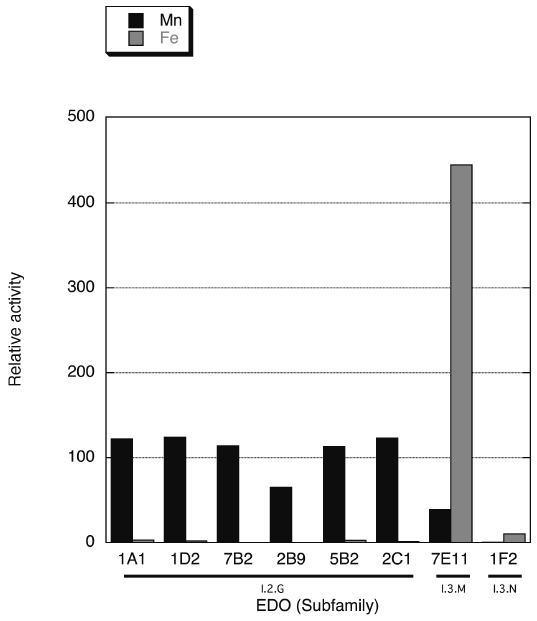

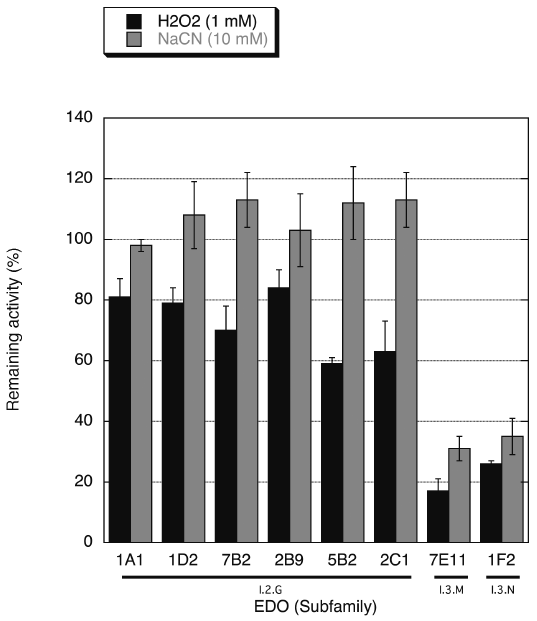
つぎに、I.2.Gサブファミリー内を詳細に見た。6種にグループ分けされた酵素のクローン数はマチマチであり、多型 (アミノ酸変異) による酵素特性の違い、多型が生まれる分子基盤について興味が持たれた。酵素の活性について詳細な速度論的な解析を行った結果、1D2と5B2は、祖先型1A1よりも2倍程度高い活性を有していることが判明した。図3と合わせて見ると、これら高活性型の子孫酵素が数ある子孫酵素の中でも優占化していることがわかる。また、安定性の評価からは、高活性の子孫酵素の安定性は祖先型1A1よりも低下しており、祖先型酵素が元々持っていた安定性を捨てて活性を向上する進化に役立てたと解釈された。
以上をまとめると、シアンなどの金属キレート存在下に抗すべく、Mnを利用するI.2.GタイプのEDOが生まれた。そのプロトタイプが1A1である。I.2.G EDOはMnを使うことで物理的強度を増し、そのエキストラの安定性を活性進化に利用することが可能となり、1A1はさらに高活性型の子孫へと進化していく、というシナリオである。蛋白質にとって一見無駄とも思える安定性が新たな機能獲得に有利であること4)、活性と安定性との間でトレードオフが見られること5) は過去にも指摘があったが、今回の実験では環境中で遺伝子が進化していく様子をリアルに捉えたものと言えよう。
6.おわりに
以上、メタゲノム手法を用いた酵素スクリーニングについて紹介したが、期待通りに新規な酵素が得られそうだとの感触は得た。実際、ここでは紹介しなかった酵素・蛋白質についても、配列や機能の点で新規なものが続々と見つかってきている。また、当初想定していなかったことであるが、環境中での遺伝子動態、進化についても多くの知見を得られることがわかった。新規な酵素イコール有用酵素とは即結びつかないが、メタゲノムを利用した有用酵素の発見、バイオプロセスの構築へと研究を展開していきたいと思っている。
文献
1) Amann, R. I., Ludwig, W., Scleifer, K. H.: Microbiol. Rev., 59, 143 (1995).
2) Handelsman, J., Rondon, M. R., Brady, S. F., Clardy, J., Goodman, R. M.: Chem. Biol., 5, 245 (1998).
3) Suenaga, H., Ohnuki, T., Miyazaki, K.: Environ. Microbiol., 9, 2289 (2007).
4) Bloom, J. D., Labthavikul, S. T., Otey, C. R., Arnold, F. H.: Proc. Natl. Acad. Sci. USA, 103, 5869 (2006).
5) Arnold, F. H., Wintrode, P. L., Miyazaki, K., Gershenson, A.: Trends Biochem Sci., 26, 100 (2001).