【トピックス】
DNAコンピュータから試験管内「人工生命」へ
木賀大介
東工大・院総合理工・知能システム科学専攻
1.はじめに
生命は複数種類の生体高分子が組み合わさって自律的に動作するシステムである。その構成要素であるDNA、タンパク質はそれぞれ生命に必要な遺伝情報、酵素活性を持つ高分子であるということが生物学者共通の認識であろう。生命をよりよく知るため、もしくは生命に関わる物質を生産するために、これらの分子はツールとしても使用されている。しかし、DNAに生物とは無関係な情報をエンコードし、これらに対して生物学者の良く知る実験操作を加えることが超並列演算となる、というDNAコンピュータの実演は1)、非常に驚きの対象であった。
DNAコンピュータのメリットとして当初は、電子計算機よりも高速な演算を行える、という点が強調されたため、より重要なメリット、すなわち、生体分子で構成されているがゆえに天然の生命との親和性が高い人工分子システムである、という利点が見落とされている観があった。しかし近年では、DNAコンピュータの発想が、生命情報を解析・活用する、また、自律的に動作する分子システムを生体分子を用いて構築する、さらには試験管内における人工生命の構築を目指す、という合成生物学的な方向に発展しており、本稿で一連の研究の流れを解説したい。
2.DNAコンピュータの本質
種々のDNAコンピュータ研究に見られる共通項は、核酸の相補鎖認識能力を活かして「検索」を行っていることである。多数の分子が混合する生体内において、正しく対応した分子同士が結合していくという状況は、情報科学的に捉えると、超並列的な検索が進行している、とみなすことができる。中でも、核酸の相補鎖認識能力は、対応関係を研究者によって簡単に規定することができる。例えば、ビーズに固定化したDNAプローブによって、一本鎖核酸のライブラリの中からプローブの相補配列を持った分子のみを取り出す操作をイメージしてもらいたい。このような、一回の実験操作において多数の候補の中から対応する相手を探し出すことが可能な溶液中の分子に対する実験操作と対照的に、電子計算機においての検索では、データ群のなかから正しく対応したデータを検索するために、候補データを一つずつ確認する必要がある。この溶液反応のメリットを活かした超並列計算機として、DNAコンピュータの当初のアピールはなされていた。
3.情報科学の問題を解くDNAコンピュータ
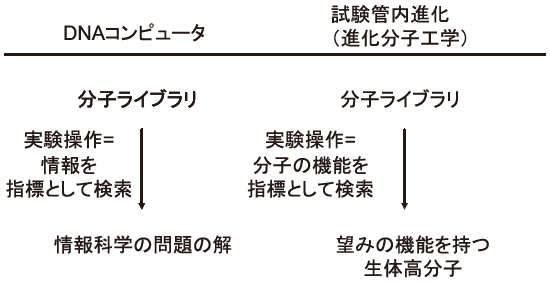
最初のDNAコンピュータである、「一筆書き」の解を検索した実験操作は、in vitro セレクション法によるアプタマーなど機能性核酸の単離のための実験操作と対応付けることができる (図1)。まず共通点としては、どちらも一本鎖核酸ライブラリの中から、目的とする配列を持った核酸を単離することが挙げられる。次に違いとして、ライブラリの作成方法と単離の基準が挙げられる。in vitroセレクション法でのライブラリは1塩基ごとにランダム化されたコンビナトリアルなライブラリであるが、DNAコンピュータでは、15-25残基程度の一まとまりの配列に意味を持たせるため、これらのまとまりごとにランダム化されたコンビナトリアルなライブラリを調製することとなる (図2)。また、アプタマーの単離では一本鎖核酸の立体構造形成による分子認識機能を基準に選択操作が行われるが、DNAコンピュータでは上述の一まとまりの配列を持っているか否かによって選択操作が行われる。
図1 DNAコンピュータと試験管内進化の対応関係
図2 Adlemanによる一筆書きの解法
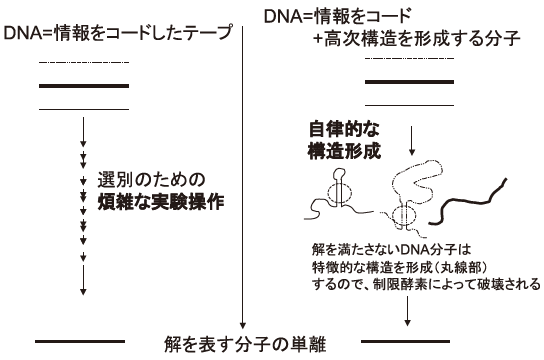
最初のDNAコンピュータでは、DNAは情報を記載した1本のテープとして扱われていたが、我々の行った次段階の研究では、一本鎖DNAが高次構造を形成する能力を活かすことを試みた2) (図3)。以前の研究では、N項目のチェック事項が合った場合、それぞれの項目に対するプローブを一度に1種類ずつチェックする、N回の実験操作が必要であった (図3左)。一方この研究では、それぞれの項目ごとにステム構造を形成するか否という基準でのチェックを可能とするような情報の書き込みを行った。ステム構造を持ったDNAを制限酵素で破壊することによって、1回の実験操作で複数の項目をチェックすることが可能となり (図3右)、解を得るための実験操作が解の候補の数 (チェック項目の数) に依存しなくなるという簡略化を達成した。

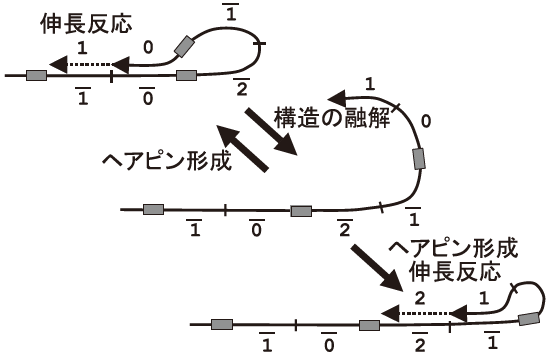
上述した方法では、一度限りの自律的なDNAの形態変化を活用したが、続く研究では、反応溶液の調製後は追加の実験操作無しに自律的な形態変化を繰り返すことを可能にした反応を開発した3,4) (図4)。DNAポリメラーゼの導入と適切なエンコードにより、ヘアピン構造の形成とこの部位からの伸長反応、ヘアピン構造の融解、別のヘアピン構造の形成と伸長反応を伸長反応と、一連の形態変化が自律的に繰り返されるようになっている。この方法を応用することで、最初に紹介した一筆書きの実験操作も、経路図の複雑さに依存せずに一回の実験操作で解を得ることが可能になっている5)。
図4 自律的に複数回の構造変化を行うDNA
4.DNAナノ構造に展開したDNAコンピュータ
DNAコンピュータ研究の展開の一つとして、DNAを用いたナノスケールの構造体の作成が挙げられる。情報科学分野では、それぞれ相互作用を規定したタイルによって、繰り返し構造など何らかの意味を持った二次元構造体を形成させるというタイルオートマトン、という概念があった。Winfree らはこの概念を用いたDNAナノ構造体の作成に成功している6)。以来、DNAコンピュータ研究の成果である、互いにホモロジーがなくなるような配列セットの開発や、情報科学者による構造形成シミュレーションなどにより、ナノ構造体の形成技術は着々と進歩している。近い将来に、非周期的DNAナノ構造体上にタンパク質の配置をプログラムした“ナノファクトリー”の構築法の確立が期待されている。
5.生命情報を解析するDNAコンピュータ
冒頭にも述べたように、DNAコンピュータ研究の初期において、電子計算機との競争にアピールの重点が置かれ、天然の生命情報との親和性へのアピールが弱すぎたと言わざるを得なかった。しかし数年前から、東大の陶山らの技術により、「核酸の配列パターンを判定する分子システム」というDNAコンピュータの本質が活用され、SNPパターン判定が行われている7,8)。これらの研究では、生物から抽出したターゲット核酸分子について、その存在量に対応した検出用DNA (DNA coded number, DCN) が生化学的な実験操作によって得られる。さらに、ターゲット核酸分子群について、1本の試験管内の反応でそれぞれに対応したDCN群を得ることも可能で、DCNの量はDNAチップを用いて検出される。さらに、PCRによる天然の遺伝子間での増幅効率は遺伝子ごとに大きく異なることに対し、DCN群は互いにホモロジーが低く、かつ、一様な物理化学的特性を持つ塩基配列を持つため、増幅効率に大きな違いはない。そこで、ターゲット遺伝子配列を持った合成DNAの希釈系列を適宜使用することで、一般的なDNAチップ解析と異なり、多種類の微量ターゲット核酸分子を絶対定量することが可能になっている。
6.生命との親和性を持ち自律的な反応を行う
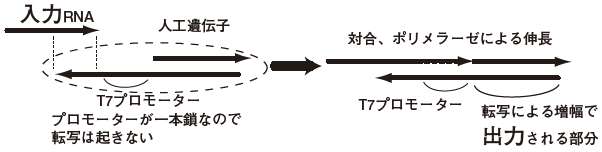
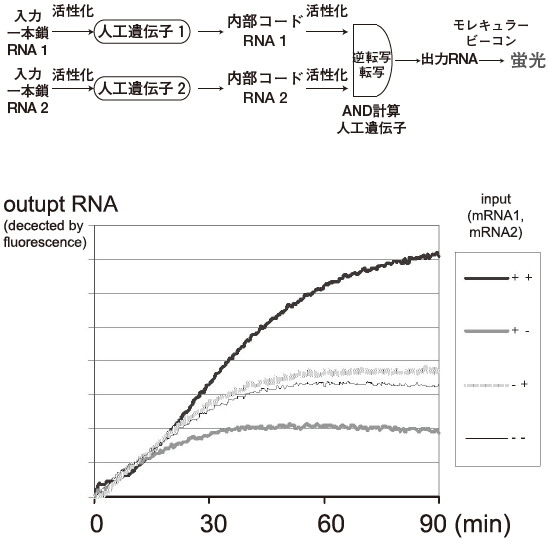
DNAコンピュータ=「試験管内人工生命」最近、陶山と筆者らは、自律的に動作することに焦点を当てたDNAコンピュータを作成している。このシステムの構成要素である「人工遺伝子」に対して、予め対応付けられた特定の部分配列を持った「入力RNA」が存在した場合に、等温条件下で逆転写酵素や転写酵素、分解酵素の連続的な作用が生じ、転写が活性化されてRNAが出力される。
この遺伝子に対する入力RNAと出力RNAの対応関係はDNAに書き込むことで自由に設定可能である (図5)。対応付けられた入力RNAは1種類とすることも2種類とすることも可能であり、結果として、それぞれは、配列の変換、AND論理演算とみなすことができる。

さらに、ある遺伝子の出力RNAが翻訳過程なしに次の遺伝子の入力としてその発現を制御することが可能なため、反応ネットワークを自由にプログラムして構築することが可能なデザインになっている。実際に我々は,3種類の人工遺伝子を組み合わせることで、特定の2種類のRNAが共に存在した場合に,その結果を示す最終産物RNAが生産されるシステムを完成している (図6上)。この産物をモレキュラービーコンを用いて定量することにより、マスターミックスとサンプルを混合した後は一定温度での保温のみで核酸サンプルのパターンを単色の蛍光として判定することが可能となっている (図6下)。このため本システムは今後、「個の医療」に必要な簡便に使用できる遺伝子診断キットとしての展開が期待できる。

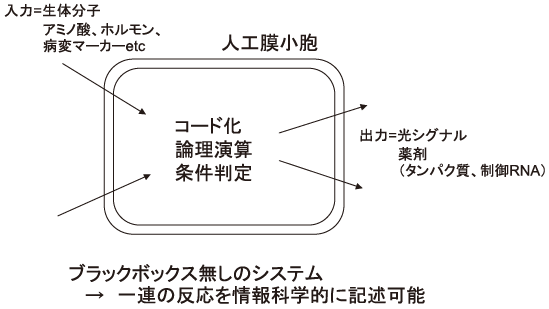
また、本システムの入出力がRNAであることを活かし、天然の生命とのより広い範囲での親和性を持たせる研究が進行中である。たとえば、特定の小分子の存在によって構造変化を生じるRNAであるリボスイッチと組み合わせることで、本システムは小分子存在パターンを判定することが可能となる。また、本システムと無細胞翻訳系を組み合わせることで、システムの出力はタンパク質とすることができるであろう。また、細胞サイズのリポソーム内で生化学反応を進行させる実験手法は各所で確立しつつあり、実際我々も本システムの人工遺伝子を動作させている。これらの成果を統合することで、体内で病変を察知して薬剤を生産するマイクロマシンが将来開発されることが期待される (図7)。
図7 自律的に動作する分子コンピュータの将来像
本研究は、生命をデザインしようとするSynthetic biology (構成的生物学、合成生物学) という分野の一つとして発展しつつある。この分野での多くの研究は、細胞内に人工的な遺伝子ネットワークを構築することを研究の手段としている。その目的は応用と理学の両面、すなわち、一度に複数の遺伝子を導入することでより進歩した遺伝子工学としよう9)、という応用と、このような複数遺伝子の導入を試行錯誤の結果ではなくデザインとして行なうための基礎データの収集とシミュレータの構築という理学的な研究からなっている10-12)。しかし、細胞を用いる限り、大腸菌ですら無視できない数の遺伝子が機能未知となっているような生命のブラックボックス性があるため、細胞の部分システムであってもシミュレーションには未だ限界がある。生の細胞を使用する研究が必要なことも明らかであるが、一度、系の中身が全て判明している生体分子システムに対してシミュレータを構築することも重要であろう。本システムはこのような解析に適しており、生化学研究者から制御科学、情報科学の研究者が集っているDNAコンピュータの研究領域の学際性もあいまって、試験管内で人工生命を構築するためのデザイン論と実践が進んでいる。
7.おわりに
DNAコンピュータの構築は、生命システムの部品を天然とは異なる方式で組み合わせる、という意味において、筆者がこれまで行ったアプタマーの単離13)や21番目のアミノ酸を使用する拡張遺伝暗号系の構築14)と通じるところがある。どの研究も「ありえた生命のかたち」を創ることで知ってみたい、という背景から始まっているが、アプタマーも拡張遺伝暗号系も、その後天然に同等のシステムが存在することが明らかになった。DNAコンピュータと同様のシステムが天然の生命に見つかる日がいつかやって来るかもしれない。
謝辞
DNAコンピューティングの研究を進めるにあたり、陶山明教授、横山茂之教授 (東京大学)、坂本健作博士 (現理化学研究所)、小宮健博士 (現東京工業大学) など、多くの共同研究者のお世話になりましたことを感謝いたします。DNAコンピュータ研究分野を主導している研究者として、特に萩谷昌己教授 (東京大学) を挙げ、御礼申し上げます。本研究の一部は科研費若手研究、特定領域研究“分子プログラミング”、JST先端計測分析技術・機器開発事業の資金によって行われました。最後に、本稿の執筆を紹介してくださいました大阪大学の松浦友亮准教授に感謝いたします。
文献
1) Adleman, L. M.: Science, 266, 1021 (1994).
2) Sakamoto, K., Gouzu, H., Komiya, K., Kiga, D., Yokoyama, S., Yokomori, T., Hagiya, M.: Science, 288, 1223 (2000).
3) Hagiya, M., Arita, M., Kiga, D., Sakamoto, K., Yokoyama, S.: Preliminary Proceedings, 3rd DIMACS Workshop on DNA Based Computers, University of Pennsylvania, 105 (1997).
4) Sakamoto, K., Kiga, D., Komiya, K., Gouzu, H., Yokoyama, S., Ikeda, S., Sugiyama, H., Hagiya, M.: Biosystems, 52, 81 (1999).
5) Komiya, K., Sakamoto, K., Kameda, A., Yamamoto, M., Ohuchi, A., Kiga, D., Yokoyama, S., Hagiya, M.: Biosystems, 83, 18 (2006).
6) Winfree, E., Liu, F., Wenzler, L. A., Seeman, N. C.: Nature, 394, 539 (1998).
7) Nishida, N., Tanabe, T., Hashido, K., Hirayasu, K., Takasu, M., Suyama, A., Tokunaga, K.: Anal Biochem, 346, 281 (2005).
8) Nishida, N., Tanabe, T., Takasu, M., Suyama, A., Tokunaga, K.: Anal Biochem, 364, 78 (2007).
9) Ro, D. K., Paradise, E. M., Ouellet, M., Fisher, K. J., Newman, K. L., Ndungu, J. M., Ho, K. A., Eachus, R. A., Ham, T. S., Kirby, J., Chang, M. C., Withers, S. T., Shiba, Y., Sarpong, R., Keasling, J. D.: Nature, 440, 940 (2006).
10) Pedraza, J. M., Van Oudenaarden, A.: Science, 307, 1965 (2005).
11) Rosenfeld, N., Young, J. W., Alon, U., Swain, P. S., Elowitz, M. B.: Science, 307, 1962 (2005).
12) Ayukawa, S., Kobayashi, A., Nakashima, Y., Takagi, H., Hamada, S., Uchiyama, M., Yugi, K., Murata, S., Sakakibara, Y., Hagiya, M., Yamamura, M., Kiga, D.: IET Synthetic Biology, 1, 64 (2007).
13) Kiga, D., Futamura, Y., Sakamoto, K., Yokoyama, S.: Nucleic Acids Res, 26, 1755 (1998).
14) Kiga, D., Sakamoto, K., Kodama, K., Kigawa, T., Matsuda, T., Yabuki, T., Shirouzu, M., Harada, Y., Nakayama, H., Takio, K., Hasegawa, Y., Endo, Y., Hirao, I., Yokoyama, S.: Proc Natl Acad Sci USA, 99, 9715 (2002).