【トピックス】
ミルクオリゴ糖の代謝に関連するビフィズス菌の酵素
山本憲二、片山高嶺
京大・生命科学、石川県大・生物資源研
1.はじめに
ビフィズス菌は健康な成人や乳幼児の腸管に生息し、善玉菌として整腸作用や抗感染症作用など宿主に良い影響を与えることが知られている腸内細菌である。また、抗アレルギー作用や抗腫瘍作用などがあることも報告されており1)、最近では腸管内の免疫調節機能の点からも注目され、プロバイオティクスとして食品や飲料への添加なども行われている。ビフィズス菌は小腸下部から大腸の腸管に生息する絶対嫌気性の菌で、特異な糖代謝系を有しており、その代謝産物として乳酸よりも酢酸を多く生成する2)。宿主が摂取した糖成分は宿主自身による消化吸収や宿主の消化管上部に生息する腸内細菌によって消費され、ビフィズス菌の生息する消化管下部には腸内細菌が容易に分解し得る糖成分はほとんど届かないので、ビフィズス菌は糖を最大限有効に利用するためのさまざまな糖分解酵素を有している。最近のゲノム解析の結果ではビフィズス菌が持つ全タンパク質の8.5%がオリゴ糖や多糖の分解に関わるタンパク質であることが示されている3)。まさに、ビフィズス菌はさまざまな糖分解酵素 (グリコシダーゼ) の宝庫である。
ビフィズス菌は人工乳で育てられた乳児の腸管に比べて、母乳で育てられた乳児の腸管に優勢に生息することが観察され、母乳中の成分にビフィズス菌を生育させる因子 (ビフィズス因子) があるのではないかと考えられている。
2.母乳中のミルクオリゴ糖
母乳中の糖質成分の95%は乳糖 (ラクトース) であり、残りの数%はミルクオリゴ糖と呼ばれるオリゴ糖である4)。ミルクオリゴ糖は100種類以上存在すると言われているが、主なミルクオリゴ糖は十数種類で、その構造はスフィンゴ糖脂質のラクト系列の糖鎖構造と同じである。すなわち、ラクトース (Galβ1→4Glc) を基本骨格として、ガラクトースとN-アセチルグルコサミンがβ-1,3 結合したラクト-N-ビオース (Galβ1→3GlcNAc)、β-1,4 結合したN-アセチルラクトサミン (Galβ1→4GlcNAc) の2種類の二糖がラクトースに結合し、シアル酸やフコースがそれらのオリゴ糖を修飾する形で結合している。従来より、このようなミルクオリゴ糖がビフィズス菌の生育に重要な因子になっているのではないかと言われている。そこで、我々はミルクオリゴ糖の分解に関わると考えられるビフィズス菌の酵素を解析した。
3.ビフィズス菌が分泌するシアリダーゼ
シアル酸は糖タンパク質や糖脂質などの複合糖質の糖鎖の非還元末端に存在する酸性の糖で、インフルエンザウィルスなど、さまざまなウィルスの受容体となっている5)。ミルクオリゴ糖にも非還元末端にシアル酸が存在するものが多く、母乳は乳幼児のウィルス感染を抑制するものの一つと考えられている。しかし、強く荷電しているシアル酸はオリゴ糖を消化する分解酵素の活性の妨げになる場合が多い。そのためにシアル酸を加水分解する酵素であるシアリダーゼはシアル酸を有するシアロオリゴ糖やシアロ糖鎖の分解に重要な酵素である。我々はBifidobacterium bifidum JCM1254株がシアリダーゼを分泌することを見出し、その遺伝子クローニングと機能解析を試みた。
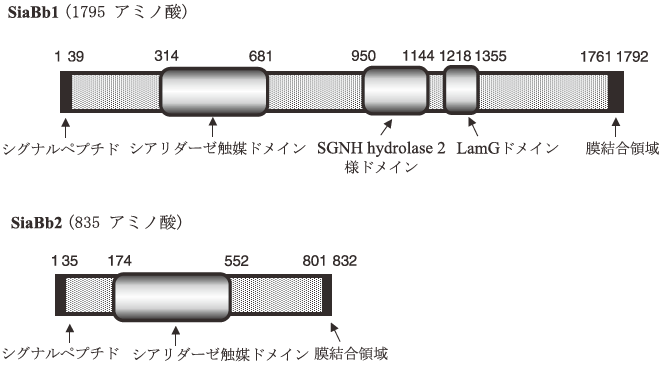
B. bifidumJCM1254株より発現クローニング法によって2種類のシアリダーゼ遺伝子を取得した。そのうちの一つのシアリダーゼSiaBb1は1795アミノ酸残基からなる分子量がおよそ189 kDaのタンパク質で、N-末端側にシグナルペプチド、C-末端側に膜結合領域を持ち、3つの異なるドメインから構成されている (図1)。すなわち、N-末端側からシアリダーゼ触媒ドメイン、SGNH hydrolase 2-likeドメイン、LamGドメインからなっている。触媒ドメインはGlycoside hydrolase (GH) のfamily 33に属する糖質分解酵素の触媒ドメインに相同性があり、細菌のシアリダーゼに特徴的なRIPモチーフやアスパラギン酸ボックス、触媒残基であるAspとTyrが良く保存されていた6)。SGNH hydrolase 2-likeドメインはエステラーゼの触媒部位と相同性があり7)、Clostridium perfringensのリパーゼ/アシルハイドロラーゼやMycoplasma pneumoniaeのリパーゼなどと約25~35%の相同性があった。
図1 ビフィズス菌のシアリダーゼのアミノ酸配列から推定されるドメイン構造
もう一つのシアリダーゼSiaBb2はSiaBb1の遺伝子のおよそ半分ほどの長さの遺伝子を持つタンパク質で835アミノ酸残基からなり、N-末端側にシグナルペプチド、C-末端側に膜結合領域を持つ。中間には触媒ドメインしか存在せず、やはりGH family 33に属する糖質分解酵素と相同性があった。SiaBb1の触媒部位との相同性は70%以上であった。SiaBb2はSiaBb1に比べて著しく高い熱安定性を示した。また、SiaBb2はSiaBb1のシアリターゼ触媒ドメインと基本的に同様な構造を持つ酵素であると考えられる。
4.ビフィズス菌が分泌する1,2-α-L-フコシダーゼ
ヒトの血液型は、赤血球の膜表面にある糖脂質や糖タンパク質の糖鎖が抗原決定基となり、その構造の相違によって決まる。血液型物質は赤血球のみならず、ヒト腸管表面にも分泌性の糖タンパク質ムチンとして発現し、ABO式血液型の抗原決定基を含む分泌性ムチンに腸管は覆われている。α-1,2-L-フコシル基はヒトABO式血液型の型物質のコア成分であるため、ヒト腸管表面には多量に存在することが推察される。このα-1,2-L-フコシル基を有するH (O) 抗原やルイス式血液型抗原が特異的なレセプターとなって、乳児に深刻な下痢症を引き起こすCampylobacter jejuni8)や胃潰瘍の促進因子であるHelicobacter pylori9)は腸管に接着することが明らかになって来ている。一方、母乳中のオリゴ糖の一つである2'-フコシルラクトース (L-Fucα 1-2Galβ1-4Glc) は、その可溶性レセプターアナログとして乳児下痢の発症を抑える働きが知られている10)。
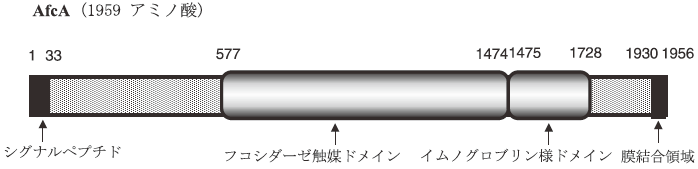
我々は、腸管より単離されたビフィズス菌の多くが分泌性の1,2-α-L-フコシダーゼを特異的に生産することを見出し、本酵素がビフィズス菌の宿主腸管内での生息に関わっているのではないかと推察した。先にシアリダーゼの遺伝子クローニングを行ったB. bifidum JCM1254株は本酵素の強い活性を有していたので、その遺伝子について大腸菌を用いた発現クローニングにより探索した11)。その結果、1,2-α-L-フコシダーゼ遺伝子をライブラリーより単離した。組換え大腸菌で発現した本酵素は1959アミノ酸残基からなる分子量約20万の巨大なタンパク質で、データベース上の機能未知タンパク質と相同性を示したが、いずれの既知のグリコシダーゼとも相同性が見出されない新規な酵素であった。N-末端側にはシグナル配列が、C-末端側には膜結合領域が存在していた (図2)。deletion解析によってその触媒ドメインを同定したが、その一次構造はCl. perfringensやBacteroides thetaiotaomicronなどの腸内細菌由来の機能未知タンパク質と高い相同性を示し、これらは新規なファミリーに属することが確認され、GH family 95が新たに設けられた。
図2 ビフィズス菌の1,2-α-L-フコシダーゼのアミノ酸配列から推定されるドメイン構造
フコシダーゼ活性を担うドメインの下流には、Immunoglobulin (Ig) -likeドメインと呼ばれる保存性の高い領域が存在していた。このドメインは、もともと病原性大腸菌が腸管に侵入する際に重要な役割を果たすタンパク質“インチミン (Intimin) ”に見出された構造であるが、糖鎖構造の認識や糖質関連酵素の構造保持などに関わることが示唆されている12)。およそ80アミノ酸残基からなる相同性の高い繰り返し配列が4回連続して存在し、主にβ-シート構造からなる。その機能は明らかではないが、触媒活性ドメインを菌体細胞壁から長く突き出すことにより腸管のフコシル基への作用を容易にしていることが推察される。また、本酵素はα-1,2-L-フコシル基をもつ母乳中のフコシルオリゴ糖やブタ胃ムチンなどの糖タンパク質糖鎖に良く作用する一方、α-1,3-L-フコシル基やα-1,4、α-1,6 結合には全く作用せず、p-ニトロフェニル-α-L-フコースなどの人工基質にも作用しない。
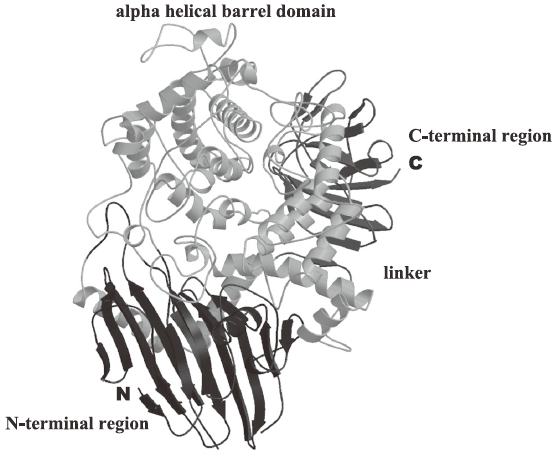
一方、フコシダーゼの活性に必須な領域 (フコシダーゼ触媒ドメイン) について、大腸菌を用いた組換えタンパク質として発現させ、精製して結晶化を行った。その結果、1.12 Åの分解能で立体構造を決定することに成功した。その全体構造はN-末端側から、逆平行β-シートからなるβ-サンドウィッチ領域、4本のα-ヘリックスから成るヘリカルリンカー領域、α-ヘリックスが入り組んだ構造をとるα-ヘリカルバレルドメイン、β-シートが豊富なβ-シートリッチ領域の大きく分けて4つの領域から構成されていた (図3)。変異体の解析結果から、触媒活性を担うのはα-ヘリカルバレルドメイン内に存在するGlu566とAsp766と推測されたが、これらの残基に変異を導入した酵素と基質などとの複合体の構造解析の結果、さらにAsn421およびAsn423の関与も示唆された。
図3 ビフィズス菌の1,2-α-L-フコシダーゼの結晶構造
5.特異な酵素ラクト-N-ビオシダーゼ
Derensy-Dronらは1999年にB. bifidumの菌体内にラクト-N-ビオースを加リン酸分解する酵素ラクト-N-ビオースホスホリラーゼを見出した13)。この事実はビフィズス菌の菌体内にラクト-N-ビオースが酵素の基質として存在する可能性があることを示している。我々はこの二糖がミルクオリゴ糖に由来すると考え、ビフィズス菌がミルクオリゴ糖からこの二糖を切り出した後、菌体内に取り込むのではないかと考えた。そこで、ラクト-N-ビオースをオリゴ糖から加水分解する酵素ラクト-N-ビオシダーゼを検索した結果、ほとんどのビフィズス菌が本酵素を有していることを見出した。一方、Lactobacillus属やLactococcus属などの他の乳酸菌は本酵素活性を示さなかった。我々はシアリダーゼや1,2-α-L-フコシダーゼを持つB. bifidum JCM1254株からラクト-N-ビオシダーゼの遺伝子のクローニングを試みた。
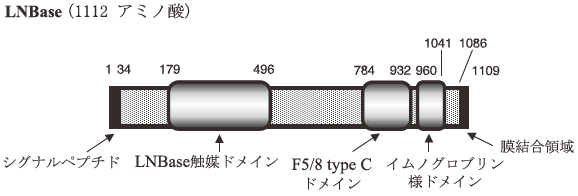
既に遺伝子クローニングが報告されている唯一の酵素であるStreptomyces sp. のラクト-N-ビオシダーゼ14) とLactococcus lactisのputativeなラクト-N-ビオシダーゼ15)の遺伝子との相同配列部分からプライマーを作成し、PCR法とコロニーハイブリダイゼーション法を組み合わせて遺伝子の全長を取得した。得られた遺伝子は大腸菌にて発現し、ラクト-N-テトラオース (Galβ1-3GlcNAc β1-3 Galβ1-4Glc) をラクト-N-ビオースとラクトースに分解することを確認した。本酵素はN-末端にシグナルペプチド、C-末端に膜結合領域を持つ1112アミノ酸残基からなるタンパク質で、GH family 20に属する糖質分解酵素と相同性を持つ触媒ドメイン、ガラクトースへの結合が示唆されるF 5/8 type Cドメイン、それにIg-likeドメインの三つのドメインから構成されていた (図4)。本酵素の活性についてp-ニトロフェニル (p-NP) 基質を用いて調べると、p-NP-ラクト-N-ビオースに高い特異性を示し、p-NP-ガラクト-N-ビオース (Galβ1→3GalNAc) に対してもp-NP-ラクト-N-ビオースの約35%の活性を示した。しかし、天然基質からはガラクト-N-ビオースをほとんど加水分解しなかった。
図4 ビフィズス菌のラクト-N-ビオシダーゼのアミノ酸配列から推定されるドメイン構造
6.ビフィズス菌の特異なミルクオリゴ糖代謝経路
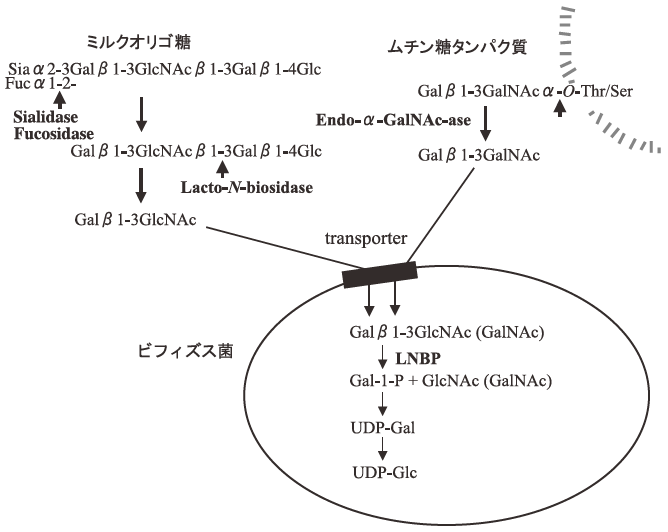
最近、北岡らはB. longum JCM1217株からラクト-N-ビオースホスホリラーゼの遺伝子クローニングに成功した16)。この酵素は次のような反応を触媒する。
Galβ1-3GlcNAc+Pi→Gal-1-P+GlcNAc生成したガラクトース-1-リン酸 (Gal-1-P) はルロワール経路 (Leloir pathway) と呼ばれる代謝系で代謝され、グルコースやガラクトースの糖供与体であるウリジン-ジリン酸グルコース (UDP-Glc) やウリジン-ジリン酸ガラクトース (UDP-Gal) が生成される。すなわち、ビフィズス菌はラクト-N-ビオースを加リン酸分解して、生体内の重要な化合物である糖供与体=糖ヌクレオチドを得ると考えられる。ラクト-N-ビオースホスホリラーゼはラクト-N-ビオースのみならず、ガラクト-N-ビオースにも作用する。我々は腸管の粘膜ムチンに広く存在するムチン型糖鎖=ガラクト-N-ビオースをタンパク質から遊離する特異的なエンド型グリコシダーゼのエンド-α-N-アセチルガラクトサミニダーゼ (Endo-α-GalNAc-ase) をビフィズス菌の多くが分泌生産することを見出している17)。このような事実より、ビフィズス菌ではEndo-α-GalNAc-aseによって腸管のムチンよりガラクト-N-ビオースを遊離し、一方、ラクト-N-ビオシダーゼによってミルクオリゴ糖からラクト-N-ビオースを遊離して、それら二糖を菌体膜に存在するトランスポーターにより菌体内に取り込み、次いでラクト-N-ビオースホスホリラーゼの作用によって糖ヌクレオチド生成経路と繋がると推察される (図5)。したがって、この代謝経路は糖供与体を生合成する経路であると言う点でビフィズス菌の生育のために極めて重要な経路と考えられる。
図5 ビフィズス菌のミルクオリゴ糖代謝の推定経路
7.おわりに
最近、我々はビフィズス菌の菌体膜にトランスポーターを構成するタンパク質でラクト-N-ビオースとガラクト-N-ビオースに特異的に結合するタンパク質を見出し、その遺伝子クローニングに成功するとともにX線結晶構造解析を行っている。すなわち、ミルクオリゴ糖の分解産物であるラクト-N-ビオースはトランスポーターを介してビフィズス菌の菌体内に取り込まれることが示唆され、その代謝経路がほぼ解明できた。ラクト-N-ビオースは他の腸内細菌によってほとんど資化されないと言う事実もあり、この二糖がビフィズス因子として有効なプレバイオティクスとなり得る可能性がある。
謝辞
本研究は食品総合研究所の北岡本光博士、東京大学大学院農学生命科学研究科の伏信進矢博士、高エネルギー加速器研究機構の長江雅倫博士、加藤龍一助教授、若槻壮市教授の多大なご支援とご協力を頂いて行われたもので、厚く御礼を申し上げます。また、本研究の遂行に多大の努力を頂いた京都大学生命科学研究科分子応答機構学分野の多くの学生、研究員に深謝致します。最後に、本稿の執筆の機会を与えて頂きました協和発酵工業の小泉聡司博士に感謝致します。
文献
1) Femia, A. P., Luceri, C., Dolara, P., Giannini, A., Biggeri, A., Salvadori, M., Clune, Y., Collins, K. J., Paglierani, M., Caderni, G.: Carcinogenesis, 23, 1953 (2002).
2) Lauer, E., Kandler, O.: Arch. Microbiol., 110, 271 (1976).
3) Schell, M. A., Karmirantzou, M., Snel, B. D., Vilanova, B., Berger, B., Pessi, G., Zwahlen, M. C., Desiere, F., Bork, P., Delley, M., Pridmore, R. D., Arigoni, F.: Proc. Natl. Acad. Sci. USA, 99, 14422 (2002).
4) Messer, M., Urashima, T.: Trends Glycosci. Glycotech., 14, 153 (2002).
5) Karlsson, K.-A.: Annu. Rev. Biochem., 58, 309 (1989).
6) Crennell, S. J., Garman, E. F., Laver, W. G., Vimr, E. R., Taylor, G. L.: Proc. Natl. Acad. Sci. USA, 90, 9852 (1993).
7) Akoh, C. C., Lee, G.-G., Liaw, Y.-C., Huang, T.-H., Shaw, J.-F.: Prog. Lipid Res., 43, 534 (2004).
8) Ruiz-Palacios, G. M., Cervantes, L. E., Ramos, P., Chavez-Munguia, B., Newburg, D. S.: J. Biol. Chem., 278, 14112 (2003).
9) Applemelk, B. J., Simoons-Smit, I., Negrini, R., Moran, A. P., Aspinall, G. O., Forte, J. G., De Vries, T., Bovin, N. V., Quan, H., Verboom, T., Maaskant, J. J., Ghiara, P., Kuipers, E. J., Bloemena, E., Tadema, T. M., Townsend, R. R., Tyagarajian, K., Monteiro, M. A., Savio, A., De Graaff, J.: Infect. Immun., 64, 2031 (1996).
10) Morrow, A. L., Ruiz-Palacios, G. M., Altaye, M., Jiang, X., Guerrero, M. L., Meinzen-Derr, J. K., Farkas, T., Chaturvedi, P., Pickering, L. K., Newburg, D. S.: Adv. Exp. Med. Biol., 554, 443 (2004).
11) Katayama, T., Sakuma, A., Kimura, T., Makimura, Y., Hiratake, J., Sakata, K., Yamanoi, T., Kumagai, H., Yamamoto, K.: J. Bacteriol., 186, 4885 (2004).
12) Kelly, G., Prasannan, S., Daniell, S., Fleming, K., Frankel, G., Dougan, G., Connerton, I., Matthews, S.: Nat. Struct. Biol., 6, 313 (1999).
13) Derensy-Dron, D., Krzewinski, F., Brassart, C., Bouquelet, S.: Biotechnol. Appl. Biochem., 39, 3 (1999).
14) Sano, M., Hayakawa, K., Kato, I.: J. Biol. Chem., 268, 18560 (1993).
15) Bolotin, A., Wincker, P., Mauger, S., Jaillon, Q., Malarme, K., Weissenbach, J., Ehrlich, S. D., Sorokin, A.: Genome Res., 11, 731 (2001).
16) Kitaoka, M., Tian, J., Nishimoto, M.: Appl. Environ. Microbiol., 71, 3158 (2005).
17) Fujita, K., Oura, F., Nagamine, N., Katayama, T., Hiratake, J., Sakata, K., Kumagai, H., Yamamoto, K.: J. Biol. Chem., 280, 37415 (2005).