【トピックス】
ミスマッチに結合する人工分子
中谷和彦
阪大産研
1.はじめに
私たちは、遺伝子の本体であるデオキシリボ核酸 (DNA) と小さな有機化合物の相互作用を研究してきた。特に、DNAのミスマッチ塩基対にだけ結合する有機化合物を、自分たちの手で設計することを目的としてきた。DNAは4つの構成成分 (核酸塩基) アデニン、グアニン、シトシン、チミンからなる二重鎖の高分子で、通常グアニンはシトシンと、アデニンはチミンとペア (塩基対) を組む。この二つの塩基対をDNAの構造を明らかにしたワトソンとクリックにちなんで、ワトソン-クリック塩基対と言う。これ以外の8種類の組み合わせ、例えば、グアニンとグアニンやシトシンとチミンのペアなどは不安定なペアでありミスマッチ塩基対と言う。私たちは2001年にグアニンとグアニンのミスマッチを特異的に認識する分子の開発に世界で初めて成功した1)。さらにその後、グアニンとアデニンのミスマッチやシトシンとシトシンのミスマッチに特異的に結合する分子を開発してきた2,3)。これらの分子はオーダーメイド医療の実現に必要不可欠な、遺伝子の一塩基多型を調べる化学センサーの心臓部分として利用できる4)。また、この化学センサーの研究中にハンチントン病発症の原因遺伝子である (CAG)nリピート配列がヘアピン二次構造を形成した際に生じるC-G塩基対に挟まれたアデニン-アデニンのミスマッチに我々の化合物が特異的に結合することを発見した5)。本稿ではミスマッチに結合するこれら人工分子の利用について紹介する。
2.ミスマッチ検出化学センサー
高い確率で生じる遺伝子変異は遺伝子多型と呼ばれ、なかでも遺伝子配列の一カ所の塩基だけが変異しているSNPs (Single Nucleotide Polymorphisms) は、個人によって同じ薬が効いたり効かなかったり、また、副作用があったり無かったりする違いを生み出す。個人に最適な治療を施すオーダーメイド医療の実現には、SNPs解析が必要不可欠な技術となる。SNPsを最も直接的に調べるには、遺伝子の塩基配列を端から順番に調べて行けばよい。しかし、一人一人の遺伝子配列を全て調べるには膨大な時間と手間そして多大なコストが掛かるため、疾病に関連するSNPsの塩基がG、A、T、Cの何れであるかを調べるSNPsタイピング手法について、簡単、安価、迅速、そして一度に大量に処理できる方法が研究されている4)。
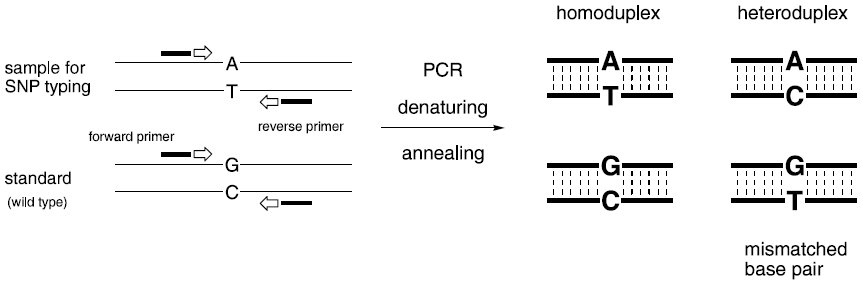
その一つの方法として、SNPsを含む領域のDNAを混合することにより生成するミスマッチ塩基対をもつDNAを、電気泳動などの分析手法や、修復酵素を使って検出するヘテロデュプレックス解析がある (図1)。検査サンプルと標準 (野性型遺伝子) サンプルを同じプライマーを用いてPCR増幅し、熱変成後、二本鎖形成させる。図1の例では、A-T、G-C配列を持つホモデュプレックスとA-C、G-Tミスマッチ塩基対を持つヘテロデュプレックスが生じる。ここで生じたヘテロデュプレックスを検出することにより、検査サンプルにSNPがあったかどうか (ない場合はヘテロデュプレックスが生じない) が判定できる。この手法は原理的に簡単であるため、電気泳動などに代わる、簡便、迅速、安価にミスマッチ塩基対を含むDNAを検出する手法の開発が期待されている。我々はミスマッチ塩基対を塩基特異的に認識する低分子リガンドを世界にさきがけて開発することに成功し、ここで開発されたミスマッチ結合分子リガンド (Mismatch Binding Ligand, MBL) を、表面プラズモン共鳴センサーに固定化した「ミスマッチ検出センサー」を開発した。
図1 ヘテロデュプレックス解析
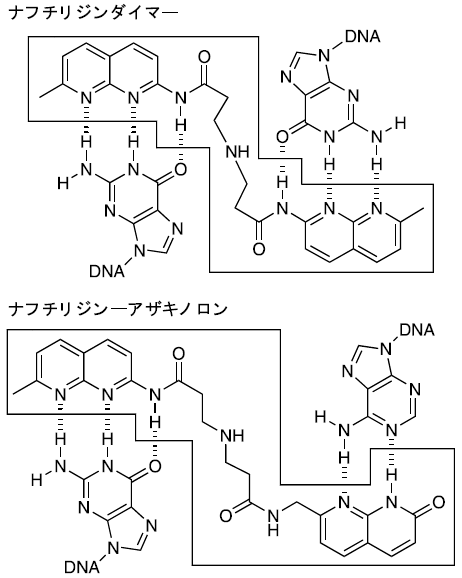
ミスマッチ塩基対の構造は、ミスマッチを構成する塩基、隣接塩基対、塩濃度、温度等の要因が複雑に絡み合い、一義的には決まらない。レセプターのように構造が決まっていてもそこに結合する分子を設計することは難しい。我々はこの問題を避けるため、標的構造を一切無視し、DNA-リガンド複合体構造の「美しさ」を指標として分子設計を進める方針を立て、ミスマッチを構成する核酸塩基と相補的な水素結合表面をもつ2つの複素環からなる分子が、ミスマッチにうまく結合することを見出した (図2)1)。ナフチリジンダイマーはグアニンと結合する複素環 (2-アミノ-1、8-ナフチリジン) を二つ持つ分子でグアニン-グアニンミスマッチと結合し、グアニン-アデニンミスマッチに結合する分子ナフチリジン-アザキノロンはアデニンと結合する8-アザキノロン環を持つ2)。
化学センサーの検出原理には表面プラズモン共鳴 (SPR) を使うことにした。当時所属していた京都大学工学研究科の齋藤研究室では、齋藤教授の「購入する機器は研究に挑戦的であること」との号令のもと、化学者に使いこなすことが難しい機器であることを承知でビアコア社のSPRを購入したが、やはりほとんど稼働していなかった。SPRの購入を推進した筆者には、家一軒程の価格の機器を稼働させていない後ろめたさがあり、とにかくSPRを使うことを考えた。 (注、化学者には使いこなし難いとは、SPRセンサーに固定化する適当な化合物がないという意味です) しかし、結果的にはSPRを自由に使える状況に有ったことが、研究に大きな広がりを与えてくれた。この経験から、異なる分野へ足を踏み入れる際には、自発的な動機より、外的な圧力要因が有効であることを学んだ。図2の分子をビアコア社のチップに固定するには、分子中央の窒素に表面固定化用の短いリンカーをつなぎ、リンカー末端のアミノ基をセンサーチップ上のカルボキシル基とカップリングさせた。
![]()
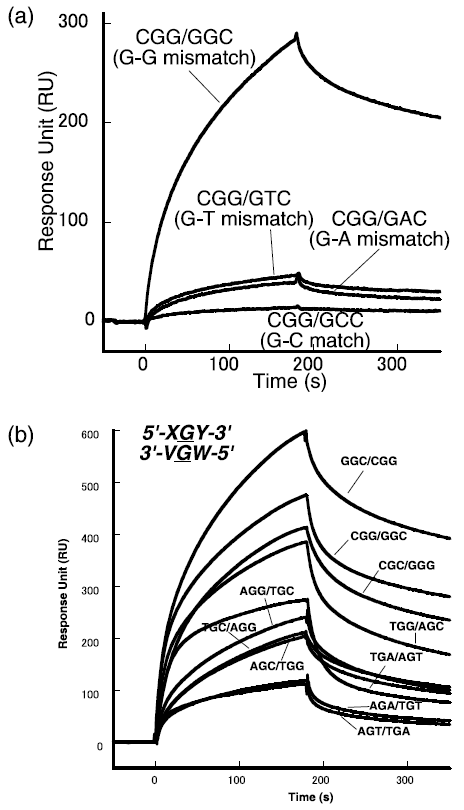
ナフチリジンダイマーを固定化したSPRセンサーで、G-G、G-A、G-Tの各ミスマッチとG-Cマッチの二本鎖を評価した (図3a)。その結果、G-Gミスマッチに対して非常に高いシグナル応答が認められたが、G-A、G-Tの各ミスマッチに対しては弱い応答しか得られなかった。一方、完全なマッチ配列では有意なシグナルが得られなかった。この結果はナフチリジンダイマーをDNAに添加して測定したDNAの融点が、G-Gミスマッチで顕著に上昇した結果を良く反映しており、センサー表面のナフチリジンダイマーとミスマッチの結合挙動がシグナルとして得られてきていることが明らかとなった。続いてミスマッチ前後の配列がナフチリジンダイマーとGGミスマッチの結合へ及ぼす影響を調べるために、G-Gミスマッチ前後の配列10種類全てをSPRセンサーで評価した (図3b)。その結果、G-Gミスマッチ前後の配列がG-Cリッチである場合には、センサーにDNAが強く結合することが示された。一方、前後配列がA-T配列リッチな場合は、DNAの結合が弱くなることが明らかとなった。これらの結果から、1) MBLとミスマッチの結合は前後配列に影響されること、2) 前後配列がGCリッチである場合にリガンドが強くミスマッチに結合することが確認された。言い換えると、MBLはミスマッチだけでなく前後配列を含めた形でミスマッチを認識していることが判る。G-Cリッチな配列での結合が有利な理由として、塩基と水素結合したヘテロ環を隣接G-C塩基対が強いスタッキングにより安定化していることが考えられる。
![]()
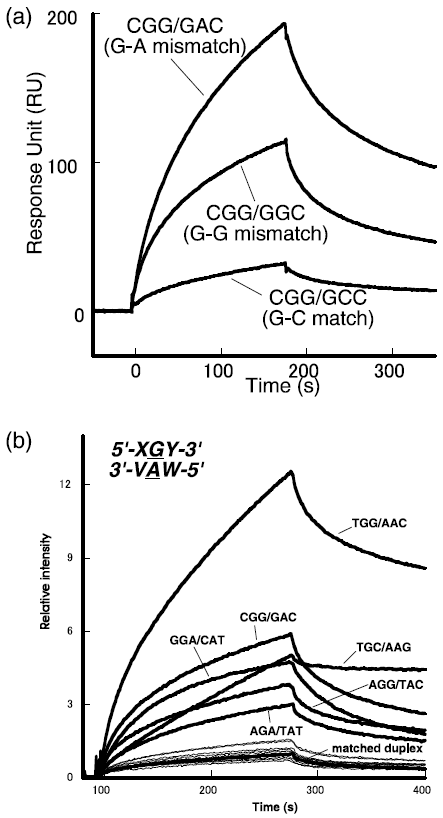
ナフチリジン-アザキノロンを固定化したセンサーでも、おおむねナフチリジンダイマー固定化センサーと同じ傾向が確認された (図4a)。ナフチリジン-アザキノロン固定化センサーの場合、G-AミスマッチだけでなくG-Gミスマッチにもかなりの程度結合することが判った。G-Aミスマッチ前後配列の影響は大きく、16種類のG-Aミスマッチのうち、有意な差で検出できる配列は6種類であった (図4b)。アデニンはグアニンに比べて水素結合が2本しか形成できないため、グアニン-ナフチリジンに比べてアデニン-アザキノロンの結合が弱く、選択性が低下していると考えられる。
![]()
3.リピート配列の長さを調べるセンサー
ゲノムには多くの繰り返し (リピート) 配列が有る。代表的なリピート配列には染色体末端にある (TTAGGG)nが繰り返されるテロメアリピートや、三塩基を一つの組として繰り返されるトリヌクレオチドリピートなどがある。CAGの繰り返し配列 (CAG)nは、このトリヌクレオチドリピート配列の一つで、その繰り返しの回数 (n) が大きくなると、遺伝性の神経変性疾患ハンチントン病を発症することが判っている。CAGリピートは健常者では6~39回程度の繰り返しだが、ハンチントン病を発症した人では最長121回程度にまで伸びていることが知られている。トリヌクレオチドリピート長は、DNA複製時に伸長する可能性があり、その際にはCAGリピートが分子内で形成するヘアピン構造の安定性が重要な要因であると考えられている。トリヌクレオチドリピートにはCAGリピートの他に、CGGリピートやCTGリピートなどがあり、それぞれが異常に伸長すると脆弱性X症候群や筋強直性ジストロフィーの発症に関係している。先に開発していたグアニン-アデニンミスマッチに結合する分子、ナフチリジン-アザキノロンは、このCAGリピートが形成するヘアピン構造に出現するCAG/CAG三つ組み配列に極めて特異的に結合することを発見した5)。ナフチリジン-アザキノロン分子 (NAと略) とCAG/CAGの複合体のNMRによる構造解析を奈良先端科学技術大学院大学の児嶋長次郎博士にお願いし、驚くべき複合体構造が明らかにされた (図5)。複合体は2つのNAと一つのCAG/CAGから構成されていた。CAG/CAGはA-A ミスマッチが二つのC-G配列で挟まれた構造であるが、A-Aミスマッチの二つのアデニンには2分子のNAのアザキノロンが結合すると同時に、両隣にあるG-C塩基対のグアニンにはナフチリジンが水素結合していた。その結果、結合する相手のいなくなったシトシンは、DNA二重鎖の外側に放り出されていた。
ハンチントン病の発症には、CAGリピートの長さが重要な要因となるので、その長さを正確に調べることが診断には大切である。CAGリピートはその繰り返しが伸長するほど折り畳まれやすくなり、その結果アデニン-アデニンミスマッチが多くなる。そのために伸長したCAGリピート程NAは結合しやすくなると考えられる。この原理を利用して、NAを固定化したSPRセンサーを使えばCAGリピートの長さを調べることが出来るのではないかと期待した。NAを固定化したSPRセンサーにCAGリピートを作用させると、CAGリピートがセンサー表面上のNAを介して金表面に捕捉され、その結果として金表面の見かけ上の質量が大きくなり信号が得られる。長さの異なるCAGリピートをNAが固定化されたSPRセンサーに作用させると、リピートの繰り返しが長くなるとそれに応じてSPR信号も大きくなった (図6)。このセンサーを使うと従来の方法よりもはるかに簡便で且つ素早くCAGリピートの長さによるハンチントン病の診断が可能となる。
4.まとめ
ミスマッチをその構成塩基を区別して認識する小分子を使って、遺伝子変異を簡便に調べる化学センサーや、CAGトリヌクレオチドリピートの長さを調べる化学センサーに応用してきた。ここで紹介した以外にも、テロメアリピートに結合する分子や、CGGトリヌクレオチドリピートに結合する分子の開発にも成功している6,7)。一旦出来てしまうといかにも簡単な分子であるが、全く手がかりがないところから探して、なんとかたどり着いたというのが現状である。ゲノムの解読が終わって、プロテオームやトランスクリプトームへ研究の流れはシフトした。我々もDNAからRNAにシフトしつつ有るが、化学的にはたった一つの水酸基の違いであるが、DNAとRNAの違いは予想以上に大きいと感じる。RNAの作る多様な二次構造に特異的に結合する小分子を見つけることが出来れば、きっと面白い研究が出来るはずであり、是非実現してみたい。
本研究は、京都大学工学研究科合成・生物化学専攻齋藤研究室と大阪大学産業科学研究所中谷研究室で行ったものであり、学生、ポスドク諸氏の努力の賜物である。この場を借りて彼らの努力に感謝します。NMR構造解析は奈良先端科学技術大学院大学の児嶋助教授との共同研究の成果であります。齋藤烈先生 (京都大学名誉教授、現日本大学教授) には、永年にわたり研究、教育について御指導御鞭撻を賜り、改めてお礼申し上げます。
文献
1) Nakatani, K., Sando, S., Saito, I.: Nat. Biotechnol., 19, 51 (2001).
2) Hagihara, S., Kumasawa, H., Goto, Y., Hayashi, G., Kobori, A., Saito, I., Nakatani, K.: Nucleic Acids Res., 32, 278 (2004).
3) Kobori, A., Horie, S., Suda, H., Saito, I., Nakatani, K.: J. Am. Chem. Soc., 126, 557 (2004).
4) Nakatani,K.: Chem. Bio. Chem., 5, 1623 (2004).
5) Nakatani, K., Hagihara, S., Goto, Y., Kobori, A., Hagihara, M., Hayashi, G., Kyo, M., Nomura, M., Mishima, M., Kojima, C.: Nat. Chem. Biol., 1, 39 (2005).
6) Nakatani, K., Hagihara, S., Sando, S., Sakamoto, S., Yamaguchi, K., Maesawa, C., Saito, I.: J. Am. Chem. Soc., 125, 662 (2003).
7) Peng, T., Nakatani, K.: Angew. Chem. Int. Ed., 44, 7280 (2005).