【トピックス】
生物はタンパク質分子を上手に使いわける
ー哺乳動物における生物自身によるタンパク質工学ー
岡本 研、西野朋子、西野武士
日医大院・医科生物化学
1.はじめに
HGP (ヒトゲノムプロジェクト) の解析が進むにつれ予想を超えて判明した事実は、遺伝子の数が他の下等な生物に比して必ずしも多くなかったことである。ここで言う遺伝子の数とはタンパク質の設計図の数という意味である。原理的には高等生物のもつ複雑さ多様さは必ずしも部品の数によらなくてもよい。事実、生物の構成原子は極めて限られており、その組合せで多様な物質を多数作り上げている。生物は限られたタンパク質分子を時間的、空間的に使い分け、細胞、臓器、個体という階層性を作り出し、多様な生物学的機能を発揮している。とはいえ高等動物の高次機能には独自の機能も必要である。今回紹介するのは、少ないタンパク質を別の機能に使っている例である。哺乳動物は、他の生物種ではみられない新たなスイッチとリレー系を有しており、生物自身によるタンパク工学を行なっている。
キサンチン酸化還元酵素は分子量30万の巨大な酵素であり、サブユニットあたりFAD、2つの[2Fe-2S]、モリブドプテリンの3種類あわせて4つの酸化還元中心を持つ。この酵素はプリン塩基の分解系で尿酸を生成する。哺乳動物の酵素は、様々な臓器以外に乳汁にも大量に含まれ、NAD+を電子受容体とする脱水素酵素型から酸素を電子受容体とする酸化酵素型へと活性変換を行なう機能を有している。その変換機構は極めて精緻であり、単に酵素が壊れて酸素フラビンの自酸化性が高まった結果とはとても言えない。最近この酵素がプリン塩基の分解系の代謝機能の他に乳汁分泌に機能していることが示唆されている。哺乳動物においてのみ脱水素酵素型から酸化酵素型へ変換する機能が存在しており、哺乳動物にとって極めて象徴的な乳汁分泌の要因となっていることが明らかになりつつある。
2.酵素の分布と代謝機能
キサンチン酸化還元酵素 (xanthine oxidore ductase; XOR) は細菌から高等植物、哺乳動物まで、広範囲の生物種が有している酵素である。本酵素は牛乳に大量に含まれているため、1902年にはアルデヒド酸化酵素として牛乳から抽出され、のちにキサンチン酸化酵素として同定されている1)。以来、金属酵素、フラビン酵素の典型例として多くの研究が行なわれてきた2)。XORの基質特異性は広く、多くのプリン、プテリン、ピリミジン、アルデヒドを水酸化する3)。ヒトにおいてはプリン分解系の最終の2段階を触媒する酵素であり、ヒポキサンチンの2位炭素原子を水酸化してキサンチンに、さらにキサンチンの8位炭素原子を水酸化して尿酸に代謝する。そのモリブデンによる反応機構は最近明確になってきた4)。
XORはNAD+を電子受容体とする大部分キサンチン脱水素酵素 (xanthine dehydrogenase; XDH) として組織中に存在するが、哺乳動物の酵素は酸素を主な電子受容体とするキサンチン酸化酵素 (xanthine oxidase; XO) へと変換する (IUBの規定では酸素分子を電子受容体とするものを酸化酵素、その他の物質を受容体とするものを脱水素酵素と定義する)5)。XOはNAD+との反応性を失う一方で、酸素を還元してスーパーオキサイドアニオン (O2-)、過酸化水素を生ずる。このときXOにより産生されるこのような活性酸素種による組織障害が虚血再還流障害をはじめとする種々の病態生理の原因であるとする指摘が多数ある6-8)。乳汁中では他の組織とは異なりXOとして存在すること、XORのノックアウトマウスでは乳腺細胞内の脂肪滴の蓄積、子の発育異常などが観察されることから、脱水素酵素から酸化酵素への変換は乳汁分泌に必要なプロセスであると推測されている9)。
3.ウシXORの結晶構造
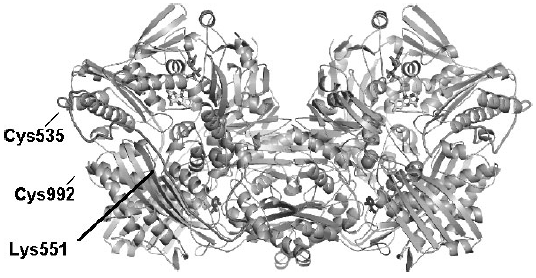
ウシ乳由来のXORの結晶化の報告は古いが10)、構造解析に足る質の良い結晶は永年得られなかった。我々は精製法に改良を加え11)、さらに温和な条件下で試料から脂質を取り除く処理 (高純度リパーゼ処理) をすることにより質の良い結晶を作ることに成功した12)。このことによりウシXORの結晶構造をXDHとして2.1 Å、XOとして2.5 Åで決定することができた13)。決定されたXORの結晶構造は155×90×70 Åの蝶型をしており (図1)、モリブドプテリンドメインを介して2個のサブユニットが結合している。一つのサブユニットは結晶構造上でも3個のドメイン構造にわけられている。それぞれのドメイン間は分子表面を走行するループで連結されており、プロテアーゼによる切断部位はこのループ上に位置する。さらにラットの脱水素酵素型変異体 (常に脱水素酵素であるように3つのシステインをセリンに置換した酵素) の構造を解いたが、基本的にはウシ脱水素酵素のそれと基本的に同じである14)。
![]()
4.酵素反応機構と分子内電子伝達
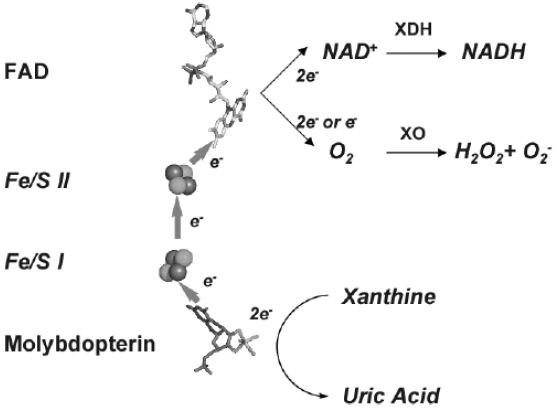
酸化還元中心は直線上に配置されており、モリブドプテリン、Fe/S I、Fe/S II、FADの順である (図2)。モリブデンでの基質水酸化の際に電子はモリブデンに渡され3)、モリブデンに渡った電子はFe/S I、Fe/S IIの順に経由してFADに渡され、その還元型からNAD+あるいは酸素分子に渡される。モリブデンとFe/S I間の距離は15.0 Åあるが、中間にはプテリン環が存在しており、プテリン環はモリブデンの保持だけで無く、電子伝達にも関与していると思われる。Fe/S IとFe/S IIの距離は12.5 Åである。結晶構造では中間に水分子が観察されており、電子は周囲のアミノ酸やこのような水分子を介して伝達されると思われる。酸化還元中心間の距離ではFADとFe/S IIがとくに接近しており、7.8 Åしか離れていない。最も接近しているのは7位のメチル基であり、中間にはGly46が存在する。この距離の近さは、酵素反応における両者の密接な関係を示唆している。それぞれのサブユニット間の酸化還元中心は約50 Å離れており、お互いのサブユニットが酵素活性において相互作用を及ぼしているとは考えがたい。
![]()
先に述べたとおり、酵素の反応中心は4つであるが、酵素の反応中心は完全還元型ではモリブデンに2個、2つの非ヘム鉄のそれぞれに1個、FADに2個あわせて6個持ちうる。ストップドフローを用いた実験によると、脱水素酵素の場合はターンオーバー中の酵素の還元状態は2電子、4電子間を行き来している15)。最終電子受容体であるフラビンは2電子還元型のFADH2と1電子還元型であるセミキノンをとりうる。FADH2と酸素またはNAD+との反応は速く、それぞれ過酸化水素またはNADHを生成する。セミキノンと酸素との反応性は低く酸素と反応してO2-を生成する16)。脱水素酵素におけるFADはセミキノン状態をとっているものが多い。この際NAD+と結合した場合はFADの電位が上昇し17)、隣接しているFe/S IIより電子が流れ込み、FADH2が生じ、2電子受容体であるNAD+の還元がスムーズに行なわれると考えられる。このようにFe/S IIは電子の中継だけでは無く、FADの反応性を制御する“electron sink”として機能していると考えられる17,18)。
一方、XOでは1電子、3電子状態が優位であり、なおかつFADの電位が変化しているため、ターンオーバー中にFADH2が多く存在することとなる。FADH2は酸素分子との反応性が高いので、容易に酸素分子の還元が起こり、過酸化水素を生成する。また、以下に述べるように構造上も酸素分子のFADに対するアプローチがより容易になっている。種々の還元状態のXORを酸素と反応させ、生成された活性酸素種を観察すると、セミキノンを持つ酵素との反応ではO2-が、FADH2を持つ酵素との反応ではH2O2が生じる8)。このことよりXDHではO2-のできる比率はXOより高い。しかし、実際の細胞内ではNAD+が存在し、XDHはNAD+と優先的に反応するため、酵素との反応は起らないと考えられている5)。すなわち、活性酸素を生成するにはXOへの変換が必須である。
5.活性変換に関わる残基
脱水素酵素型から酸化酵素型への変換を引き起こす要因は酵素タンパク質の2つの分子内ジスルフィド結合の形成、またはタンパク質分解酵素による部分切断である。ウシ、ラットXDHの化学修飾19,20)、ラットXDHの変異酵素を解析した結果14)より活性変換をおこすのはCys535とCys992の間のジスルフィド結合の形成であると考えられる (図1)。
プロテアーゼによる分解ではラット酵素はトリプシンによりLys551か21)、ウシ酵素はパンクレアチン (膵臓の抽出液) によりLys569が22)切断されることにより、不可逆的にXOに変換する。
これら活性変換に関わる残基はFADとモリブデンのドメインを連結するリンカー部分に集中している (図1)。即ち、リンカーそのものの移動が引き金と考えられる。なおCys535とCys992のジスルフィド結合がNAD+を抑えることが知られている16)。
6.活性変換に関わる分子スイッチとリレー系
プロテアーゼ切断部位であるLys551とLeu569がFADとモリブデンのドメインを連結するリンカー上に位置しており、XOではこのリンカーが消失していることは先に述べた。ジスルフィド結合を形成して活性変換に関わるCys535とCys992の距離は推定で15 Åであり、結合するには遠い。おそらく可動性の高いリンカー上に存在しているCys535の方がCys992に接近すると考えられる。その際、Cys535が存在しているリンカーは大きく位置移動をすると考えられる。
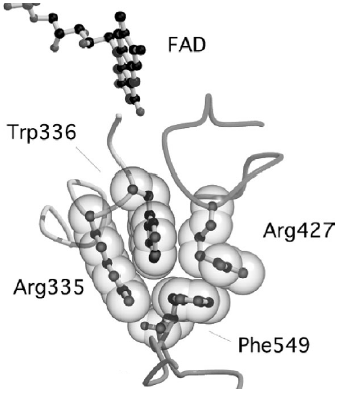
このリンカーの移動が活性変換を起こすとしても、基質結合部位とは酵素分子の反対側であり、最も近いところでも16 Å離れている。これら遠くの部位の変化が直接FAD周囲の環境の変化を起こすとは考えがたく、さらなる構造変化の連鎖が存在すると考えられる。リンカー上の残基ではPhe549が活性中心ループを構成するArg427と近接しているのが特徴である。我々はコンフォメーション変化の連鎖がPhe549を介すと考えた。Phe549付近を詳細に観察すると、Arg427の他にもArg335、Trp336を交えた緊密なクラスターの存在が分かった (図3)16)。それぞれの残基の側鎖はファンデルワールス相互作用、π-カチオン相互作用を取りうる様にパッキングされており、さらにArg335とPhe549、Glu332が水素結合を形成している。この特徴的なクラスターはXOでは大きく異なった形をとっている。XOではリンカー上のPhe549が消失している。それに伴って残基間の相互作用が消失し、Trp336の側鎖が90°スイングして、Arg335、Trp336、Arg427の間隔は広がっている。このクラスターに属する残基を変異させた酵素で活性変換の解析を行ない、これら3残基を分子間力の少ないアラニンに変換したところ、いずれの変異酵素でもXDH、XO間の活性変換は著しく阻害され、XO型の活性を持つようになった16)。活性変換を起こさないRhodobacter capsulatus のXORの結晶構造をKisker らのグループが分子置換法により決定したが22)、この酵素ではドメイン間のリンカー部が最初から存在しない。さらにTrp336に相当する残基がアルギニンであり、周囲残基と多数の水素結合を形成していて、哺乳動物のトリプトファンのようにスイングして周囲のコンフォメーション変化を起こすことは考えがたい。すなわちそのようなシステムは持っていない。
図3 πカチオン相互作用により形成されるアミノ酸クラスター
XOではクラスターの解離が起きるのに伴い間隙が生じ、この間隙は分子を貫通してFADにまで達している。そして、このとき新たに溶媒に露出をするのが酸素とアダクトを形成するC4位であり、人工フラビンを用いた実験結果とよく一致する。XOにおいては新たに生じたこの裏口ともいうべき溶媒チャンネルの形成がFADに対する酸素分子の接近経路であると考えられる。逆に、XDHではクラスターがこの間隙を閉鎖し、酸素分子との反応を抑制する原因の一つとなっていると考えられる16)。
7.結晶構造でのXDHとXOの違い
XDHとXOとのX線結晶構造上の違いは主にドメイン間を結合するリンカー部分である。XOでは中間のドメインとC端のドメインを連結する529~570間のループが約70 Åの長さに渡って消失している。
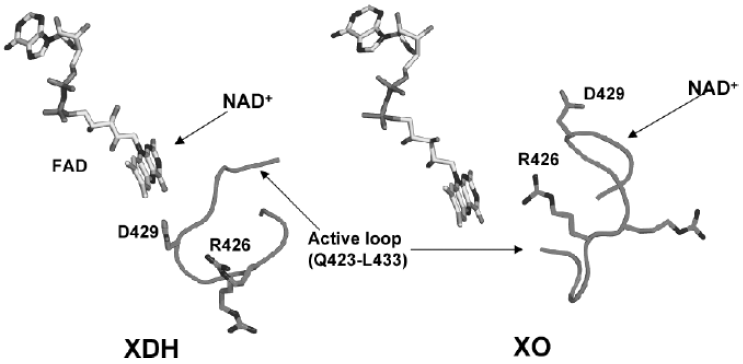
もう一つの大きな違いはFAD近傍の423~433のループである。XDOにおいてはこのループはNAD+結合部位とは反対側に存在していて、XOにおいてはこのループがNAD+結合部位を埋めるようにFADの前面に移動している (図4)。XOにおいてNAD+との反応性が消失するのは、このループの移動による基質結合部位の消失がその理由である。このループ上に存在するGlu426はXDHではFADのC6に近接している。しかし、XOではGlu426に替わってArg429が同部位に接近する。このことはFAD周囲の静電的環境をドラマティックに変化させる。このFAD周囲で認められた残基の再配置は後述する人工フラビンと入れ替えた酵素の解析結果と良く一致する。
図4 キサンチン脱水素酵素/酸化酵素のFAD周囲構造の違い
それ以外のフォールディングはXDH、XO間で良く一致している。特に鉄硫黄中心とモリブドプテリンを含むドメインにはほとんど構造的な差が無い。このことは酵素学的にも確かめられており、これらドメインに含まれる酸化還元中心の電位に変化は無く、モリブドプテリンで触媒される反応についてもXDH、XO間でキネティカルに差が無いという観察結果と一致する。
8.XDHとXOでのFAD周囲のタンパク環境の違い
熱力学的、分光学的にXDHとXO間ではFADの性質が大きく異なる。ウシ酵素のFAD/FADHカップルの酸化還元電位は-270 mV であり、FADH・/FADH2では-410 mVである23)。他の生物種のXDHでもおおむねこの値に近い。これは熱力学的にFADの一電子還元型であるセミキノンが安定であることを示しており、実際に酵素の還元途中、あるいは酸素とのターンオーバー中にはセミキノンに由来する吸収スペクトルが明確に観測される。一方XOではFAD/FADHが-332 mVであり、FADH・/FADH2が-234 mVであり24)、2電子還元型のFADが安定である。実際にXOの還元、ターンオーバー中にセミキノンは少量しか観察できない。
人工フラビンを通常のFADと置換し、活性中心付近の静電的環境、溶媒への露出度を探るプローブとして用いた一連の実験によりXDHとXO間でのFAD周囲の環境の違いが明らかにされている。8-SH FADのpKは3.8と低く、FADの代わりに酵素に組み込むと多くの場合アニオン型であり、XOの場合もこれに相当する。しかしXDHに組み込んだ場合はプロトン化され、吸収スペクトルの明確な変化を認める25,26)。XDHに組み込んだ場合は、入れ替えた8-SH FADのpKは4単位以上も下降していると考えられる。このことはXDHのFAD周囲にプロトン供与基が存在し、そのことがFADの青色セミキノンの安定性に強い影響を与えていることを示している27)。さらに、XOではFADの4a位、6位が溶媒に露出しているが、XDHでは埋もれていることが観察された16)。4a位は酸素分子の還元の際に直接関与する部位であり、この部位が溶媒に露出していることはXOにおける酸素との高い反応性の理由と考えられる。これら人工フラビンを用いた観察結果から示唆されたFAD周囲の環境の変化は前述したXDH、XOの結晶構造の相違点ときわめて良く一致する。
9.おわりに
以上述べたとおり脱水素酵素から酸化酵素への変換機構は極めて理にかなった精緻な機構を取っているといえる。この変換は最近牛乳に存在する酵素によりおこることがわかって来た。すなわち、牛乳はシステムとして脱水素酵素から酸化酵素への変換機構を有しているということである。哺乳動物では生物自身のタンパク工学により、プリン分解という代謝機能に加えて哺乳動物での別の生理機能を有していることを示唆している。現在分子レベルの知見を基に、そのことを証明するため生物学的検討を進めている。
文献
1) Schardinger, F.: Z. Untersuch. Nahrungs Genussmittel, 5, 1113 (1902).
2) Massey, V., Harris, C. M.: Biochem. Soc. Trans., 25, 750 (1997).
3) Hille, R.: Chem. Rev., 96, 2757 (1996).
4) Okamoto, K., Matsumoto, K., Hille, R., Eger, B. T., Pai, E. F., Nishino, T.: Proc. Natl. Acad. Sci. USA, 101, 7931 (2004).
5) Nishino, T.: J. Biochem., 116, 1 (1994).
6) McCord, J. M.: N. Engl. J. Med., 312, 159 (1985).
7) 西野武士: 蛋白質核酸酵素, 34, 1978 (1989).
8) Hille, R., Nishino, T.: FASEB J., 9, 995 (1995).
9) Vorbach, C., Scriven, A., Capecchi, M. R.: Genes Dev., 16, 3223 (2002).
10) Avis, P. G., Bergel, F., Bray, R. C., Shooter, K. V.: Nature, 173, 1230 (1954).
11) Nishino, T., Nishino, T., Tsushima, K.: FEBS Lett., 131, 369 (1981).
12) Eger, B., Okamoto, K., Enroth, C., Sato, M., Nishino, T., Pai, E. F., Nishino, T.: Acta Crystallogr. Sect. D Biol. Crystallogr., 56, 1656 (2000).
13) Enroth, C., Eger, B. T., Okamoto, K., Nishino, T., Nishino, T., Pai, E. F.: Proc. Natl. Acad. Sci. USA, 97, 10723 (2000).
14) Nishino, T., Okamoto, K., Kawaguchi, Y., Hori, H., Matsumura, T., Eger, B. T., Pai, E. F., Nishino, T.: J. Biol. Chem., 280, 24888 (2005).
15) Schopfer, L. M., Massey, V., Nishino, T.: J. Biol. Chem., 263, 13528 (1988).
16) Kuwabara, Y., Nishino, T., Okamoto, K., Matsumura, T., Eger, B. T., Pai, E. F., Nishino, T.: Proc. Natl. Acad. Sci. USA, 100, 8170 (2003).
17) Nishino, T., Nishino, T.: J. Biol. Chem., 264, 5468 (1989).
18) Olson, J. S., Ballou, D. P., Palmer, G., Massey, V.: J. Biol. Chem., 249, 4363 (1974).
19) Nishino, T., Nishino, T.: J. Biol. Chem., 272, 29859 (1997).
20) Rasmussen, J. T., Rasmussen, M. S., Petersen, T. E.: J. Dairy Sci., 83, 499 (2000).
21) Amaya, Y., Yamazaki, K., Sato, M., Noda, K., Nishino, T., Nishino, T.: J. Biol. Chem., 265, 14170 (1990).
22) Truglio, J. J., Theis, K., Leimkuhler, S., Rappa, R., Rajagopalan, K. V., Kisker, C.: Structure (Camb.), 10, 115 (2002).
23) Hunt, J., Massey, V., Dunham, W. R., Sands, R. H.: J. Biol. Chem., 268, 18685 (1993).
24) Porras, A. G., Palmer, G. J.: J. Biol. Chem., 275, 11617 (1982).
25) Massey, V., Schopfer, L. M., Nishino, T., Nishino, T.: J. Biol. Chem., 264, 10567 (1989).
26) Saito, T., Nishino, T., Massey, V.: J. Biol. Chem., 264, 15930 (1989).
27) Ghisla, S., Massey, V.: Biochem. J., 239, 1 (1986).