【トピックス】
NMRによるタンパク質構造解析の展望
ーMusashiタンパク質によるRNAの認識機構の解析を例にー
宮ノ入洋平、片平正人
横浜市大・国際総合科学
1.はじめに
タンパク質や核酸などの生体高分子の機能を知る上で、それらの立体構造は、非常に重要な情報を与えてくれる。生体高分子の立体構造を解析する手法としては、主にX線結晶解析とNMR法の2つが挙げられる。NMR法の特徴として、生体高分子を溶液状態で解析することができる点が挙げられる。このことは、生体内環境に近い状態で生体高分子の構造情報が得られることを意味している。ここでいう構造情報というものは、我々が普段目にする、固定された立体構造という静的な性質だけでなく、運動性や構造多形性といった動的な性質も含まれている。動的な構造情報は、酵素反応や基質認識といった生体高分子の機能発現に関連しており、非常に重要な因子である。
一方で、NMR法では解析できる分子量に限界があるため、30 kDa を超えるような分子に関しては、分子認識などの生命活動を十分に議論するだけの高分解能な立体構造を得ることは、簡単ではない。しかしながら、TROSY法や部位特異的な安定同位体標識法の開発などにより、近年では50 kDa を超える分子複合体についても、立体構造の解析が可能となってきた。
本稿では、RNA結合タンパク質であるMusashiタンパク質を例に、NMRを用いた立体構造解析や分子間相互作用、運動性の解析について紹介したい。
2.Musashiタンパク質
まず、Musashiタンパク質の名前であるが、これはショウジョウバエを用いた研究に由来している。野生型ショウジョウバエでは、一個の外感覚器から、一本の感覚剛毛が生えているが、Musashiを欠失させた変異ショウジョウバエでは、一個の外感覚器から二本の感覚剛毛が生えていた1)。このような表現型から、二刀流の宮本武蔵になぞらえて、Musashiタンパク質と名づけられた。
Musashiタンパク質は、線虫やホヤなどの無脊椎動物からヒトに至る様々な種において保存されており、細胞の運命決定や発生経路に影響を与えると考えられている。実際に、マウスMusashiタンパク質 (mMSI) については、神経前駆細胞の分化を促進するnumbタンパク質のmRNAに結合することが明らかにされた。さらに、この結合によってmMSIはnumb mRNAの翻訳を抑制し、その結果分化を抑制して、神経前駆細胞の自己再生を促すことが明らかにされた2)。
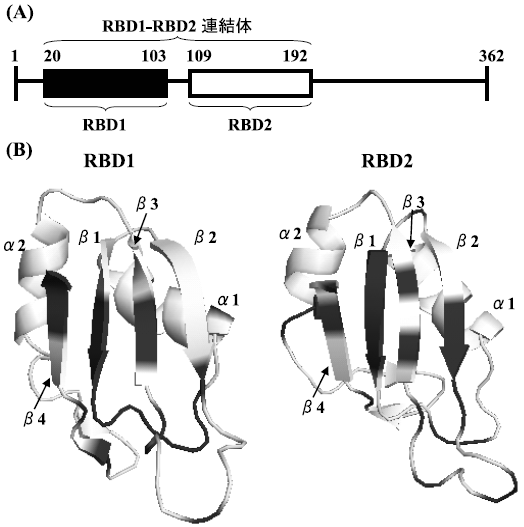
mMSIは362個のアミノ酸によって構成されており、Ribonucleoprotein (RNP) 型のRNABinding Domain (RBD) を2つ (N末側よりRBD1, RBD2と命名) 有している。mMSIは、これら2つのRBDを用いてr([G/A] U1-3 AGU) といったRNAの塩基配列を、特異的に認識することがin vitroの実験で明らかとなった2)。そこで、上記の認識配列を有する標的RNA r(GUUAGUUAGUUAGUU) を設計し、RBD1(CYS20-LYS103) とRBD2(LYS109-SER191) について標的RNAとの相互作用をゲルシフト法によって解析した。RBD1とRBD2のアミノ酸配列の相同性は76%であり、両者は非常によく似た性質を有していると考えられていた。しかしながら、ゲルシフト解析の結果、RBD1はRBD2よりもはるかに強いRNA結合能を有していることがわかった。また、RBD1-RBD2連結体 (CYS20-SER191) についても、同様のゲルシフトの解析を行った。その結果、RBD1-RBD2連結体は、RBD1単独の時よりも、さらに強い結合能を有していることが明らかにされた3)。これは、RBD1-RBD2連結体において、RBD1とRBD2が協調的に働いていることを示唆している。我々は、まずRBD1 とRBD2のRNA結合能の差を解明するために、各ドメインについて立体構造の解析、標的RNAとの相互作用解析、更に運動性の解析を行った。また、RBD1-RBD2連結体と標的RNA複合体についても立体構造および相互作用の解析を行い、RNAの認識機構の解明を目指した。
3.RBD1とRBD2のRNA結合能の差
3-1 各RBDの立体構造
RBD1とRBD2はアミノ酸配列が非常によく似ているにもかかわらず、RNA結合能に関しては大きく異なる性質を示している。我々は、このRNA結合能の差を解明するために、各RBDについて様々な解析を行った。
まず、各RBDについて、NMRを用いて立体構造の解析を行った。RBD1およびRBD2について15N 標識されたサンプルを調製し、NOESYやTOCSYといったNMR測定を行った。NOESYでは空間的に距離が近い (5 Å以内) 水素原子間においてシグナルを得ることができる。一方、TOCSYにおいては、化学結合を介した水素原子間においてシグナルを得ることができる。このような特徴を基に、水素原子間の距離情報や二面角情報を抽出していき、構造解析に利用した。両ドメインの立体構造は、反平行型のβシートが2本のαへリックスによって裏打ちされた、RNP型に特徴的なフォールドを示していた3,4)(図1)。つまり、RBD1とRBD2は基本的には、よく似たフォールドを保持しており、立体構造の面からだけでは、両ドメイン間のRNA結合能の違いを見出すことはできなかった。そこで我々は、各RBDについて標的RNAとの相互作用領域を特定し、その領域における物性を詳細に解析することにした。

3-2 各RBDの標的RNAとの相互作用領域
まず、相互作用領域を特定するために、NMRを用いて、化学シフト変化の解析を行うことにした。この解析では、15N標識した各RBDに標的RNAを少しずつ滴定していき1H-15N HSQCを測定する。1H-15N HSQCでは、タンパク質主鎖のアミド窒素とアミド水素との間の相関を得ることができ、ひとつのシグナルがひとつのアミノ酸に対応している。標的RNAとの結合に関わるアミノ酸は、標的RNAの滴定に伴って、磁気的な環境が変化するために、シグナルの位置 (化学シフト) が変化していく。したがって、化学シフトの変化が大きいところを、相互作用領域として同定することができる。標的RNAの滴定に伴い、RBD1とRBD2では、共にβシート領域において大きなシフト変化が見られていた (図1)。したがって、両ドメインとも、βシートを利用して標的RNAと相互作用していることが分かった。そこで、両ドメインのシート領域に注目し、その物性を解析することにした。
3-3 各RBDの表面静電ポテンシャルの解析4)
まず、分子表面の電荷分布を調べた。各RBDの立体構造を基に、解析ソフトGRASP5)を用いて分子表面の静電ポテンシャルを算出した。その結果、RBD1のβシート領域ではLYS残基やARG残基の側鎖に由来する正電荷が多く存在していることがわかった。一方で、RBD2のβシート領域には顕著な電荷分布は見られておらず、電気的に中性であった。表面静電ポテンシャルは、RNAとの静電相互作用に大きな影響を与える。RNAは、その骨格にリン酸基を有しており、基本的には負に帯電している。RBBD2とは異なり、RBD1のβシート領域は正電荷が集中していることから、RNAと静電相互作用をするのに有利だと考えられる。このような、相互作用領域における表面静電ポテンシャルの違いが、RBD1とRBD2のRNA結合能の違いを生み出していることが考えられた。
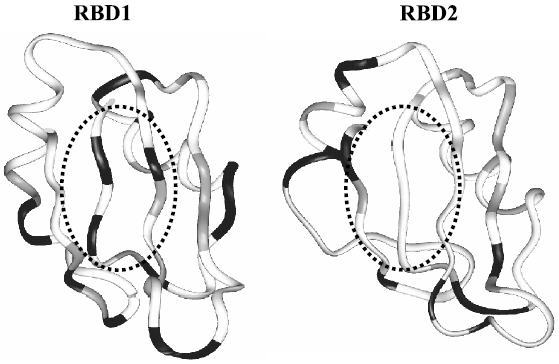
3-4 各RBDの運動性の解析4)
ここまでの解析は、タンパク質の静的な側面しか議論しておらず、溶液状態で働くタンパク質の機能を詳細に議論するには、情報が乏しい。そこで、我々は各RBDについて運動性の解析を行い、動的な観点からRNA結合能の差を解明することを目指した。
タンパク質の分子内運動の情報を得るためには、緩和時間等の測定が必要となる。我々は15N標識されたRBD1およびRBD2について、タンパク質主鎖のアミド窒素を対象に緩和時間等の測定を行い、タンパク質主鎖の内部運動の解析を行った。NMRの測定によって、アミド窒素のT1,T2およびアミド水素とのNOEといった3種類の情報を得ることができる。次に、これらの情報を用いて、モデルフリー解析6)というものを行う。この解析では、タンパク質主鎖のアミド窒素とアミド水素の結合を一つのベクトルとみなし、ベクトルの動くことのできる範囲や動きの速度を算出し、内部運動を記述している。
mMSIの各RBDについて、緩和時間等の測定を行い、モデルフリー解析を行ったところ、以下のようなことがわかった。まず、RBD1については、βシート中央部において、ミリ秒およびナノ秒オーダーの内部運動が存在していた。一方、RBD2のβシート中央部には、そのような運動は検出されなかった (図2)。βシートの中央部には、両ドメインともに3つのPHE残基によって形成された疎水パッチ構造が形成されており、RNA塩基の認識に重要な役割を担っていることが知られている。このような相互作用領域における運動性は、標的RNAを認識する上で重要な役割を果たすと考えられる。実際に、mMSIと同様なRNP型のRNA結合タンパク質であるAUF1タンパク質やU1Aタンパク質のRBDは、タンパク質単体時には、相互作用領域において、ミリ秒オーダーの運動性を有していた7,8)。しかし、標的核酸と結合して複合体を形成すると、その運動性は抑えられていた。このことから、相互作用領域における運動性は、誘導適合を引き起こして標的核酸との強固な結合を生み出していると考えられる。mMSIのRBD1にみられる相互作用領域の運動性も、標的RNAの認識に有利に働いている可能性がある。

RBD1とRBD2はアミノ酸配列や構造はよく似ているにもかかわらず、RNA結合能に大きな差がみられていた。この差は、相互作用領域における表面静電ポテンシャルや、運動性の違いによって生み出されると示唆された。
4.RBD1-RBD2連結体とnumb 15merとの相互作用
さて、RBD1-RBD2連結体はRBD1単独の時よりも、さらに強いRNA結合能を有している。このことから、RBD1-RBD2 連結体においては、RBD2も標的RNAとの相互作用に関わっていることが考えられる。つまり、単独ではゲルシフトで検出されなかったRBD2のRNA結合能が、連結体においては有意な貢献をなしている事になる。このような現象は如何にして生み出されているのであろうか?この疑問を解明するため、我々は、RBD1-RBD2連結体と標的RNAとの相互作用様式を解析することにした。
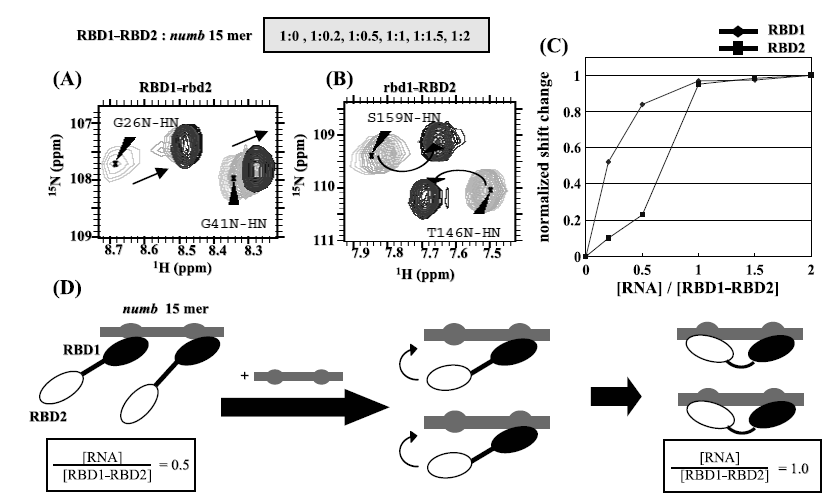
まず我々は、15N標識されたBRD1-RBD2連結体を調製し、前述した化学シフト変化法を行うことによって、標的RNAとの相互作用の解析を行うことにした。標的RNAとしては、numb mRNAから設計したnumb 15mer [r(UAGGUAGUAGUUUUA)] を使用した。このnumb 15merには、mMSIの認識配列であるGUAGUという配列が、2つ重なるように存在している。RBD1-RBD2連結体にnumb 15merを滴定していくと、各RBDのβシート領域とRBD1とRBD2の間のリンカー領域のアミノ酸において、大きな化学シフト変化がみられた。このことからRBD1-RBD2連結体では、βシートとドメイン間リンカーを介して、RNAと相互作用していることが示唆された。さらに化学シフトの変化を詳細に解析したところ、興味深い現象が見られた。
今回の実験では、RBD1-RBD2連結体に対して、numb 15merをモル比で1:0, 1:0.2, 1:0.5, 1:1, 1:1.5および1:2となるように滴定していった。RBD1-RBD2連結体のRBD1領域においては、1:0から1:0.5において大きく化学シフトが変化していた。しかし、それ以上RNAを加えても、化学シフトに変化がみられなかった (図3A)。一方で、RBD2領域においては、1:0から1:0.5までRNAを加えていっても、化学シフトにほとんど変化がみられなかった。しかし、さらにRNAを加え、モル比が1:1になると、急にシグナルが移動し、大きな化学シフト変化を示していた (図3B)。RBD1-RBD2連結体に対してnumb 15merが少ない状態 (1:0~1:0.5) では、RBD2領域がRNAに結合する前に、他のRBD1-RBD2連結体分子のRBD1領域が、結合してしまったと考えられる。そのため、RBD2領域はRNAとの相互作用に関与することができず、化学シフトに変化がみられなかったと考えられる。これはおそらく、RBD1の結合後さらにRBD2も結合するには、何らかの構造変化が必要で、しかもこの構造変化にはある程度の時間が必要とされるために生じた現象だと考えられる。一方で、RBD1-RBD2連結体に対してnumb 15mer が等量もしくはそれ以上存在している状態では (1:1~1:2), numb15mer中の結合配列がRBD1領域で占拠されることがないため、RBD2領域もnumb 15merに結合することができ、化学シフトに変化がみられたと考えられる (図3C)。

RBD1-RBD2連結体では各ドメインがRNAとの相互作用において、異なる役割を担っていると考えられる。RBD1は先に述べた静電相互作用や構造的な揺らぎを利用して、標的RNAに強く結合する性能を有しており、これを活かして標的配列の探索を行っている。一方RBD2は、標的RNAが2つ目の結合配列を有する場合にのみ、多少時間はかかるが相互作用をし、より強固な複合体を形成するのに貢献していると考えられる。
5.RBD1-RBD2連結体-numb 15mer複合体の構造解析
現在、我々はRBD1-RBD2連結体とnumb 15merの複合体について、立体構造の解析を進めており、塩基配列特異的なRNA認識機構および翻訳抑制のメカニズムの解明を目指している。NMRによる立体構造解析では、NOEから水素原子間の距離情報が得られる。多くの水素原子間について距離制限を設けることにより、全体の立体構造を決定していく。このNOEから得られる距離情報は、最大で5 Å程度であり、局所的な距離情報しか得ることができない。したがって、RBD1-RBD2連結体のようなサンプルでは、各RBDの構造に関しては比較的容易に決定することができるが、RBD1とRBD2の相対的な位置関係や、分子の全体像を正確に求めることは簡単ではない。そこで我々は、双極子相互作用の情報を利用することにした。タンパク質主鎖のアミド窒素 (N) とアミド水素 (H) 間の双極子相互作用は、N-HベクトルとNMR装置内の静磁場とのなす角度に依存している。したがって、タンパク質中の様々なN-Hに対して双極子結合を求めることで、タンパク質の静磁場に対する配向が明らかとなり、タンパク質の全体像を決定することができる。しかしながら、溶液中ではタンパク質が自由に運動することができるため、N-H軸と静磁場がなす角度は、ありとあらゆる値を示し、双極子結合の値は平均化され0ゼロとなってしまう。したがって、溶液中で双極子結合の値を得るためには、分子の運動をある程度制限することが必要となる。具体的には、タンパク質溶液にバイセルなどの“障害物”を加えたり、タンパク質溶液をポリアクリルアミドゲルに導入することによって、タンパク質の運動を制限する。これにより、双極子結合は完全には平均化されないので、残余双極子結合として観測することが可能となる9,10)。我々は、RBD1-RBD2連結体-numb 15mer複合体について、ポリアクリルアミドゲルを用いて残余双極子結合を求め、複合体におけるRBD1とRBD2の相対配置を決定した。その結果、RBD1とRBD2は相互作用領域であるβシートを、互いに少し向かい合わせたような配置で存在していることが見出された (図4)。先に述べたようにnumb 15merは、各RBDのβシート領域およびドメイン間リンカーと相互作用することが分かったので、複合体においてはnumb 15merは多少折れ曲がった形で存在すると考えられる。
6.おわりに
本稿では、NMR法を用いたmMSIの構造解析およびRNAとの相互作用の解析を紹介した。最近、溶液NMR法で得られる運動性が、酵素反応速度と密接に関与していることが示され、注目されている11)。現在までのところ“柔軟性”や“構造的な揺らぎ”といった表現しかできず、運動性の実体は不明瞭であるが、生体高分子の機能を解明する上で、NMRから得られる運動性は有用な情報となるであろう。
NMR法は、他の分光法に比べマイルドな測定法である。したがって、様々な試料について非破壊的に原子レベルの解析をすることが可能である。このような特徴を生かして、現在では、生体高分子の解析において、In-Cell NMR法が注目を浴びている。これは、本稿で紹介したようなタンパク質の立体構造や、相互作用に関する情報を、細胞状態の試料から直に得る方法である。現在までに、大腸菌や酵母、アフリカツメガエルの卵母細胞などを用いた測定例が報告されている12,13)。将来的には、生体高分子の構造や機能を、生体内で生きたまま解析することにより、より詳細で高次な生命機能を解明することが可能となるであろう。
謝辞
本稿の内容は慶応大学医学部の岡野栄之教授との共同研究によって得られた成果であり、ご協力に感謝致します。
文献
1) Nakamura, M., Okano, H., Blendy, J. A., Montell, C.: Neuron, 13, 1 (1994).
2) Imai, T., Tokunaga, A., Yoshida, T., Hashimoto, M., Mikoshiba, K., Weinmaster, G., Nakafuku, M., Okano, H.: Mol. Cell. Biol., 21, 3888 (2001).
3) Nagata, T., Kanno, R., Kurihara, Y., Uesugi, S., Imai, T., Sakakibara, S., Okano, H., Katahira, M.: J. Mol. Biol., 287, 315 (1999).
4) Miyanoiri, Y., Kobayashi, H., Imai, T., Watanabe, M., Nagata, T., Uesugi, S., Okano, H., Katahira, M.: J. Biol. Chem., 278, 41309 (2003).
5) Nicholls, A., Sharp, K. A., Honig, B.: PROTEINS, 11, 282 (1991).
6) Lipari, G., Szabo, A.: J. Am. Chem. Soc., 104, 4546 (1982).
7) Katahira, M., Miyanoiri, Y., Enokizono, Y., Matsuda, G., Nagata, T., Ishikawa, F., Uesugi, S.: J. Mol. Biol., 311, 973 (2001).
8) Mittermaier, A., Varani, L., Muhandiram, D. R., Kay, L. E., Varani, G.: J. Mol. Biol., 294, 967 (1999).
9) Tjandra, N., Bax, A.: Science, 278, 1111 (1997).
10) Chou, J. J., Gaemers, S., Howder, B., Louis, J. M., Bax, A.: J. Biomol. NMR, 21, 377 (2001).
11) Eisenmesser, E. Z., Millet, O., Labeikvsky, W., Korzhney, D. M., Wolf-Watz, M., Bosco, D. A., Skalicky, J. J., Kay, L. E., Kern, D.: Nature, 438, 117 (2005).
12) Serber, Z., Keatinge-Clay, A. T., Ledwidge, R., Kelly, A. E., Miller, S. M., Dötsch, V.: J. Am. Chem. Soc., 123, 2446 (2001).
13) Burz, D. S., Dutta, K., Cowburn, D., Shekhtman, A.: Nature Methods, 3, 91 (2006).