【トピックス】
超好熱アーキアの新規フラビン含有アミノ酸脱水素酵素の機能開発
川上竜巳、櫻庭春彦、大島敏久
徳島大・工
1.はじめに
生体には、エネルギー生産、生体物質の生合成や分解代謝などに関与する多くの酸化還元酵素が存在する。酸化還元酵素は、アルコール、アミノ酸、糖、アミンなどの電子供与体と電子受容体間の電子移動を伴う反応を触媒するが、電子受容体が酸素である酸化酵素や酸素添加酵素と、NAD(P)、FADなどを電子伝達体とする還元酵素や脱水素酵素に大別される。脱水素酵素としては、アルコール脱水素酵素、グルタミン酸脱水素酵素など、NAD(P)を電子受容体とするものが多く見出され、それらの詳細な機能と構造解析やバイオセンサーやバイオリアクター用素子として応用開発が進んでいる1,2)。一方、NAD(P)ではなくFADやFMNなどのフラビンを電子受容体とする一群の脱水素酵素が存在する。これらの多くは、生体膜に存在する電子伝達系の初発酵素として、アミノ酸、有機酸、糖などの電子を取り込む機能をもっており、代表的なものとして、NADH脱水素酵素やコハク酸脱水素酵素などが古くから知られている3,4)。これらの脱水素酵素は人工の酸化還元色素である2,6-ジクロロインドフェノールやフェリシアン化カリウムなどを電子受容体として活性測定に利用するため、色素依存性脱水素酵素 (dye-DH) と呼ばれる。この脱水素酵素は酸化還元色素をメディエータとして基質の電子を電極へ直接導入できることから、電気化学的な検出が可能なバイオセンサーへの利用が期待されている5)。しかし、一般に膜結合性酵素であるので、その機能開発には膜からの可溶化が必要となるが、可溶化の過程で酵素が不安定化するため、機能性素子としての応用開発は進んでいない。また、これらの酵素は電子伝達タンパク質と複合体を形成するなど多様な構造をとるために、機能解析や構造解析もNAD(P)依存性の脱水素酵素と比較して随分遅れているのが現状である。
その一方で、好熱菌や超好熱菌と呼ばれる微生物は、常温菌などと比べて温度、pH、有機溶媒などに対して高い耐性を示す酵素を生産する。中でも超好熱菌の生産する酵素は水の沸点である100℃でも全く失活しない高度耐熱性を示すものもあり、産業への新たな応用が期待されている。超好熱菌はそのほとんどが分類学上アーキア (古細菌,始原菌ともいう) に属する。アーキアは現存する最古の生物とされており、真核生物、バクテリア (真正細菌) とも異なる第3の生物と呼ばれ、これまでにない代謝系や新奇な酵素を生産することから生物進化の観点からも興味深い研究対象である6)。著者らは、超好熱菌におけるdye-DHのスクリーニングを試み、これまでにL-プロリン、D-プロリンを基質とする色素依存性アミノ酸脱水素酵素を見出した7-11)。本稿ではこれらのうち、Thermococcales目の超好熱アーキアに分布する色素依存性L-プロリン脱水素酵素 (dye-L-ProDH) の機能と構造、応用について紹介する。
2.Thermococcus profundusの色素依存性L-プロリン脱水素酵素 (Tp-ProDH)
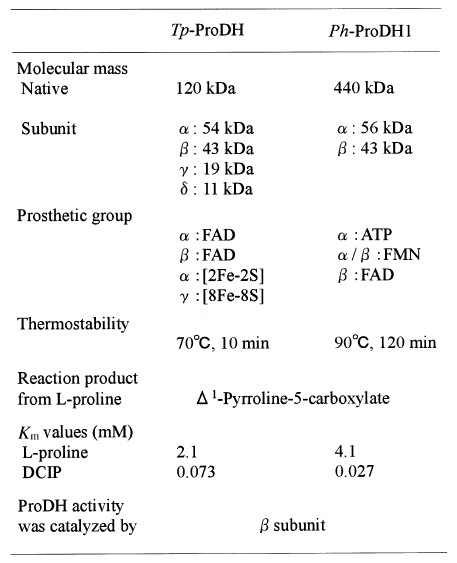
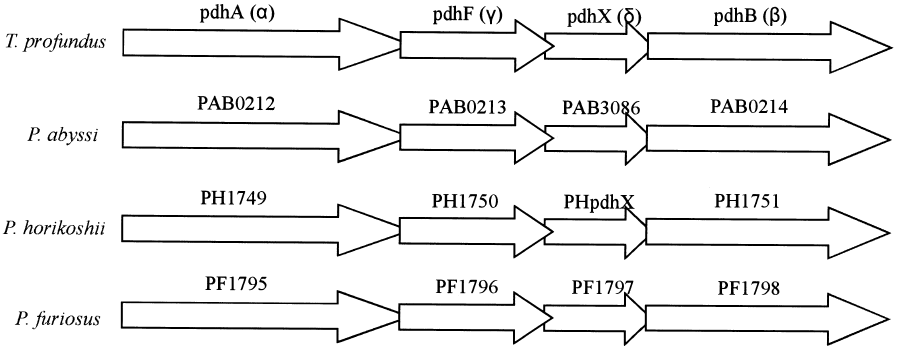
数種の超好熱アーキアを培養し、それらの細胞抽出液中のアミノ酸や有機酸などを基質とする色素依存性脱水素酵素を検索した。その結果、L-プロリンからΔ1-ピロリン-5-カルボン酸への反応を触媒するdye-L-ProDHを数種の超好熱アーキアに見出した (表1)。著者らはまず、比活性が最も高かったT. profundusの酵素について精製及び機能解析を行った7,8)。多くのdye-DHが膜酵素として単離されている中でこの酵素は膜には結合しておらず、可溶性画分に存在していた。精製の結果、Tp-ProDHは4種類のサブユニット (α: 54, β: 43, γ: 19, δ: 11kDa) からなり、その分子量は約120kDaであることからα β γδ構造をとる複合体酵素であることが分かった。また、Tp-ProDHは70℃、10分間の熱処理でも失活せず、pH4-10の広いpH領域で安定であるなど、高い安定性を示した。その他の酵素化学的性質については表2に示す。次に本酵素をコードする遺伝子のクローニングを行い、全長3,399bpの塩基配列を決定した。α β γδの各サブユニット遺伝子 (pdhA, pdhB, pdhF, pdhX) はクラスターを形成しており、pdhA-pdhF-pdhX-pdhBの順で並んでいる。Tp-ProDHと類似した遺伝子クラスターは、ゲノム解析情報から、特にThermococcales 目のアーキアにおいて、分布することが分かった (図1)。このことから本酵素がThermococcales 目において重要な生理的役割を担っていると考えられる。
表1 超好熱アーキアにおけるdye-L-proDHの分布
表2 Tp-ProDHとPh-ProDHの酵素化学的性質
図1 Tp-ProDH遺伝子クラスターとThermococcales目のアーキアに存在する類似の遺伝子クラスター
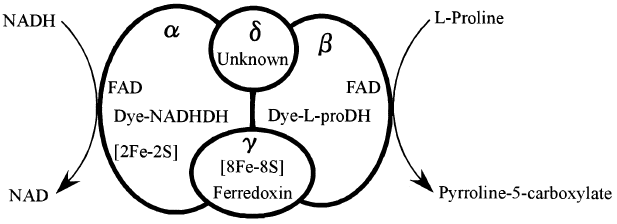
次に4種のサブユニットの単独発現系をそれぞれ構築して、各サブユニットの機能解析を行った (表2)。まず、dye-L-ProDH 活性について検討したところ、この活性はβサブユニットに検出できた。次にαサブユニットの機能解析から、色素依存性NADH脱水素酵素 (dye-NADHDH) 活性が検出できた。複合体酵素からもdye-NADHDH活性が検出されることから、αサブユニットはdye-NADHDHであることが確認できた。これまでにひとつの酵素で2種類の関連した酵素反応を触媒できるものは見出されている。例えばPutA (プロリンオキシダーゼ) は、本酵素と同様にL-プロリンからΔ1-ピロリン-5-カルボン酸へのFAD依存性の酸化活性とΔ1-ピロリン-5-カルボン酸からL-グルタミン酸へのNAD依存性の脱水素酵素活性を併せ持っている12)。しかし、本酵素のように、L-プロリンとNADHの脱水素酵素を併せもつ複合体酵素はこれまでに報告されておらず、本酵素が最初の例である。さらにγサブユニットは2つの[4Fe-4S]の鉄イオウクラスターを持つ電子伝達タンパクであるフェレドキシン様タンパク質であることが分かった。δサブユニットの機能については現在のところ、まだ不明である。以上の結果から、Tp-ProDHはL-プロリン脱水素酵素、NADH脱水素酵素、フェレドキシン、機能未知タンパク質から構成される新規複合体酵素であることが判明した (図2)。この酵素複合体の生理的機能が、L-プロリンからΔ1-ピロリン-5-カルボン酸への変換反応を行うのか、逆のL-プロリン合成反応であるのかについては、δサブユニットの機能とともに今後の解明が待たれる。
図2 Tp-ProDHの模式図
3.Pyrococcus horikoshii の色素依存性L-プロリン脱水素酵素
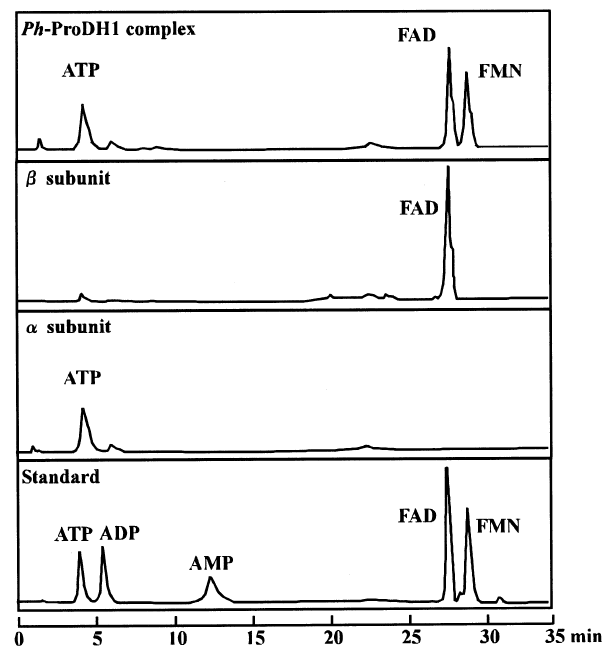
当初の酵素活性のスクリーニングにおいて、P. horikoshii及びT. peptonophilusからは電気泳動的に移動度が異なる2種類のdye-L-ProDH活性バンドが検出された。また、図1にも示したようにP. horikoshiiにもTp-ProDHと同様の遺伝子クラスターが存在する。これらのことからP. horikoshiiにはTp-ProDHと同タイプのdye-L-ProDHに加えて、別の新規なdye-L-ProDHが存在することが予想できた。これら2種類のdye-L-ProDHをPh-ProDH1、Ph-ProDH2として両酵素の特徴を解析した9)。細胞粗抽出液から両酵素を高度に分離・精製した結果、Ph-ProDH2がTp-ProDHと同様の4種類のサブユニットからなる複合体酵素であり、各サブユニット遺伝子も予想したものと一致した。一方、Ph-ProDH1は56 (α) と43kDa (β) の2種類のサブユニットからなる分子質量約440kDaのα4 β4構造をとる複合体酵素であることが分かった。Ph-ProDH1のα、βサブユニットはそれぞれPH1363、PH1364遺伝子によってコードされており、この遺伝子クラスターもThermococcales目のアーキアに広く分布していた。Ph-ProDH1はTp-ProDHと比較して、より高い熱安定性を示し、90℃、120分間の熱処理によっても失活しなかった (表2)。Ph-ProDH1の特筆すべき点は含まれる補欠分子の種類である。HPLC解析の結果から、Tp-ProDHはαとβサブユニットそれぞれに1分子ずつFADを含み、酵素複合体として2分子のFADを含んでいる。一方、Ph-ProDH1はFADだけでなくFMN、さらにはATPも含有していることが明らかになった (図3)。トリメチルアミン脱水素酵素でADPが結合している例は報告されているが13)、ATPが結合している脱水素酵素の例はなく、本酵素が最初である。さらにこれらの補欠分子がどのサブユニットに結合しているかを、単独発現させたサブユニットで分析した結果、FADがβサブユニットに、ATPがαサブユニットに含まれていることが判明した。しかしFMNはどちらのサブユニットからも検出できなかった。このことからFMNはα、βサブユニットの境界に存在することが予想された。また、機能解析からβサブユニットにdye-L-ProDH活性が認められたが、αサブユニットにはTp-ProDHに認められたNADHDH活性は認められなかった。
図3 Ph-ProDH1の補欠分子のHPLC解析
4.Ph-ProDH1のX線結晶構造解析と新しい電子伝達系
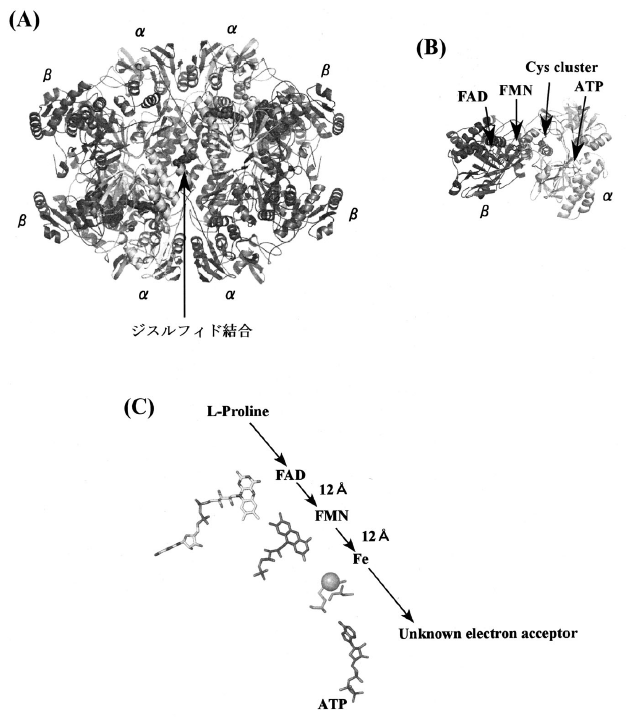
Tp-ProDHとPh-ProDH1について、さらに詳細な構造的知見を得るため、酵素の結晶化を行った。Tp-ProDHの結晶は現在のところまだ得られていないが、Ph-ProDH1については結晶化に成功し、2.8 Åの分解能でその構造を決定した10)。Ph-ProDH1の基本構造はαβヘテロダイマーであり、2つのCys381αがジスルフィド結合によって架橋された(αβ)2のテトラマー構造を構成し、2分子の(αβ)2テトラマー構造が重なり合うようにして全体構造である(αβ)4オクタマーを構成していた (図4)。その構造解析で、色素依存性L-プロリン脱水素酵素複合体の構造と機能に関する下記に記す興味ある新規知見が得られた。
図4 Ph-ProDH1の全体構造(A)とαβ構造(B)及び新しい電子伝達系(C)
HPLC解析で明らかにしたように、FADはβサブユニットに、ATPはαサブユニットに結合していた。一方、HPLC解析ではわからなかったFMNの結合部位は、構造解析からα、β両サブユニットに挟まれるように結合していることが判明した。さらにαサブユニット中には4個のシステイン残基 (Cys413、Cys415、Cys447、Cys452) から形成されたCys-cluster部位や、5個のトリプトファン残基のうち4個が集まって形成された疎水性に富むTrp-cluster部位が見出された。Cys-clusterは、そのドメインのアミノ酸配列から[2Fe-2S]の鉄イオウクラスターと予想されたが、構造解析からは鉄の電子密度は確認できなかった。しかし、生化学的分析やEPRスペクトル分析からは鉄が存在する結果が得られることから、鉄は結晶化の過程で酵素から遊離したのかもしれない。また、X線結晶解析から疎水性に富むTrp-cluster部位に、結晶化に利用した緩衝液の成分であるCAPSが結合した構造が得られたことから、この部位は、生理的にはCAPSに代わるキノンなどの疎水性に富む電子伝達体が結合すると予想される。
以上の生化学的解析とX線結晶構造解析から、次のような新しい電子伝達系がPh-ProDH1に存在すると考えられる (図4、C)。まず、電子供与体のL-プロリンから、電子がβサブユニットのFADに取り込まれFADH2が形成される。次に電子は12 Å離れたβとαサブユニットの接触面に位置するFMNへと伝達され、さらにそのFMNから12 Åの距離にあるαサブユニットのCyscluster部位の鉄へと伝達される。最終的に電子はαサブユニット中の疎水性に富むTrp-cluster部位に結合する未同定の電子受容体へと流れる。なお、αサブユニットに存在するATPの役割については現在のところ不明であるが、電子伝達には直接関与せず、高次構造の保持や電子伝達の調節因子として機能していることが予想される。
5.おわりに
今回、Thermoccocales 目のアーキアに特異的に分布する2種類のdye-L-ProDHの機能と構造解析の結果について紹介した。常温性生物由来の色素依存性脱水素酵素は、安定性の低さから解析が困難でその研究は遅れていた。このような状況の中で筆者らは安定性に優れた超好熱菌の酵素を用いることでこれまで解析できなかったdye-DH の機能と構造解析に成功できた。安定性に優れたdye-DH は応用面においても可能性を秘めている。筆者らは既にTp-ProDHを素子とする電極型センサーを構築し、電気化学的に検出可能なL-プロリン定量用バイオセンサーの構築に成功している14)。また、このTp-ProDHを素子とする電極型センサーとPCR法による肺炎病原菌のレジオネラ属菌のDNA増幅を組み合わせた特異的な電気化学的検出センサーの開発に成功している15)。酵素ハンドブックによると16)、85 種を越える色素依存性脱水素酵素が存在し、アミノ酸、有機酸、エネルギー、薬剤などの代謝系に重要な役割をもつことが報告されているが、機能や構造についての知見は極めて少ない。その中で高い安定性をもつ超好熱菌由来の色素依存性脱水素酵素は、機能と構造解析、応用開発において突破口を開くものとして、極めて重要な役割をもつことが期待できる。最近のゲノム解析から急激に蓄積している超好熱菌のゲノム情報を踏まえ、著者らは今後さらに超好熱菌の種々の色素依存性脱水素酵素と関連酵素の機能開発を進展させる予定である。
文献
1) Kula, M. R., Kragl, U.: In Stereoselective Biocatalysis, Marcel Dekker Inc, 839-866 (2000).
2) Ohshima, T., Soda, K.: In Stereoselective Biocatalysis, Marcel Dekker Inc, 877-902 (2000).
3) Dancey, G. F., Levine, A. E., Shapiro, B. M.: J. Biol. Chem., 251, 5911-5920 (1976).
4) Hederstedt, L., Holmgren, E., Rutberg, L.: J. Bacteriol., 138, 370-376 (1979).
5) Frew, J. E., Hill, H. A.: Anal. Chem., 59, 933A-944A (1987).
6) 古賀洋介, 亀倉正博(編): 古細菌の生物学, 東京大学出版会 (1998).
7) Sakuraba, H., Takamatsu, Y., Satomura, T., Kawakami, R., Ohshima, T.: Appl. Environ. Microbiol., 67, 1470-1475 (2001).
8) Kawakami, R., Sakuraba, H., Ohshima, T.: Extremophiles, 8, 99-108 (2004).
9) Kawakami, R., Sakuraba, H., Tsuge, H., Goda, S., Katunuma, N., Ohshima, T.: FEBS J., 272, 4044-4054 (2005).
10) Tsuge, H., Kawakami, R., Sakuraba, H., Ago, H., Miyano, M., Aki, K., Katunuma, N., Ohshima, T.: J. Biol. Chem. in press (2005).
11) Satomura, T., Kawakami, R., Sakuraba, H., Ohshima, T.: J. Biol. Chem., 277, 12861-12867 (2002).
12) Maroy, S. R.: In Cellular and molecular biology, vol. 1, American Society for Microbiology, 1513-1519 (1987).
13) Lim, M. W., Mathews, S. F., Steenkamp, D. J.: J. Biol. Chem., 263, 3075-3078 (1988).
14) 廣瀬由鷹, 木村友和, 鄭海涛, 末信一朗, 堀照夫, 片山秀夫, 川上竜巳, 大島敏久: 日本生物工学会平成16 年度大会, 3J09-3 (2004).
15) 大島敏久: 微生物の取扱いと利用・応用技術, 情報機構(東京) 224-234 (2003).
16) Enzyme Nomenclature, Academic Press (1992).