【トピックス】
プログラマブル人工タンパク質創製
芝 清隆
癌研・蛋白創製
1.はじめに
ねらった機能や構造をもったタンパク質を自由自在に設計・合成できるようになることが人工タンパク質研究の目標の1つである。タンパク質科学研究で蓄積した知識に基づき、合理的に特定の立体の構造を形成するアミノ酸配列をデザインしようとするのが蛋白工学 (プロテインエンジニアリング) である1)。これに対して、とにかくいろいろ作ってみて、その中から目的とする人工タンパク質を結果的に選び出すことさえできればよし、とするのが進化分子工学である2)。合理性よりはむしろ「選択」を重視した戦略である。
この小解説ではわれわれが開発してきた人工タンパク質創製法、MolCraft3,4)を紹介する。この手法は蛋白工学の合理性と進化分子工学の選択重視の両方の性質を兼ね備えている。すなわち、ある程度はねらった機能を合理的に人工タンパク質に与えることができるが、予測不可能な部分はライブラリーからの選択で切り抜けよう、といった半合理的・半選択重視的戦略の人工タンパク質創製法である。
2.機能モチーフとプログラマブル人工タンパク質
「モチーフ」とはここではタンパク質の一次構造上に同定・定義される短いアミノ酸配列を意味する。モチーフが定義されるには、例えば、ヒト、マウス、酵母が共通してもつタンパク質で、種を超えて保存された配列として同定される場合がある (図1A)。あるいは同じ生物ゲノム内でも、遺伝子重複で進化した姉妹タンパク質の配列の比較から保存配列として見いだされることもある。このような進化を通して保存されている配列は、なんらかの機能、あるいは構造上への選択圧、すなわち、何らかの重要な働きをもつと考えて良い。

単に配列が保存されているから重要であろう、という段階から一歩進むと、積極的にその保存された配列を他の配列に変化させたりする操作などから (多くの場合、アミノ酸を性質の異なった種類のものに変換することにより機能が失われる)、モチーフと特定の生物機能との関連がさらに証明されていくことになる (図1B)。
さらに進んで、そのモチーフが特定の機能を発揮するための必要十分条件であることを示すためには、そのモチーフ配列を取り出し、他の分子に移植する実験をおこなう。モチーフを移植された新しい分子が、そのモチーフに関連づけられた機能を獲得した場合に、モチーフと機能との関連が一層力強いものとなる (図1C)。あるいは、場合によっては取り出したモチーフ配列のみからなるペプチドに、その関連づけられた機能を発揮する能力が見いだされる。
このように、特定の機能と対応付けられた短いアミノ酸配列-モチーフの情報が蓄積してくると、次には、これらのモチーフを組み合わせて、天然には存在しないような組合せのタンパク質が創製できないか、といった興味がわいてくる。すなわち、Aといった機能に対応したモチーフ-Aを1つの天然タンパク質から抽出し、別の天然タンパク質からは、Bといった機能に対応したモチーフ-Bを抽出し、これらを連結することによって、AとBの両方の機能をもつ人工タンパク質を創ろうという試みである (図2)。すなわち、機能モチーフに関する知識を合理的に利用して、機能Aおよび機能Bをプログラムして、新たな複合機能分子を創る、プログラマブル人工タンパク質創製である。
図2 モチーフ・プログラミング
現在、網羅的なタンパク質の性質解析研究が大規模に進んでおり、機能モチーフの情報を加速度的に蓄積している。このようなゲノム知識を再構築して応用利用する、ポストゲノム研究の1つとも捉えることができる。
3.モチーフ・プログラミングの難しさ
特定の機能に対応付けられたモチーフ配列を組み合わせて、天然にはない機能の組合せをもった人工タンパク質を創るのがプログラマブル人工タンパク質である。天然タンパク質をペプチド配列に分解して、機能に関連したペプチド配列だけを再配置して新しい人工タンパク質を創ろうとする試みである。概念的には単純な作業であるが、実際にこのような実験を進めていると、解決すべきいくつかの問題が出てくる。これらの問題をまとめると次の2つになる。
(1) モチーフ配列を、本来その配列が埋込まれている天然タンパク質から切り出した場合、対応付けられている機能を失う場合がある。
モチーフ配列が特定の機能を担うことを説得性高く示すには、天然タンパク質から切り離したペプチドモチーフ (化学合成したペプチドと考えてよい) そのものが、対応付けられた機能を持つことを証明すれば良い。しかしながら、多くの場合、このような切り出されたペプチドモチーフは、対応付けられた機能を失う場合が多い。切り出す範囲が間違っていたのでは、という可能性も考えられるが、切り出したペプチドを今度は親タンパク質とは全く関係の無い別のタンパク質に移植してやると (多くの場合、このような移植実験は組み換えDNAレベルでおこなう)、本来の機能が再活性化される場合がある。この場合も、必ず再活性化されるというわけではなく、移植相手のタンパク質や移植場所によって活性が復活しない場合もある。あきらかに、モチーフを取り囲むタンパク質の環境がモチーフの活性に影響を与えているわけであるが、どのような環境にモチーフをもってくることが、効率よい活性の発現につながるかは、少なくとも現在では予測不可能な部分である。したがって、モチーフをいろいろなコンテクストで骨格タンパク質に埋込んだライブラリーを作製し、その中から期待した活性をもったクローンを選ぶといった、「選択」重視の進化工学的アプローチが必要となってくる。
(2) ペプチド配列をどんどん連結してポリペプチド化していったとしても、天然タンパク質のように安定した立体構造をとれず、多くの場合不溶性のポリペプチドとなる。
機能モチーフに関して単純な足し算が成り立つとすると、いろいろなモチーフ配列をどんどん連結していけばいくらでも複雑な多機能ペプチドができることになる。しかしながら、このような単純デザインのポリペプチドには、天然のタンパク質が示す折りたたみ構造は期待できない。多くの場合は不溶性ポリペプチドとなって沈殿してしまうか、あるいは特定構造をもたないポリペプチドとして溶液中に存在する。天然タンパク質レベルとは言わないまでも、プログラマブル人工タンパク質にある程度の折りたたみ構造を与えるのが望ましいと考えられる。
上記のような2つの問題点をふまえて、われわれの開発したMolCraftによるモチーフ・プログラミング実験を紹介したい。
4.MolCraftを用いた人工シグナル分子の創製
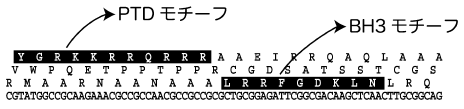
ここでは、MolCraftを用いた人工タンパク質創製実験として、アポトーシスに関わるBH3モチーフとタンパク質形質導入に関わるPTDモチーフを組み合わせて新しいシグナル分子を創製した研究を紹介する5)。
BH3モチーフはアポトーシスに関連したシグナル経路を形成するタンパク質 (Bcl-2、Bax、Bid等) に共有されている機能モチーフである6)。BH3モチーフには基本的にはアポトーシス誘導能力があると考えてよいが、われわれは9残基という短いBH3モチーフ (多くのBH3モチーフを用いた実験は20-25残基のペプチドが用いられている) をNoxaと呼ばれる天然タンパク質から抽出して用いている。一方のPTDモチーフは、HIVのtatタンパク質に由来する11残基のモチーフで、既にタンパク質のみならず核酸や化学化合物などの細胞内への導入モチーフとして利用されている7)。
BH3モチーフとPTDモチーフを足し合わせて期待できる複合機能は、「細胞内に自動的に進入してアポトーシスを誘導」する活性である。試しに、BH3モチーフとPTDモチーフを単純に連結したペプチドを化学合成し、このペプチドの挙動を調べてみた。その結果、この連結ペプチドは細胞の中に入ってはいくものの、アポトーシスを誘導することは無かった5)。予想通り (?) ペプチドモチーフの単純な足し算は成り立たなかったわけだ。BH3モチーフが何らかのコンテクストで人工タンパク質中に埋込まれないと、期待する複合活性は得られないと考えられる。
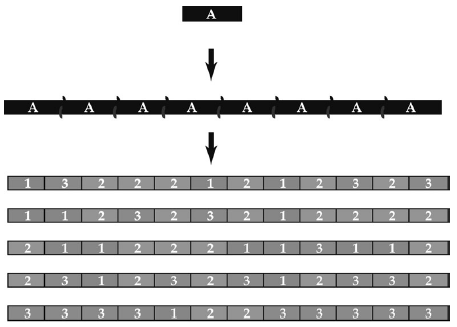
次にMolCraftによる人工タンパク質創製を進めるために、まず図3のような人工的な小さな遺伝子 (マイクロ遺伝子) を設計した。このマイクロ遺伝子は全長72塩基しかなく、その読み枠の1つにBH3モチーフを、他の読み枠にPTDモチーフをコードするのが特徴である。このマイクロ遺伝子をMicrogene Polymerization Reaction (MPR) 法3)と呼ばれる特徴的な反応条件でのDNAポリメラーゼによる複製反応を進めることにより、タンデムに重合することができる。MPR法の大きな特徴として、集合体の連結点にランダムに塩基の挿入・欠失がおこる。したがって、重合体のマイクロ遺伝子の読み枠が、連結点でランダムにシフトすることになる。すなわち、MPR法で得られたマイクロ遺伝子の重合体の翻訳産物は、ブロック単位であるマイクロ遺伝子がコードする3つのペプチドの組み合わせ的な重合体となる (図4)。
図3 マイクロ遺伝子

マイクロ遺伝子の異なる読み枠にBH3モチーフとPTDモチーフを埋込むことと、MPR法で読み枠をランダムにずらしながらこのマイクロ遺伝子を重合することによって、BH3モチーフとPTDモチーフがいろいろな順番、いろいろな数で連結した人工タンパク質ライブラリーを調製することができる。このようにして得られた人工タンパク質は、BH3モチーフとPTDモチーフ配列以外にも、マイクロ遺伝子の3つの読み枠がコードする人工的なペプチドをいろいろな割合で含むことになる。したがって、マイクロ遺伝子をデザインする際には、このモチーフとは関係のないペプチド配列にも、二次構造形成能力や適度な疎水性度などを与えるようにDNA配列を選んでいる。
このようにして調製したBH3モチーフとPTDモチーフを埋込んだ人工タンパク質ライブラリーの中から、高頻度に「細胞の中に自動的に進入しアポトーシスを誘導する」といった複合活性をもつクローンが含まれていた。面白いことに、同じ数のモチーフを同じ順番で含むクローンであっても、その活性はクローンによって大きく異なった。この活性の違いが何に由来するのかは、現時点では不明である。あるいは、このような実施例を積み重ねることにより活性の違いを生み出す条件が見えてくるのであろうが、少なくとも現時点では、「いろいろ作ってみて」、その中から「期待した活性をもつクローンを選択する」といった進化分子工学的なアプローチが重要であろう。すなわち、MolCraft の前半が、特定の機能に関連づけたモチーフ配列をマイクロ遺伝子の中にプログラムする、といった蛋白工学的な合理的側面をもつのに対し、後半は、ライブラリーからの選択といった進化分子工学的側面をもつ。進化分子工学では特定の機能や構造にバイアスのかかっていない「ランダム」な配列集団をライブラリーとして用いているのに対して、MolCraft では特定の機能に大きくバイアスのかかったライブラリーを用いている。したがって、スクリーニングの母集団も何億、何十億といった単位ではなく、何十、何百といった数で十分である。
5.MolCraft の展開
上の例では、天然には存在しないモチーフの組合せから、人工的なシグナル分子を創製する研究を紹介した。このような研究の延長線上には、治療を目的とした人工タンパク質製剤の開発や、信号伝達ネットワーク研究の新しいツールとしての可能性が考えられる。
現在われわれが注目しているMolCraftの展開研究の1つに生物ワールドと無機材料ワールドの橋渡しをする人工タンパク質創製研究がある。プログラムするモチーフは、何も天然タンパク質に由来するものに限る必要はない。進化分子工学から創製される人工モチーフもプログラム単位として用いることができる。進化分子工学から創出される人工モチーフは多くの場合、結合活性をもったペプチド=ペプチド・アプタマーである。さらに、このようなペプチドアプタマーは、そのターゲットが、最近生体高分子から無機材料へと広がりつつある。われわれの研究室でも、チタン表面8)、カーボンナノ化合物表面9)に特異的に結合するペプチドアプタマーを進化分子工学的手法で取得した。この人工モチーフを種々の生物活性モチーフとMolCraftで組み合わせることにより、例えばチタンインプラントの生体親和性を高める人工タンパク質や、薬物送達分子としてのカーボンナノ化合物10)表面の改質に利用できるような人工タンパク質の創製が可能となるであろう。
アミノ酸をブロック単位とした組合せから人工ペプチドを創製するのが進化分子工学である。今後は、ここに示したような、ある程度活性や構造をもったペプチド (マイクロ遺伝子) をブロック単位として、その組合せからより複雑な人工タンパク質を創製する研究がますます盛んになるものと思われる。
文献
1) Ulmer, K. M.: Science, 219, 666 (1983).
2) Szostak, J. W.: Trends Biochem. Sci., 17, 89 (1992).
3) Shiba, K., Takahashi, T., Noda, T.: Proc. Natl. Acad. Sci. USA, 94, 3805 (1997).
4) Shiba, K.: J. Mol. Catlys. B, 28, 145 (2004).
5) Saito, H., Honma, T., Minamisawa, T., Yamazaki, K., Noda, T., Yamori, T., Shiba, K.: Chem. Biol., 11, 765 (2004).
6) Opferman, J. T., Korsmeyer, S. J.: Nat. Immunol., 4, 410 (2003).
7) Vocero-Akbani, A., Chellaiah, M. A., Hruska, K. A., Dowdy, S. F.: Methods Enzymol., 332, 36 (2001).
8) Sano, K., Shiba, K.: J. Am. Chem. Soc., 125, 14234 (2003).
9) Kase, D., Kulp, J. L., Yudasaka, M., Evans, J. S., Iijima, S., Shiba, K.: Langmuir, 20, 8939 (2004).
10) Murakami, T., Ajima, K., Miyawaki, J., Yudasaka, M., Iijima, S., Shiba, K.: Mol. Pharm., 1, 399 (2004).