【トピックス】
未知細菌の探索
― 地球は未知の細菌で満ち溢れている―
花田 智
産総研・生物機能工学
1.はじめに
昨年の11 月、新聞紙面の片隅を「細菌に新門発見」という見出しが付けられた小さな記事が飾った。「系統的に新奇な細菌が発見され、Gemmatimonas aurantiaca と名付けられた。そして、その発見がきっかけとなり新門の提案がなされた」ということであった。この「細菌系統における新門提案・認定」というのが極めて希なことであり、また本邦初の快挙であったのだ (なお、「門」と言う分類階級は細菌、すなわちバクテリア、の中で最も大きなものである)。本菌株は我々の研究グループにより下水処理場の活性汚泥から分離され、命名・提案されたものである。このような「系統的に全く新しい細菌」の発見は希有なことだと思われるかもしれない。しかし、我々の研究グループでは、このG. aurantiaca の他に、新門の提案までは到達しないまでも、それに準ずる「系統的に極めて新規な細菌」を様々な環境中から続々と分離することに成功している。では、どうしてこのような菌株の分離培養に我々は成功しているのか?その理由は「我々が極めて単純ではあるが従来とは異なるアプローチで菌株の分離に望んでいること」と「環境中には未だ発見されていない未知菌株が大量に存在しているということ」であると考えている。つまり、環境中の微生物生態系の大半は未知微生物で占められており、人と違う視点で分離培養に取り組めば、既存菌株と異なるものが発見出来て当然なのである。
未だ明らかになっていない環境微生物生態系とその全容解明のために我々の研究グループがなぜ分離培養という古典的方法を用いているのかについて、この与えられた紙幅で述べ、そして、我々の取っている分離戦略、それにより我々が発見したG. aurantiacaを含む系統的に極めて新規な数種の細菌について (手前味噌であることを承知で) 論じたい。
2.地球は未知の細菌で満ち溢れている
極めて小さく単純で、最も単純な生命様式のひとつである細菌 (バクテリア) は地球上の至る所に分布している。例えば土の中や海中、空気中、あるいは我々の皮膚の上など、文字通りいかなる場所においても、もし顕微鏡を持っているのであれば、私たちはそれらを簡単に観察することが出来る。現在までに6000種以上の細菌が環境中から分離培養され、その形や性質が明らかにされているが、実際にはその数 (または、バリエーション) は、環境生態系を構成する全細菌の1% にも満たないと考えられている。裏を返せば、細菌群集の99%以上が素性の知れないもので占められているということになる。つまり、一握りの土やコップ一杯の海水ですら、数え切れないほど多量な未知細菌を含んでいることになる。
3.培養に依存しない環境微生物生態系多様性解析が示唆したもの
当然、ここで言う「存在が認められている細菌種」というのは分離培養され、その菌学的性質が明らかとなっている (換言すれば、種名が命名されている) ものを指している。しかし、ここ10年、このような分離培養に全く依存しない環境細菌群集の多様性解析手法が盛んに行われるようになってきた。これは環境中の細菌に含まれる16S rRNA遺伝子配列を標的として直接、細菌の多様性を解析する手法が中心となっており、未だ分離培養に成功していない細菌を含め、細菌群集の系統的多様性や構造の完全解明を可能とするものである。これら「培養非依存型の細菌群集構造解析」は、それまでの菌株の分離・培養に基づいた見識が、環境微生物生態系のほんの一部を反映したものに過ぎないことを明確に示したのである1,2)。また、細菌の系統的多様性についても、分離菌株を対象とした系統分類体系に比べ遙かに大きいことが示唆されている。つまり、環境中には未だ分離培養に成功していない未知菌株が多数存在していることが、このような遺伝学的手法により客観的に確かめられたと言ってよい。
では、それら多様な細菌の個々の機能や環境中での役割は何なのだろうか? それを明らかにすることがこれからの重要な課題である。環境中の機能遺伝子を対象とした多様性解析、FISH法 (やMAR-FISH法) を用いた特定細菌の検出・定量・機能解析等の分子生物学的手法を組み合わせ、環境細菌群集の構造と機能、そして各々の細菌の役割を総合的に解明しようとする試みも、当然始まっている。しかし、そのような分子生物学的手法を用いたアプローチだけではなく、新規細菌株の分離培養という極めて古典的な手法も有効であると我々は考えている。分離培養という手法は純粋培養菌体を手に入れるということに他ならず、極めて根気のいる作業であり、時間がかかるものではある。しかし、それによりその細菌の形態学的・系統学的情報のみならず、一気に生理学的・生化学的データを同時に入手出来ると言った利点がある。新たなる視点で戦略を立て効率的なやり方で望んでいけば、これは十分に実りある結果が期待出来る手法である。その方向性が誤っていない限りにおいて、系統的に極めて新規な (同時に新規な機能を有する) 未知菌株の発見に至ることが出来るものと言える。
4.未知細菌を発見する戦略
細菌株の分離培養では、一般に寒天培地を用いた平板培養法 (白金耳を用いた菌液の塗り広げ) や液体培地を用いた限界希釈法 (確率的に細胞数が1個となる段階までの希釈) が使われる。我々の研究グループでも、分離に関して、このような従来の方法を基本的に用いている。しかし、従来法をそのまま使ったのでは、新規性の高いものを得る可能性は低くなってしまう。未知細菌の分離の効率化を図るため、私たちは以下の4つの戦略を立てている:
1) 現行の分離培地に若干の変更を加える:多くの環境中の栄養濃度は極めて低いので、従来に比べ低栄養の培地を使用する等。
2) 生育速度 (コロニー出現速度) の遅いものをねらう:従来はコロニー出現時期が早期なものを研究対象にしている事が多かった。
3) 「普通ではない環境」を分離対象とする:温泉や大深度地下、化学物質汚染土壌等には特殊な細菌が生息している可能性がある。
4) 二者培養 (co-culture) での増殖を試みる:単独では生育出来ない細菌を、その共生細菌とともに培養する。
これら戦略を単独で、または組み合わせて用いることにより、従来のものとは系統的に (同時にその性質も) 異なる新規な細菌を環境中から効率的に分離することに成功したのである。
5.発見された系統的に極めて新規な細菌たち
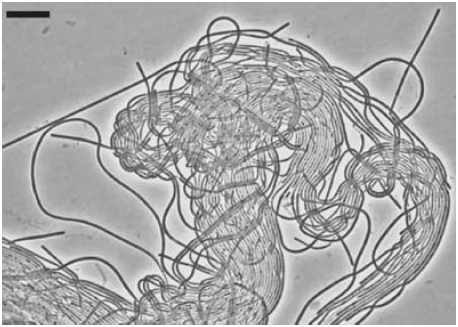
実際、これらの戦略を組み合わせて用いることにより、高次分類階級レベル (科、目、綱、亜門等) で系統的に新規な細菌の分離に成功している (図1)。例えば、非酸素発生型光合成を行う新規好熱性繊維状光合成細菌Roseiflexus castenholzii (図1) は温泉中の微生物被膜から、シアノバクテリアとの共培養により増殖に成功し、後に純粋分離されたものである3)。この細菌に関しては新属新種としての提案しかなされていないが、十分に新科または新目の提案が可能な系統分類学的新規性を持つものである。また、硫黄を大量に含む温泉水の白色ミクロバイアル・マットから単離された新規硫酸還元細菌Thermodesulfobium narugense はいずれの既存硫酸還元細菌とも系統的に隔たっており、その新属新種提案では、同時に新科Thermodesulfobiaceaeを提案した4)。また、高温嫌気リアクターと温泉からそれぞれ分離された繊維状細菌、Anaerolinea thermophila, Caldilinea aerophilaは新属新種としての提案しかなされていないが、いずれも亜門レベルでの新規性が認められているものである5)。
![]()
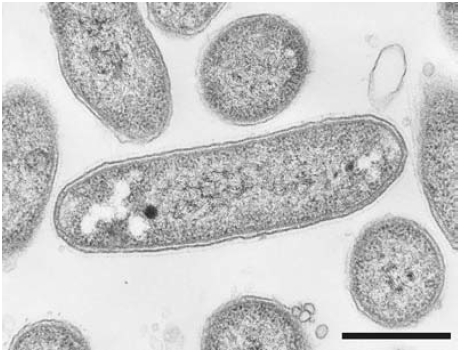
国内初の「新門」の提案の標準菌株となったG. aurantiaca (図2) もこのような研究の中で発見されたものである。本細菌は、従来の方法に比べ長い培養時間 (2週間以上) の後に現れてくる増殖速度の遅い (正しくはコロニー出現時期が遅い) ものを選択するという手法を用いて、廃水処理システム中の活性汚泥より単離されたものである。分離された新単離株は、細胞の直径が0.7µm、長さは2.5-3.2µm程度の酸素呼吸によって生育するという形態もその生育条件も一般的と思われる菌株であったが、16S rRNA遺伝子配列に基づく系統解析からいかなる既存菌株とも配列相同性が低く、系統的に大きく隔たっていることが明らかとなった。このような系統分類学的特殊性を受けて、本菌株を完全に新たな分類門 (Gemmatimonadetes phyl. nov.) を代表する新属新種、Gemmatimonas aurantiaca gen. and sp. nov.として提案し、受理された6)。(ここで述べたもの以外に、我々の研究グループが中心となって分離と提案を行った新属・新種に関しての主要な論文7-13)を文献情報のみではあるが、文末 (文献) に列記した。系統的新規性に関しては上記のものほどではないが、みな興味深い生理学的特徴を持っている。)
![]()
6.新規単離菌株の新規機能
「系統的に新規な細菌は機能的にも新規なものを有するはずである」と言う幾分楽観的過ぎるとも言える考えは、しかし、あながち間違ってはいない。既存細菌と大きく系統的に隔たっているものは、その機能遺伝子の構造や配列が大きく異なっているのみならず、その機能タンパク質 (酵素) 自体の高次構造や構成まで異なっていることが多い。また、細菌が生産する生体物質にも新規なものが見られる。先に挙げた好熱性繊維状細菌R. castenholzii の持つ光合成器官は光合成細菌の中でも最も単純な構造を持っていることが明らかになってきており、また、この細菌は新規カロテノイド糖エステル (ketomyxocoxanthin glucoside ester) 有している14)。この新規色素は両親媒性的特性を持つ抗酸化物質であり、新たな機能性食品・添加物への開発に結びつく可能性がある。また、温泉で発見された新規硫酸還元細菌T. narugenseの硫酸還元に関係する主要酵素は、構成こそ他の硫酸還元細菌と大きな差は見られなかったが、そのアミノ酸の1次配列はいかなる硫酸還元細菌のものとも高い相同率を示さず、機能や特性に明らかな差違がある可能性が示唆されている4)。
当然このような機能的新規性は、新門提案の基準菌株となった新規菌株G. aurantiacaにおいても発見されるはずである。本菌株の特筆すべき性質は、そのリンの取り込み能力であり、生育環境である下水処理システム中においてもリン除去に関して重要な役割を果たしていることが示唆されている。リンは河川や湖沼の富栄養化を引き起こす原因物質であり、下水のリン除去というのは重要な問題のひとつであるが、それを担っている細菌についてはいくつか知見はあるものの12,15,16)、その全体像は未だ明らかになっているとは言い難い。加えて、生物学的リン除去の制御についても解決しなければいけない問題が多いと言った現状である。既知菌株から遠く隔たっているというこの細菌の系統的位置から考えて、全く新しいリン取り込みメカニズムが細胞内に存在して可能性があると予想している。現在、これを遺伝子レベルで明らかにしようとゲノム配列の全解明が計画されている。このゲノム解析を通じて遺伝子レベルでの新規なリン蓄積機構が解明されれば、リン除去技術の向上、ならびに化学工業への用途につながるリン酸化関連反応酵素の開発に関しての重要な情報が得られるものと期待されている。
7.微生物生態系という壮大なジグソーパズル
環境微生物生態系をひとつの大きなジグソーパズルと考える。そして個々の微生物種がひとつひとつのピースである。ピースの表面にはその細菌の諸性質が細かく書き込まれている。パズルのピースの大部分は未だ様々な環境中に埋もれており、我々に発見されるのを待っている。環境微生物生態系の全容解明という究極の目的のためには、一枚でも多くの「隠されたピース」を探し出すことが重要である。この隠れたピース探しは極めてタフで時間を喰う研究方法である。しかし、未知なるピースに未知なる絵柄が書かれているように、未知なる細菌は未知なる機能を有している。つまり、新規細菌の探索とは新規機能、新規酵素、新規遺伝子の探索と等価なのである。微生物生態系という壮大なジグソーパズルの完成 (=微生物生態系の全容解明) は、何時達成するとも知れない。しかし、隠されたピース探し (=未知細菌の探索) の過程で、「社会に大変革をもたらすことになるような未知機能」を発見してしまうことは十分にあり得るものと信じて疑わない。
文献
1) Hugenholtz, P., Goebel, B. M., Pace, N. R.: J. Bacteriol., 180, 6793 (1998).
2) Hugenholtz, P., Pitulle, C., Hershberger, K. L., Pace, N. R.: J. Bacteriol., 180, 366 (1998).
3) Hanada, S., Takaichi, S., Matsuura, K., Nakamura, K.: Int. J. Syst. Evol. Micribiol., 52, 187 (2002).
4) Mori, K., Kim, H., Kakegawa, T., Hanada, S.: Extremophiles, 7, 283 (2003).
5) Sekiguchi, Y., Yamada, T., Hanada, S., Ohashi, A., Harada, H., Kamagata, Y.: Int. J. Syst. Evol. Microbiol., 53, 1843 (2003).
6) Zhang, H., Sekiguchi, Y., Hanada, S., Hugenholtz, P., Kim, H., Kamagata, Y., Nakamura, K.: Int. J. Syst. Evol. Microbiol., 53, 1155 (2003).
7) Hattori, S., Kamagata, Y., Hanada, S., Shoun, H.: Int. J. Syst. Evol. Microbiol., 50, 1601 (2000).
8) Zhang, H., Hanada, S., Shigematsu, T., Shibuya, K., Kamagata, Y., Kanagawa, T., Kurane, R.: Int. J. Syst. Evol. Microbiol., 50, 743 (2000).
9) Shintani, T., Liu, W. T., Hanada, S., Kamagata, Y., Miyaoka, S., Suzuki, T., Nakamura, K.: Int. J. Syst. Evol. Microbiol., 50, 201 (2000).
10) Mori, K., Hanada, S., Maruyama, A., Marumo, K.: Int. J. Syst. Evol. Microbiol., 52, 1675 (2002).
11) Liu, W. T., Hanada, S., Marsh, T. L., Kamagata, Y., Nakamura, K.: Int. J. Syst. Evol. Microbiol., 52, 1845 (2002).
12) Hanada, S., Liu, W. T., Shintani, T., Kamagata, Y., Nakamura, K.: Int. J. Syst. Evol. Microbiol., 52, 883 (2002).
13) Tamaki, H., Hanada, S., Kamagata, Y., Nakamura, K., Nomura, N., Nakano, K., Matsumura, M.: Int. J. Syst. Evol. Microbiol., 53, 519 (2003).
14) Takaichi, S., Maoka, T., Yamada, M., Matsuura, K., Haikawa, Y., Hanada, S.: Plant Cell Physiol., 42, 1355 (2001).
15) Nakamura, K., Hiraishi, A., Yoshimi, Y., Kawaharasaki, M., Masuda, K., Kamagata, Y.: Int. J. Syst. Bacteriol., 45, 17 (1995).
16) Onda, S., Takii, S.: J. Gen. Appl. Microbiol., 48, 125 (2002).